GLM: 线性回归#
本教程改编自 Thomas Wiecki 的一篇博客文章“推理按钮:使用 PyMC 轻松实现贝叶斯 GLM”。
虽然贝叶斯方法相对于频率学方法的理论优势已在其他地方进行了详细讨论(请参阅下面的延伸阅读),但阻碍更广泛采用的主要障碍是可用性。 这有点讽刺,因为贝叶斯统计的美妙之处在于其通用性。 频率学统计涉及提出一个特定于手头应用的检验统计量,而使用贝叶斯方法,您可以完全按照您认为合适的方式定义您的模型,然后点击推理按钮(TM)(即运行神奇的 MCMC 采样算法)。
在过去的几十年中,已经创建了一些很棒的贝叶斯软件包,包括 JAGS、BUGS 和 Stan,但它们是为非常了解自己想要构建的模型的统计学家编写的。
不幸的是,“绝大多数统计分析不是由统计学家执行的”——因此我们真正需要的是为科学家而不是统计学家提供的工具。 PyMC 可以轻松地为手头的应用构建统计模型,而无需考虑各种拟合算法的实现方式。
线性回归#
在本例中,我们将从最简单的 GLM – 线性回归开始。 总的来说,频率学家认为线性回归如下:
其中 \(Y\) 是我们想要预测的输出(或因变量),\(X\) 是我们的预测变量(或自变量),\(\beta\) 是我们想要估计的模型系数(或参数)。 \(\epsilon\) 是一个误差项,假设它服从正态分布。
然后我们可以使用普通最小二乘法 (OLS) 或最大似然法来找到最佳拟合 \(\beta\)。
概率重构#
贝叶斯学派对世界采取概率的观点,并用概率分布来表达这个模型。 我们上面的线性回归可以重构为
换句话说,我们将 \(Y\) 视为一个随机变量(或随机向量),其每个元素(数据点)都根据正态分布分布。 该正态分布的均值由我们的线性预测变量提供,方差为 \(\sigma^2\)。
虽然这本质上是相同的模型,但贝叶斯估计有两个关键优势:
先验:我们可以通过对参数设置先验来量化我们可能拥有的任何先验知识。 例如,如果我们认为 \(\sigma\) 可能很小,我们会选择一个在低值上具有更多概率质量的先验。
量化不确定性:我们不会像上面那样获得 \(\beta\) 的单一估计,而是获得关于 \(\beta\) 的不同值有多大可能性的完整后验分布。 例如,如果数据点很少,我们对 \(\beta\) 的不确定性会非常高,并且我们会得到非常宽的后验分布。
PyMC 中的贝叶斯 GLM#
为了开始在 PyMC 中构建 GLM,我们首先导入所需的模块。
import arviz as az
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
import xarray as xr
import pymc as pm
from pymc import HalfCauchy, Model, Normal, sample
print(f"Running on PyMC v{pm.__version__}")
Running on PyMC v5.15.1+68.gc0b060b98.dirty
RANDOM_SEED = 8927
rng = np.random.default_rng(RANDOM_SEED)
%config InlineBackend.figure_format = 'retina'
az.style.use("arviz-darkgrid")
生成数据#
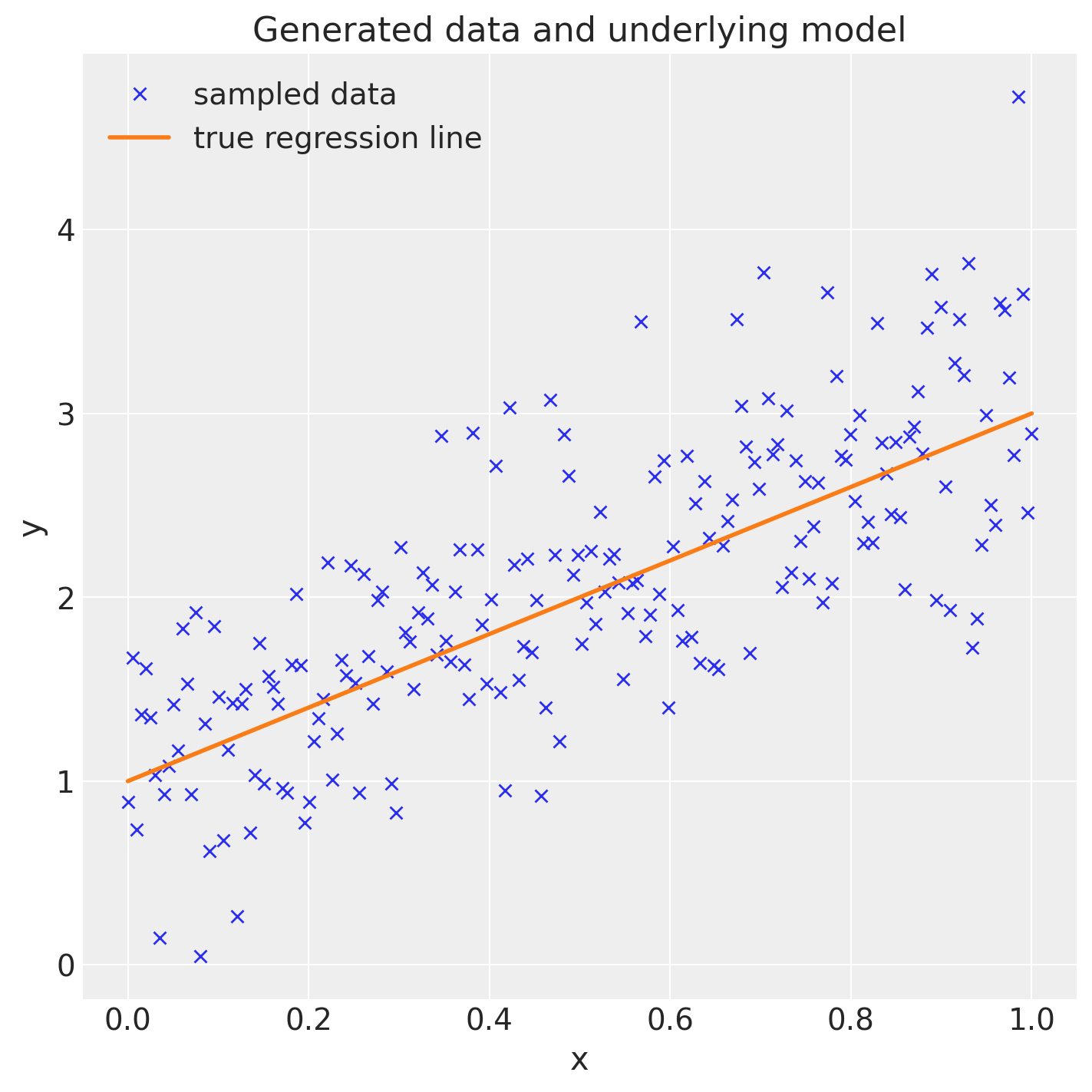
本质上,我们正在创建一条由截距和斜率定义的回归线,并通过从正态分布中采样来添加数据点,其中均值设置为回归线。
size = 200
true_intercept = 1
true_slope = 2
x = np.linspace(0, 1, size)
# y = a + b*x
true_regression_line = true_intercept + true_slope * x
# add noise
y = true_regression_line + rng.normal(scale=0.5, size=size)
data = pd.DataFrame({"x": x, "y": y})
fig = plt.figure(figsize=(7, 7))
ax = fig.add_subplot(111, xlabel="x", ylabel="y", title="Generated data and underlying model")
ax.plot(x, y, "x", label="sampled data")
ax.plot(x, true_regression_line, label="true regression line", lw=2.0)
plt.legend(loc=0);
估计模型#
让我们将贝叶斯线性回归模型拟合到这些数据。 在 PyMC 中,模型规范发生在 with 表达式中,称为上下文管理器。 默认情况下,模型使用 NUTS 采样器 进行拟合,从而生成表示潜在模型参数的边际后验分布的样本轨迹。
with Model() as model: # model specifications in PyMC are wrapped in a with-statement
# Define priors
sigma = HalfCauchy("sigma", beta=10)
intercept = Normal("Intercept", 0, sigma=20)
slope = Normal("slope", 0, sigma=20)
# Define likelihood
likelihood = Normal("y", mu=intercept + slope * x, sigma=sigma, observed=y)
# Inference!
# draw 3000 posterior samples using NUTS sampling
idata = sample(3000)
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [sigma, Intercept, slope]
Sampling 4 chains for 1_000 tune and 3_000 draw iterations (4_000 + 12_000 draws total) took 12 seconds.
对于了解概率编程的人来说,这应该相当容易理解。 但是,非统计学家会知道这一切是做什么的吗? 此外,请记住,这是一个非常简单的模型,在 R 中只需一行代码即可完成。 如果有多个潜在的转换后的回归变量、交互项或链接函数,也会使这变得更加复杂且容易出错。
为了使事情更简单,bambi 库采用 formula 线性模型说明符,从中创建设计矩阵。 然后 bambi 为每个系数添加随机变量,并为模型添加适当的似然性。
如果未安装 bambi,您可以使用 pip install bambi 安装它。
try:
import bambi as bmb
except ImportError:
!{sys.executable} -m pip install -q --upgrade bambi
import bambi as bmb
model = bmb.Model("y ~ x", data)
idata = model.fit(draws=3000)
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [y_sigma, Intercept, x]
Sampling 4 chains for 1_000 tune and 3_000 draw iterations (4_000 + 12_000 draws total) took 19 seconds.
代码更简洁,但这段代码与之前的规范执行完全相同的操作(如果需要,您也可以更改先验和其他所有内容)。 bambi 解析 formulae 模型字符串,为每个回归变量(在本例中为 Intercept 和斜率 x)添加随机变量,添加似然性(默认情况下,选择正态分布),以及所有其他变量(sigma)。 最后,bambi 通过使用 statsmodels 估计频率学线性模型,将参数初始化到一个良好的起点。
如果您不熟悉 R 的语法,'y ~ x' 指定我们有一个输出变量 y,我们想要将其估计为 x 的线性函数。
分析模型#
贝叶斯推断不仅给我们提供一条最佳拟合线(如最大似然法那样),而是提供了所有可能的参数的完整后验分布。 让我们绘制参数的后验分布以及我们绘制的单个样本。
az.plot_trace(idata, figsize=(10, 7));

左侧显示了我们的边际后验分布——对于 x 轴上的每个参数值,我们在 y 轴上获得一个概率,该概率告诉我们该参数值的可能性有多大。
这里有几件事需要注意。 首先是我们的单个参数(左侧)的采样链看起来是同质且平稳的(没有大的漂移或其他奇怪的模式)。
其次,每个变量的最大后验估计(左侧分布中的峰值)非常接近用于生成数据的真实参数(x 是回归系数,sigma 是我们正态分布的标准差)。
因此,在 GLM 中,我们不仅有一条最佳拟合回归线,而是有很多条。 后验预测图从后验分布(截距和斜率)中获取多个样本,并为每个样本绘制一条回归线。 我们可以使用后验样本直接手动生成这些回归线。
idata.posterior["y_model"] = idata.posterior["Intercept"] + idata.posterior["x"] * xr.DataArray(x)
_, ax = plt.subplots(figsize=(7, 7))
az.plot_lm(idata=idata, y="y", num_samples=100, axes=ax, y_model="y_model")
ax.set_title("Posterior predictive regression lines")
ax.set_xlabel("x");
/home/ricardo/miniconda3/envs/pymc/lib/python3.11/site-packages/arviz/plots/lmplot.py:211: UserWarning: posterior_predictive not found in idata
warnings.warn("posterior_predictive not found in idata", UserWarning)
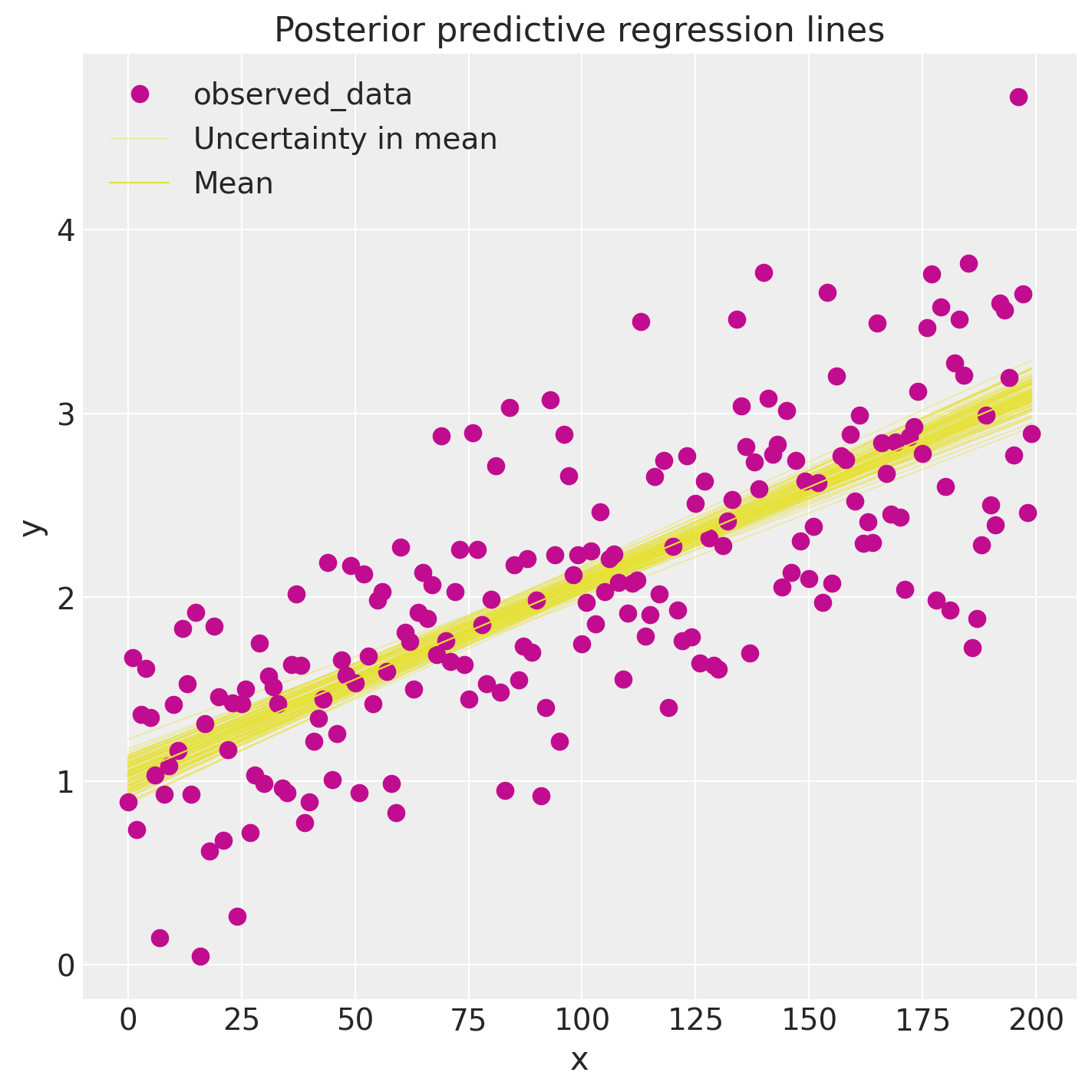
正如您所看到的,我们估计的回归线与真实的回归线非常相似。 但由于我们只有有限的数据,我们的估计中存在不确定性,这里用线的变异性表示。
总结#
可用性目前是贝叶斯统计学更广泛应用的巨大障碍。
Bambi允许使用从 R 借用的便捷语法进行 GLM 规范。 然后可以使用pymc进行推断。后验预测图使我们能够评估拟合度以及我们在其中的不确定性。
延伸阅读#
如需更多背景知识,以下是一些关于贝叶斯统计学的优秀资源:
John Kruschke 的优秀著作 Doing Bayesian Data Analysis。
作者:Thomas Wiecki
%load_ext watermark
%watermark -n -u -v -iv -w -p pytensor
Last updated: Tue Jun 25 2024
Python implementation: CPython
Python version : 3.11.8
IPython version : 8.22.2
pytensor: 2.20.0+3.g66439d283.dirty
pymc : 5.15.0+1.g58927d608
arviz : 0.17.1
xarray : 2024.2.0
numpy : 1.26.4
pandas : 2.2.1
sys : 3.11.8 | packaged by conda-forge | (main, Feb 16 2024, 20:53:32) [GCC 12.3.0]
matplotlib: 3.8.3
bambi : 0.13.0
Watermark: 2.4.3