贝叶斯估计取代 T 检验#
Running on PyMC v4.2.2
%config InlineBackend.figure_format = 'retina'
az.style.use("arviz-darkgrid")
rng = np.random.default_rng(seed=42)
问题#
此模型复现了贝叶斯估计取代 t 检验中使用的示例 [[Kruschke,2013 年]](https://doi.org/10.1037/a0029146)。。
几种统计推断程序涉及两组的比较。我们可能对一组是否比另一组更大,或者仅仅是与另一组不同感兴趣。我们需要一个统计模型来实现这一点,因为真实差异通常伴随着测量或随机噪声,这使得我们无法仅从根据观测数据计算出的差异中得出结论。
比较两个(或多个)样本的事实标准是使用统计检验。这涉及表达零假设(通常声称组之间没有差异),并使用选择的检验统计量来确定在假设下观测数据的分布是否合理。当计算出的检验统计量高于某个预先指定的阈值时,就会发生这种拒绝。
不幸的是,正确地进行假设检验并不容易,并且其结果非常容易被误解。设置统计检验涉及用户的几个主观选择(例如,要使用的统计检验、要检验的零假设、显着性水平),这些选择很少基于手头的问题或决策来证明是合理的,而是通常基于完全任意的传统选择 [[Johnson,1999 年]](https://doi.org/10.2307/3802789)。。它为用户提供的证据是间接的、不完整的,并且通常夸大了反对零假设的证据 [[Goodman,1999 年]](https://doi.org/10.7326/0003-4819-130-12-199906150-00008)。。
一种更具信息性和有效性的组比较方法是基于估计而不是检验的方法,并且由贝叶斯概率而不是频率学驱动。也就是说,我们不是检验两组是否不同,而是追求估计它们有多么不同,这从根本上来说更具信息性。此外,我们还包括与该差异相关的不确定性估计,其中包括由于我们对模型参数的知识不足(认知不确定性)和由于系统的固有随机性(偶然不确定性)而产生的不确定性。
示例:药物试验评估#
为了说明这种贝叶斯估计方法在实践中如何工作,我们将使用 Kruschke [[2013 年]](https://doi.org/10.1037/a0029146) 中的一个虚构示例,该示例涉及药物评估的临床试验评估。该试验旨在通过比较治疗组(接受药物的个体)和对照组(接受安慰剂的个体)的智商评分来评估“智能药物”的疗效,该药物应该可以提高智力。治疗组和对照组分别有 47 名个体和 42 名个体。
# fmt: off
iq_drug = np.array([
101, 100, 102, 104, 102, 97, 105, 105, 98, 101, 100, 123, 105, 103,
100, 95, 102, 106, 109, 102, 82, 102, 100, 102, 102, 101, 102, 102,
103, 103, 97, 97, 103, 101, 97, 104, 96, 103, 124, 101, 101, 100,
101, 101, 104, 100, 101
])
iq_placebo = np.array([
99, 101, 100, 101, 102, 100, 97, 101, 104, 101, 102, 102, 100, 105,
88, 101, 100, 104, 100, 100, 100, 101, 102, 103, 97, 101, 101, 100,
101, 99, 101, 100, 100, 101, 100, 99, 101, 100, 102, 99, 100, 99
])
# fmt: on
df1 = pd.DataFrame({"iq": iq_drug, "group": "drug"})
df2 = pd.DataFrame({"iq": iq_placebo, "group": "placebo"})
indv = pd.concat([df1, df2]).reset_index()
sns.histplot(data=indv, x="iq", hue="group");
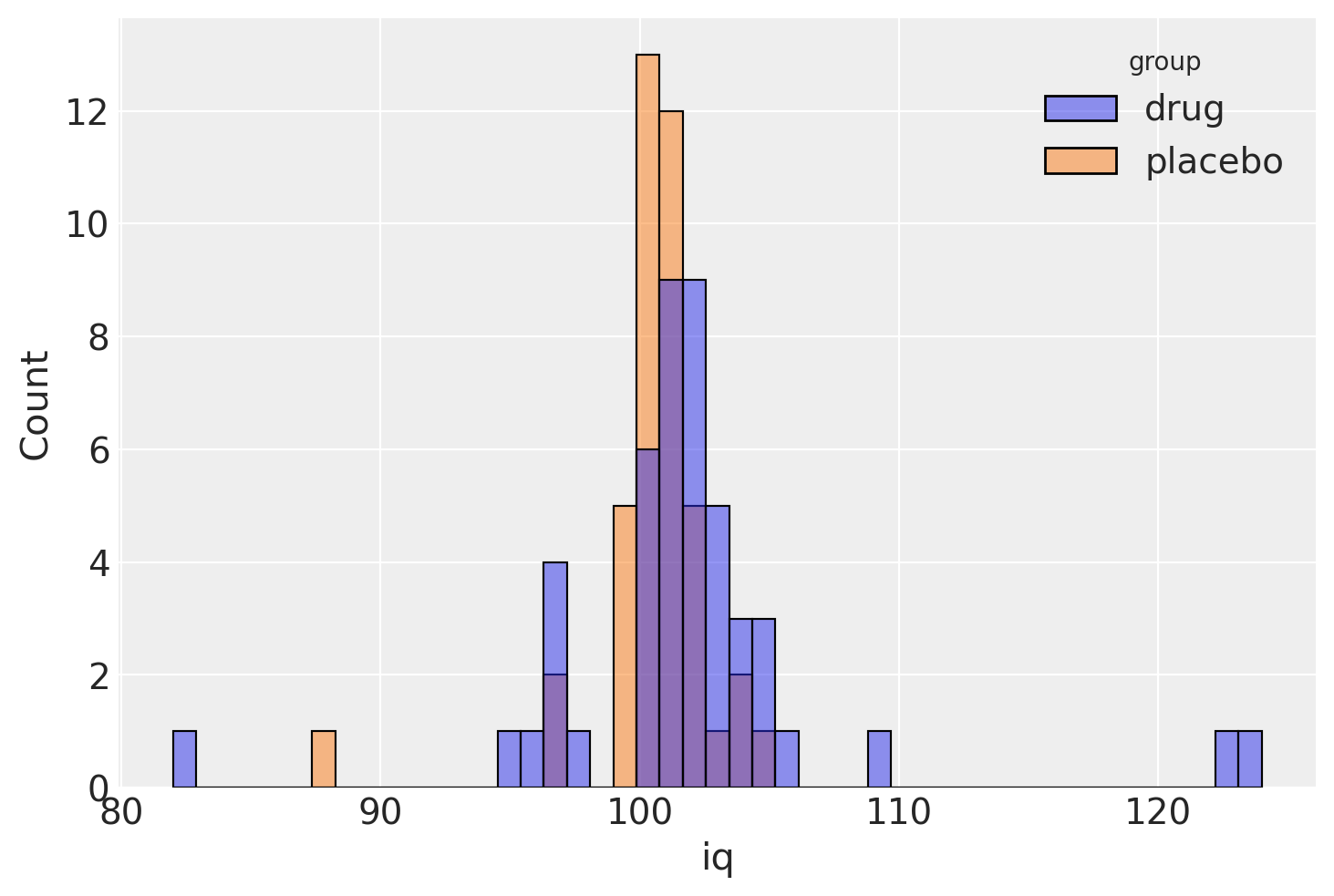
贝叶斯推断方法的第一步是指定与问题相对应的完整概率模型。对于此示例,Kruschke 选择 Student-t 分布来描述每组分数的分布。此选择增加了分析的稳健性,因为相对于正态分布,T 分布对异常值观测不太敏感。三参数 Student-t 分布允许指定均值 \(\mu\)、精度(逆方差) \(\lambda\) 和自由度参数 \(\nu\)
自由度参数本质上指定数据的“正态性”,因为 \(\nu\) 的值越大,分布就越收敛于正态分布,而小值(接近于零)会导致更重的尾部。因此,我们模型的似然函数指定如下
作为简化假设,我们将假设正态程度 \(\nu\) 对于两组是相同的。当然,我们将对均值 \(\mu_k, k=1,2\) 和标准差 \(\sigma_k\) 使用单独的参数。由于均值是实值,我们将对它们应用正态先验,并将超参数任意设置为数据的合并经验均值和两倍的合并经验标准差,这会将非常分散的信息应用于这些量(并且重要的是,先验不偏向于任何一方)。
组标准差将被赋予一个均匀先验,该先验范围覆盖结果变量 IQ 变异性的合理值范围。
在 Kruschke 的原始模型中,他对组标准差使用了非常宽的均匀先验,从合并的经验标准差除以 1000 到合并的标准差乘以 1000。这是一个糟糕的先验选择,因为关于人类认知测量的非常基本的先验知识表明,变异性永远不可能像这个上限那么高。智商是一种标准化测量,因此这限制了给定人群的智商值的变异程度。当您对这些值施加如此宽的均匀先验时,您实际上是在不可接受的值上赋予了很大的先验权重。在此示例中,实际差异很小,但总的来说,最好将您可用的尽可能多的先验信息应用于先验分布的参数化。
我们将改为将组标准差设置为具有 \(\text{Uniform}(1,10)\) 先验
sigma_low = 10**-1
sigma_high = 10
with model:
group1_std = pm.Uniform("group1_std", lower=sigma_low, upper=sigma_high)
group2_std = pm.Uniform("group2_std", lower=sigma_low, upper=sigma_high)
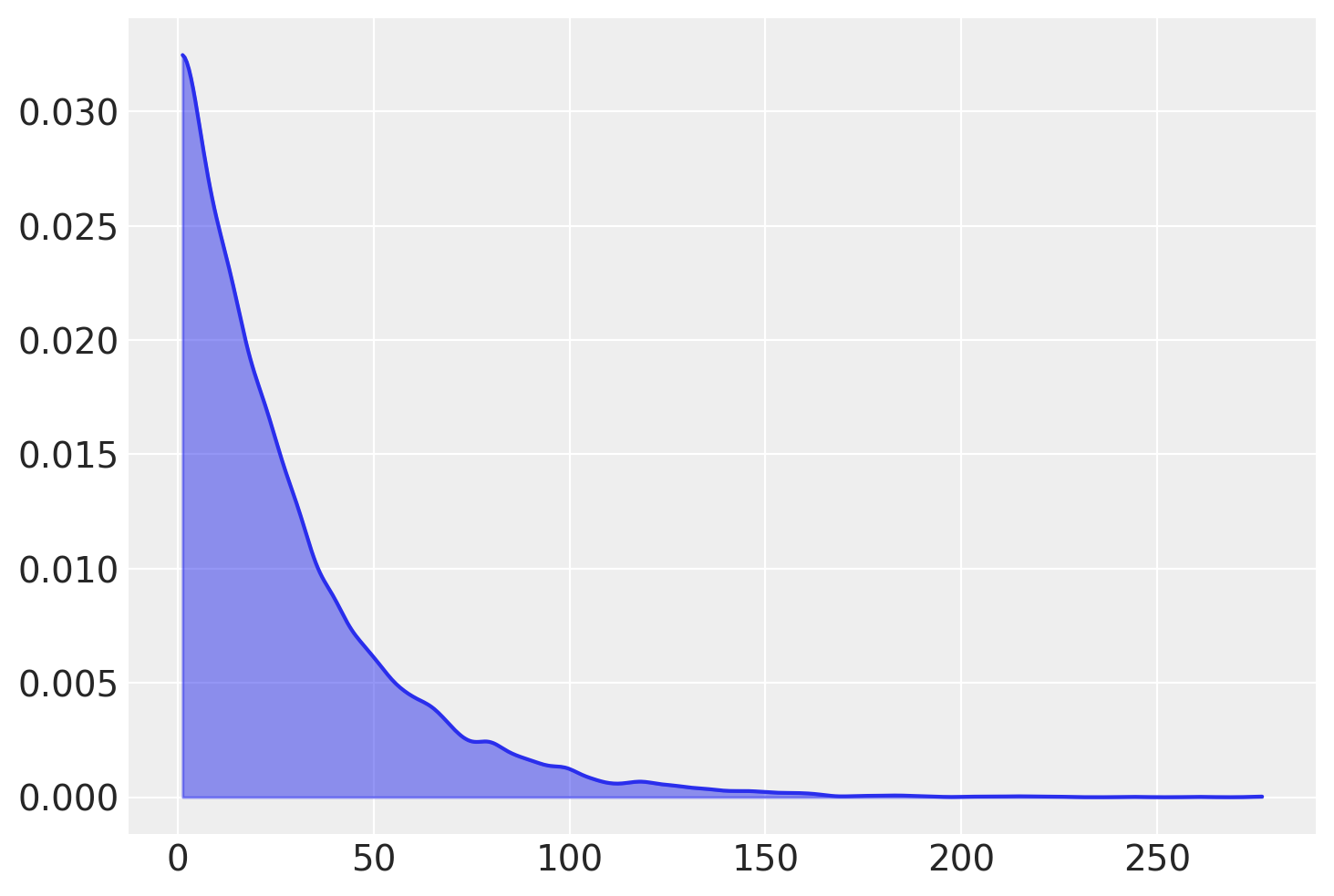
我们效仿 Kruschke,通过使 \(\nu\) 的先验呈指数分布且均值为 30;这在参数区域上分配了较高的先验概率,这些区域描述了 Student-T 分布下从正态数据到重尾数据的范围。
with model:
nu_minus_one = pm.Exponential("nu_minus_one", 1 / 29.0)
nu = pm.Deterministic("nu", nu_minus_one + 1)
nu_log10 = pm.Deterministic("nu_log10", np.log10(nu))
az.plot_kde(rng.exponential(scale=29, size=10000) + 1, fill_kwargs={"alpha": 0.5});
由于 PyMC 以精度而不是标准差来参数化 Student-T,因此我们必须在指定似然性之前转换标准差。
with model:
lambda_1 = group1_std**-2
lambda_2 = group2_std**-2
group1 = pm.StudentT("drug", nu=nu, mu=group1_mean, lam=lambda_1, observed=iq_drug)
group2 = pm.StudentT("placebo", nu=nu, mu=group2_mean, lam=lambda_2, observed=iq_placebo)
在完全指定了我们的概率模型之后,我们可以将注意力转向计算感兴趣的比较,以便评估药物的效果。为此,我们可以在模型中为组均值之间的差异和组标准差之间的差异指定确定性节点。将它们包装在命名的 Deterministic 对象中会向 PyMC 发出信号,表明我们希望记录采样的值作为输出的一部分。作为组的联合度量,我们还将估计“效应量”,即均值差异按标准差的合并估计值进行缩放。这个量可能更难解释,因为它不再与我们的数据单位相同,但该量是所有四个估计参数的函数。
with model:
diff_of_means = pm.Deterministic("difference of means", group1_mean - group2_mean)
diff_of_stds = pm.Deterministic("difference of stds", group1_std - group2_std)
effect_size = pm.Deterministic(
"effect size", diff_of_means / np.sqrt((group1_std**2 + group2_std**2) / 2)
)
现在,我们可以拟合模型并评估其输出。
with model:
idata = pm.sample()
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [group1_mean, group2_mean, group1_std, group2_std, nu_minus_one]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 20 seconds.
The acceptance probability does not match the target. It is 0.8983, but should be close to 0.8. Try to increase the number of tuning steps.
The acceptance probability does not match the target. It is 0.8916, but should be close to 0.8. Try to increase the number of tuning steps.
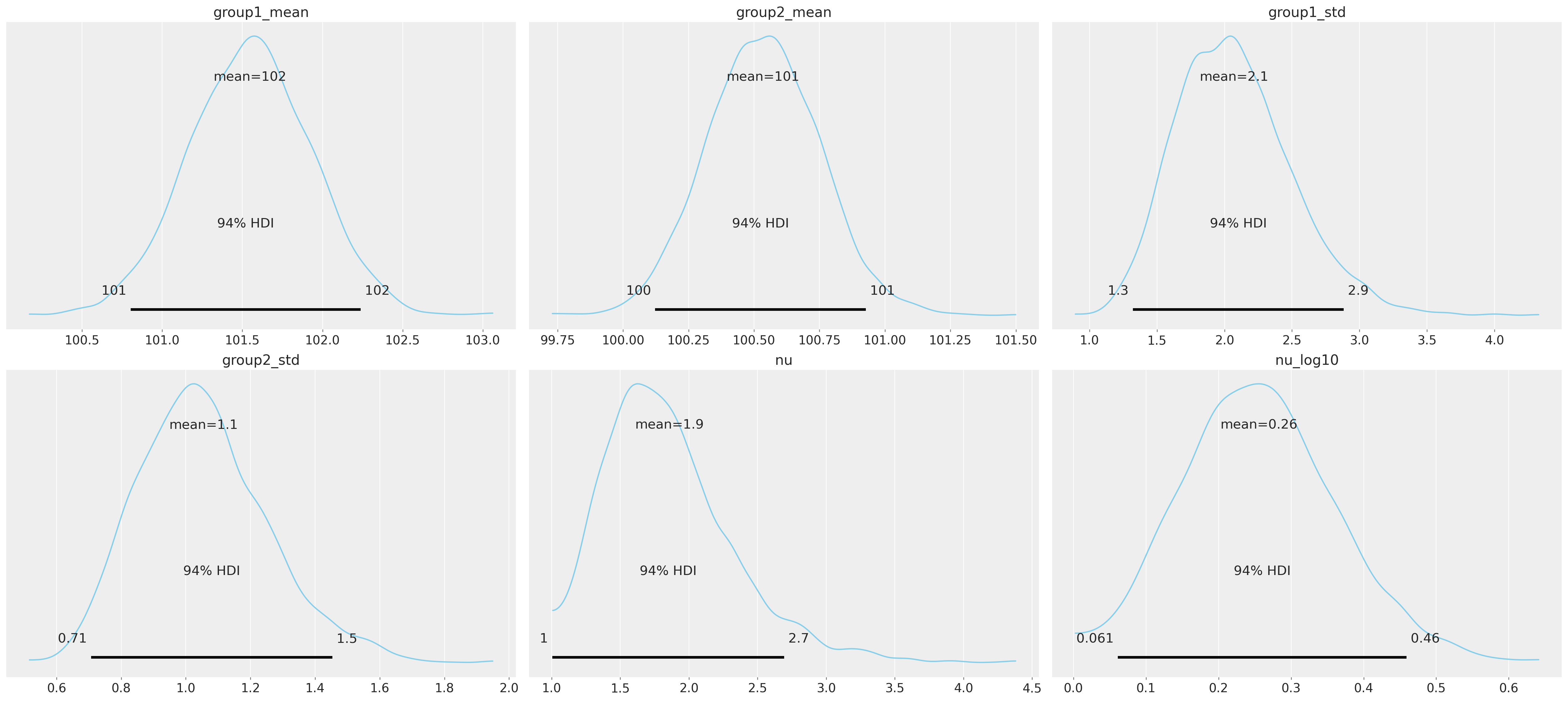
我们可以绘制模型的随机参数。Arviz 的 plot_posterior 函数复现了 [[Kruschke,2013 年]](https://doi.org/10.1037/a0029146) 中描述的信息性直方图。这些直方图总结了参数的后验分布,并呈现了 95% 的可信区间和后验均值。下面的图表是使用来自 2 条链的每条链的最后 1000 个样本合并在一起构建的。
az.plot_posterior(
idata,
var_names=["group1_mean", "group2_mean", "group1_std", "group2_std", "nu", "nu_log10"],
color="#87ceeb",
);
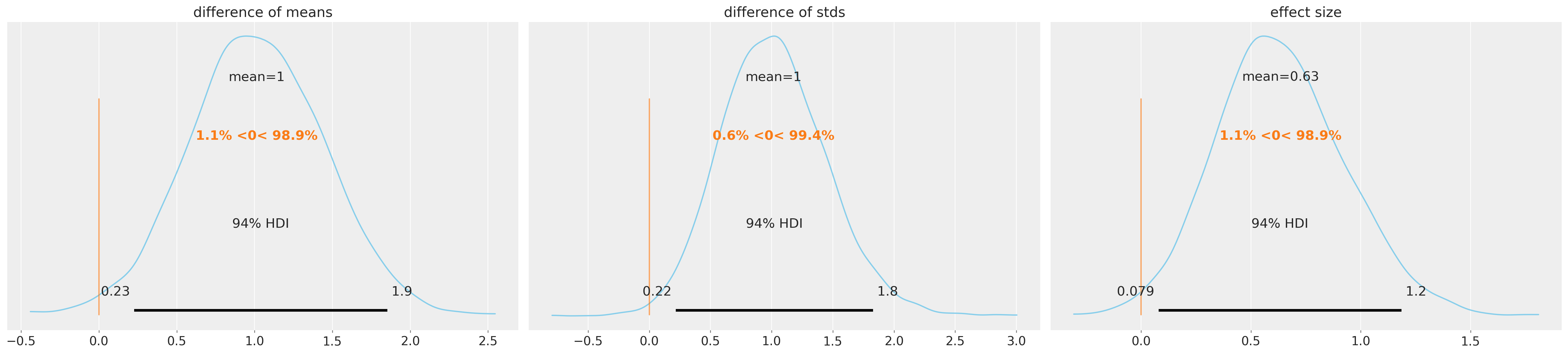
查看下面的组差异,我们可以得出结论,对于所有三个度量,两组之间都存在有意义的差异。对于这些比较,使用零作为参考值 (ref_val) 很有用;提供此参考值会产生后验分布在该值任一侧的累积概率。因此,对于均值差异,至少 97% 的后验概率大于零,这表明组均值在可信范围内是不同的。效应量和标准差的差异也类似地为正。
这些估计表明,“智能药物”既增加了预期分数,也增加了样本中分数的变异性。因此,这并不排除某些接受者可能受到药物的不利影响,同时其他人受益的可能性。
az.plot_posterior(
idata,
var_names=["difference of means", "difference of stds", "effect size"],
ref_val=0,
color="#87ceeb",
);
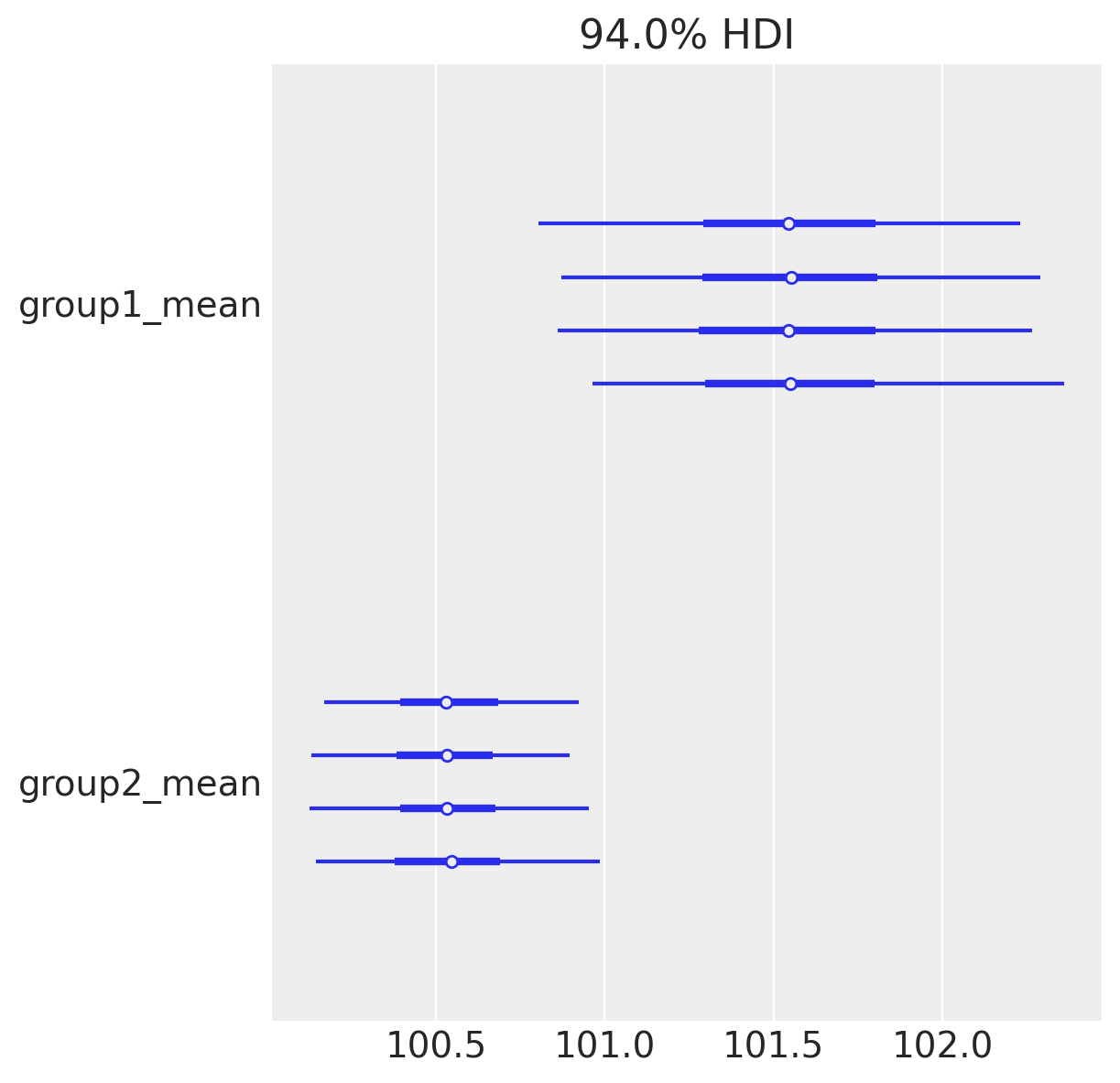
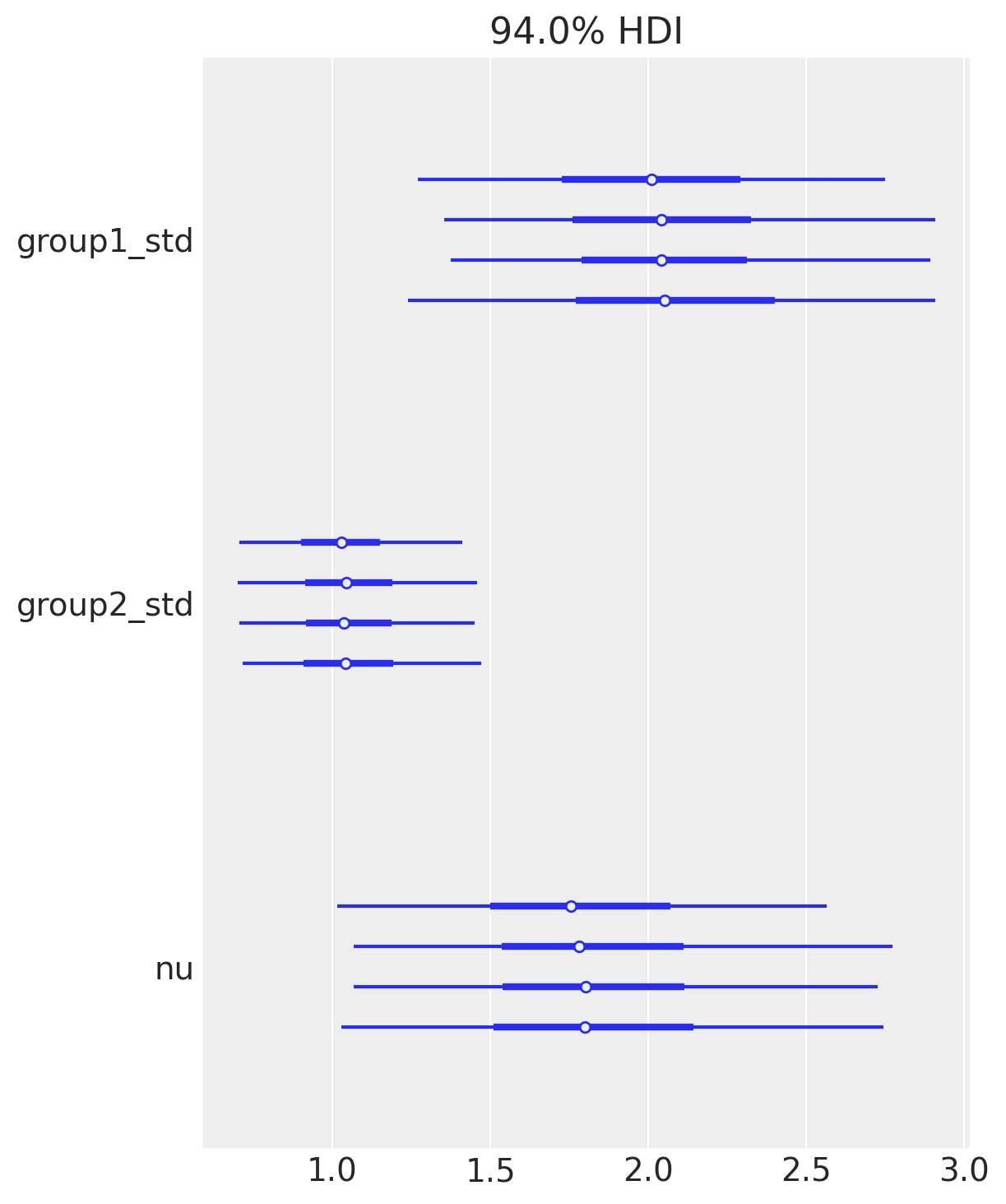
当在具有多个链的跟踪上调用 plot_forest 时,它还会绘制潜在尺度缩减参数,该参数用于揭示缺乏收敛的证据;此处接近于 1 的值表明模型已收敛。
az.plot_forest(idata, var_names=["group1_mean", "group2_mean"]);
az.plot_forest(idata, var_names=["group1_std", "group2_std", "nu"]);
az.summary(idata, var_names=["difference of means", "difference of stds", "effect size"])
| 均值 | 标准差 | hdi_3% | hdi_97% | mcse_均值 | mcse_标准差 | ess_bulk | ess_tail | r_hat | |
|---|---|---|---|---|---|---|---|---|---|
| 均值差异 | 1.014 | 0.436 | 0.225 | 1.854 | 0.007 | 0.005 | 3880.0 | 2915.0 | 1.0 |
| 标准差差异 | 1.015 | 0.435 | 0.218 | 1.828 | 0.007 | 0.005 | 3749.0 | 2876.0 | 1.0 |
| 效应量 | 0.635 | 0.295 | 0.079 | 1.185 | 0.005 | 0.003 | 4058.0 | 2577.0 | 1.0 |
作者#
由 Andrew Straw 于 2012 年 12 月创作 (best)
由 Thomas Wiecki 于 2015 年移植到 PyMC3
由 Chris Fonnesbeck 于 2020 年 12 月更新
由 Andrés Suárez 于 2022 年 1 月移植到 PyMC4 (pymc-examples#52)
参考文献#
John K. Kruschke。贝叶斯估计取代 t 检验。实验心理学杂志:通论,142(2):573–603,2013 年。URL:https://doi.org/10.1037/a0029146,doi:10.1037/a0029146。
Douglas H. Johnson。统计显着性检验的微不足道。野生动物管理杂志,63(3):763,1999 年 7 月。URL:https://doi.org/10.2307/3802789,doi:10.2307/3802789。
Steven N. Goodman。迈向循证医学统计。1:p 值谬误。内科医学年鉴,130(12):995,1999 年 6 月。URL:https://doi.org/10.7326/0003-4819-130-12-199906150-00008,doi:10.7326/0003-4819-130-12-199906150-00008。
水印#
%load_ext watermark
%watermark -n -u -v -iv -w
Last updated: Wed Nov 09 2022
Python implementation: CPython
Python version : 3.10.6
IPython version : 8.5.0
numpy : 1.23.4
seaborn: 0.12.0
arviz : 0.13.0
pandas : 1.5.1
pymc : 4.2.2
Watermark: 2.3.1
许可声明#
此示例库中的所有笔记本均根据 MIT 许可证 提供,该许可证允许修改和再分发以用于任何用途,前提是保留版权和许可声明。
引用 PyMC 示例#
要引用此笔记本,请使用 Zenodo 为 pymc-examples 存储库提供的 DOI。
重要提示
许多笔记本都改编自其他来源:博客、书籍…… 在这种情况下,您也应该引用原始来源。
另请记住引用您的代码使用的相关库。
这是一个 bibtex 中的引用模板
@incollection{citekey,
author = "<notebook authors, see above>",
title = "<notebook title>",
editor = "PyMC Team",
booktitle = "PyMC examples",
doi = "10.5281/zenodo.5654871"
}
一旦渲染,它可能看起来像