采样器统计信息#
import arviz as az
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
import pymc as pm
%matplotlib inline
print(f"Running on PyMC v{pm.__version__}")
WARNING (pytensor.tensor.blas): Using NumPy C-API based implementation for BLAS functions.
Running on PyMC v4.0.0b6
az.style.use("arviz-darkgrid")
plt.rcParams["figure.constrained_layout.use"] = False
在检查收敛性或调试行为不端的采样器时,仔细查看采样器正在执行的操作通常很有帮助。为此,一些采样器为每个生成的样本导出统计信息。
作为一个最小的例子,我们从标准正态分布中采样
model = pm.Model()
with model:
mu1 = pm.Normal("mu1", mu=0, sigma=1, shape=10)
with model:
step = pm.NUTS()
idata = pm.sample(2000, tune=1000, init=None, step=step, chains=4)
Multiprocess sampling (4 chains in 2 jobs)
NUTS: [mu1]
Sampling 4 chains for 1_000 tune and 2_000 draw iterations (4_000 + 8_000 draws total) took 7 seconds.
注意: NUTS 提供以下统计信息(这些是采样器使用的内部统计信息,使用 PyMC 时您无需对它们执行任何操作,要了解更多信息,请参阅pymc.NUTS。
idata.sample_stats
<xarray.Dataset>
Dimensions: (chain: 4, draw: 2000)
Coordinates:
* chain (chain) int64 0 1 2 3
* draw (draw) int64 0 1 2 3 4 5 ... 1995 1996 1997 1998 1999
Data variables: (12/13)
lp (chain, draw) float64 -17.41 -11.12 ... -13.76 -12.35
perf_counter_diff (chain, draw) float64 0.0009173 0.0009097 ... 0.0006041
acceptance_rate (chain, draw) float64 0.8478 1.0 ... 0.8888 0.8954
energy_error (chain, draw) float64 0.3484 -1.357 ... -0.2306 -0.2559
energy (chain, draw) float64 21.75 18.45 16.03 ... 19.25 16.51
tree_depth (chain, draw) int64 2 2 2 2 2 2 2 2 ... 2 2 3 2 2 2 2 2
... ...
diverging (chain, draw) bool False False False ... False False
step_size (chain, draw) float64 0.8831 0.8831 ... 0.848 0.848
n_steps (chain, draw) float64 3.0 3.0 3.0 3.0 ... 3.0 3.0 3.0
perf_counter_start (chain, draw) float64 2.591e+05 2.591e+05 ... 2.591e+05
process_time_diff (chain, draw) float64 0.0009183 0.0009112 ... 0.0006032
max_energy_error (chain, draw) float64 0.3896 -1.357 ... 0.2427 0.303
Attributes:
created_at: 2022-05-31T19:50:21.571347
arviz_version: 0.12.1
inference_library: pymc
inference_library_version: 4.0.0b6
sampling_time: 6.993547439575195
tuning_steps: 1000样本统计变量定义如下
process_time_diff: 绘制样本所花费的时间,由 python 标准库 time.process_time 定义。这计算所有 CPU 时间,包括 BLAS 和 OpenMP 中的工作进程。step_size: 当前积分步长。diverging: (布尔值) 指示是否存在从起始点开始能量偏差较大且随后终止轨迹的 leapfrog 跃迁。“较大”定义为max_energy_error超过阈值。lp: 模型的联合对数后验密度(直到加性常数)。energy: 接受的提议的哈密顿能量值(直到加性常数)。energy_error: 初始点和接受的提议之间哈密顿能量的差异。perf_counter_diff: 绘制样本所花费的时间,由 python 标准库 time.perf_counter(挂钟时间)定义。perf_counter_start: 绘制计算开始时 time.perf_counter 的值。n_steps: 计算的 leapfrog 步数。它与tree_depth相关,n_steps <= 2^tree_dept。max_energy_error: 初始点和提议树中所有可能的样本之间哈密顿能量的最大绝对差值。acceptance_rate: 提议树中所有可能的样本的平均接受概率。step_size_bar: 当前最佳已知步长。在调优样本之后,步长设置为此值。这应该在调优期间收敛。tree_depth: 平衡二叉树中树加倍的次数。
一些注意事项
NUTS 使用的一些样本统计信息在转换为
InferenceData时会重命名,以遵循 ArviZ 的命名约定,而另一些是 PyMC3 特有的,并在生成的 InferenceData 对象中保留其内部 PyMC3 名称。InferenceData还存储其他信息,如日期、使用的版本、采样时间和调优步数作为属性。
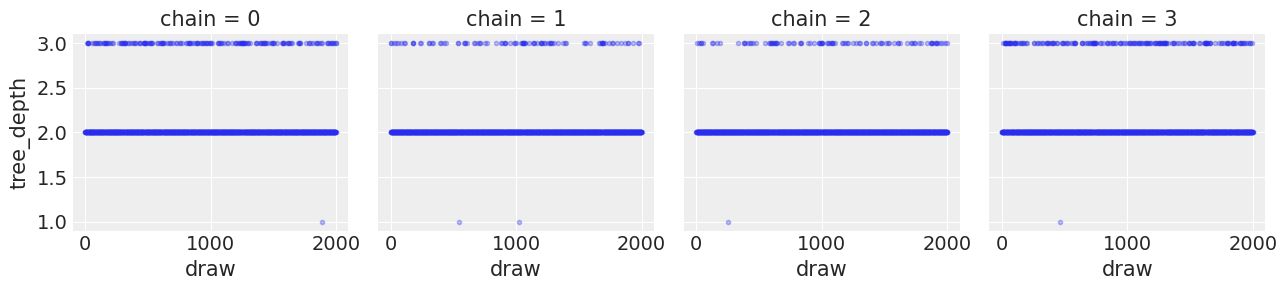
idata.sample_stats["tree_depth"].plot(col="chain", ls="none", marker=".", alpha=0.3);
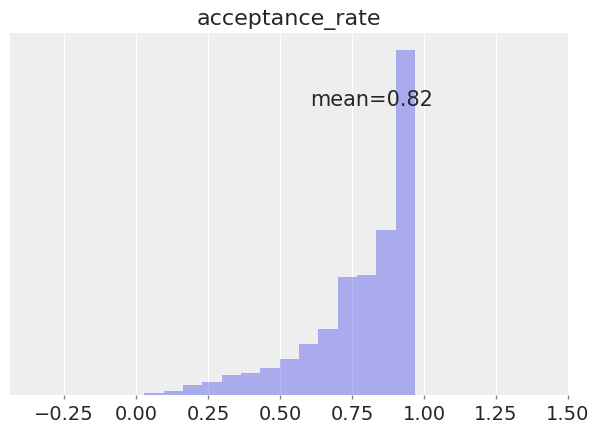
az.plot_posterior(
idata, group="sample_stats", var_names="acceptance_rate", hdi_prob="hide", kind="hist"
);
我们检查是否有任何偏差,如果有,有多少?
idata.sample_stats["diverging"].sum()
<xarray.DataArray 'diverging' ()> array(0)
在这种情况下,未发现偏差。如果存在任何偏差,请查看 此 notebook,以获取有关处理偏差的信息。
比较能量水平的总体分布与连续样本之间能量的变化通常很有用。理想情况下,它们应该非常相似
az.plot_energy(idata, figsize=(6, 4));

如果能量水平的总体分布具有更长的尾部,则采样器的效率将迅速下降。
多个采样器#
如果同一模型使用多个采样器(例如,用于连续变量和离散变量),则导出的值将沿新轴合并或堆叠。
coords = {"step": ["BinaryMetropolis", "Metropolis"], "obs": ["mu1"]}
dims = {"accept": ["step"]}
with pm.Model(coords=coords) as model:
mu1 = pm.Bernoulli("mu1", p=0.8)
mu2 = pm.Normal("mu2", mu=0, sigma=1, dims="obs")
with model:
step1 = pm.BinaryMetropolis([mu1])
step2 = pm.Metropolis([mu2])
idata = pm.sample(
10000,
init=None,
step=[step1, step2],
chains=4,
tune=1000,
idata_kwargs={"dims": dims, "coords": coords},
)
Multiprocess sampling (4 chains in 2 jobs)
CompoundStep
>BinaryMetropolis: [mu1]
>Metropolis: [mu2]
Sampling 4 chains for 1_000 tune and 10_000 draw iterations (4_000 + 40_000 draws total) took 15 seconds.
list(idata.sample_stats.data_vars)
['p_jump', 'scaling', 'accepted', 'accept']
两个采样器都导出 accept,因此我们为每个采样器获得一个接受概率
az.plot_posterior(
idata,
group="sample_stats",
var_names="accept",
hdi_prob="hide",
kind="hist",
);
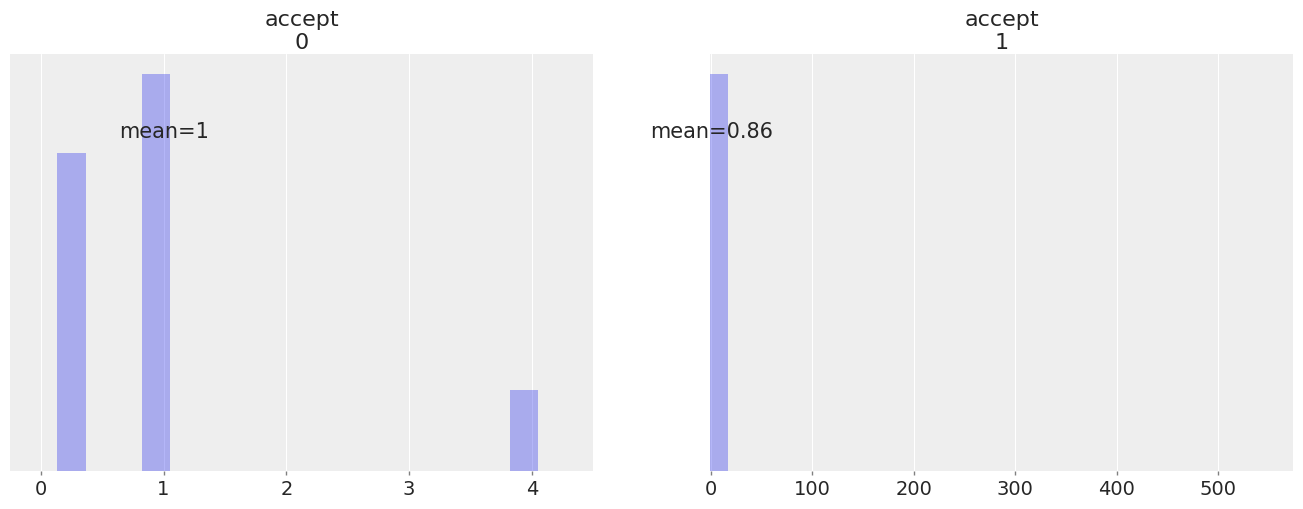
我们注意到 accept 有时会取非常高的值(从低概率区域跳到高得多的概率区域)。
# Range of accept values
idata.sample_stats["accept"].max("draw") - idata.sample_stats["accept"].min("draw")
<xarray.DataArray 'accept' (chain: 4, accept_dim_0: 2)>
array([[ 3.75 , 573.3089824 ],
[ 3.75 , 184.17692429],
[ 3.75 , 194.61242919],
[ 3.75 , 88.51883672]])
Coordinates:
* chain (chain) int64 0 1 2 3
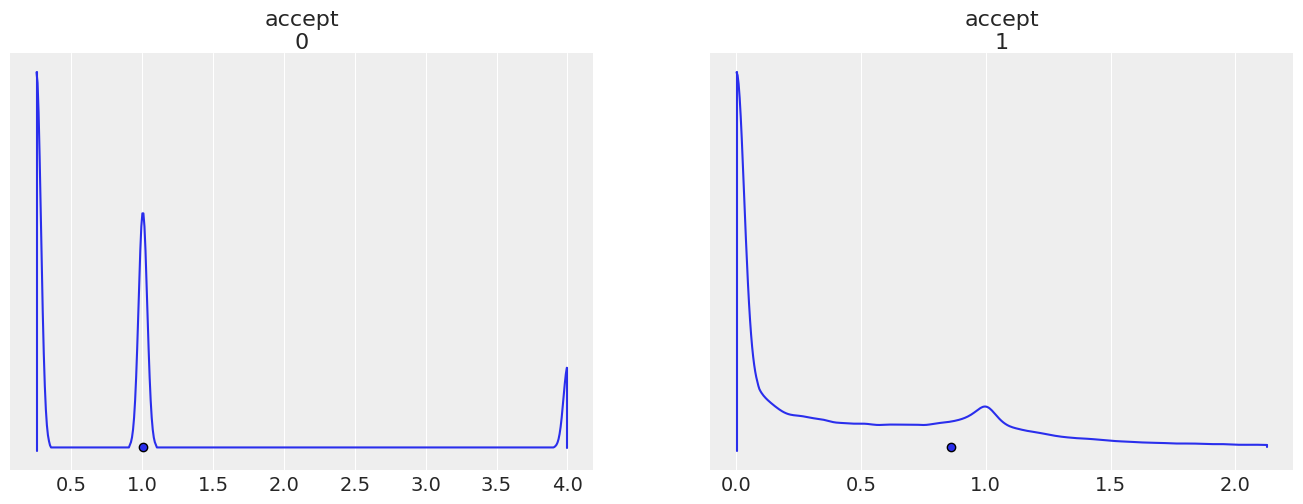
* accept_dim_0 (accept_dim_0) int64 0 1# We can try plotting the density and view the high density intervals to understand the variable better
az.plot_density(
idata,
group="sample_stats",
var_names="accept",
point_estimate="mean",
);
水印#
%load_ext watermark
%watermark -n -u -v -iv -w
Last updated: Tue May 31 2022
Python implementation: CPython
Python version : 3.10.4
IPython version : 8.4.0
arviz : 0.12.1
numpy : 1.23.0rc2
pymc : 4.0.0b6
matplotlib: 3.5.2
pandas : 1.4.2
Watermark: 2.3.1
许可证声明#
此示例库中的所有 notebook 均根据 MIT 许可证 提供,该许可证允许修改和再分发用于任何用途,前提是保留版权和许可证声明。
引用 PyMC 示例#
要引用此 notebook,请使用 Zenodo 为 pymc-examples 存储库提供的 DOI。
重要提示
许多 notebook 改编自其他来源:博客、书籍... 在这种情况下,您也应该引用原始来源。
另请记住引用您的代码使用的相关库。
这是一个 bibtex 中的引用模板
@incollection{citekey,
author = "<notebook authors, see above>",
title = "<notebook title>",
editor = "PyMC Team",
booktitle = "PyMC examples",
doi = "10.5281/zenodo.5654871"
}
渲染后可能看起来像