高斯过程:HSGP 参考和入门#
希尔伯特空间高斯过程近似是一种低秩 GP 近似,特别适合在概率编程语言(如 PyMC)中使用。它使用预先计算和固定的基函数集来近似 GP,这些基函数不依赖于协方差核的形式或其超参数。它是一种参数化近似,因此在 PyMC 中进行预测可以像使用线性模型一样通过 pm.Data 或 pm.set_data 完成。您不需要定义非参数 GP 所依赖的 .conditional 分布。这使得将 HSGP 而不是 GP 集成到您现有的 PyMC 模型中容易得多。此外,与许多其他 GP 近似不同,HSGP 可以用于模型中的任何位置以及任何似然函数。
它也很快。未近似 GP 每个 MCMC 步骤的计算成本为 \(\mathcal{O}(n^3)\),其中 \(n\) 是数据点的数量。对于 HSGP,它是 \(\mathcal{O}(mn + m)\),其中 \(m\) 是基向量的数量。重要的是要注意,采样速度也很大程度上取决于后验几何。
HSGP 近似确实有一些限制
它只能与平稳协方差核一起使用,例如 Matern 族。
HSGP类与任何实现power_spectral_density方法的Covariance类兼容。对于Periodic协方差有一个特例,它在 PyMC 中由HSGPPeriodic实现。它在输入维度上扩展性不佳。如果您的 GP 是在诸如时间序列的一维过程或二维空间点过程上,则 HSGP 近似是一个不错的选择。当输入维度大于 3 时,它可能不是一个有效的选择。
它可能难以处理变化更迅速的过程。如果您尝试建模的过程相对于域的范围变化非常快,则 HSGP 近似可能无法准确表示它。我们将在后面的章节中展示如何设置近似的精度,这需要在近似的保真度和计算复杂度之间进行权衡。
对于较小的数据集,完整的未近似 GP 可能仍然更有效.
此实现的次要目标是通过可访问的实现来实现灵活性,其中核心计算以模块化方式实现。对于基本用法,用户可以使用 .prior 和 .conditional 方法,并将 HSGP 类基本上视为 pm.gp.Latent(未近似 GP)的直接替代品。更高级的用户可以绕过这些方法,而使用 .prior_linearized,这将 HSGP 暴露为参数模型。对于具有多个 HSGP 的更复杂模型,用户可以直接使用诸如 pm.gp.hsgp_approx.calc_eigenvalues 和 pm.gp.hsgp_approx.calc_eigenvectors 之类的函数。
参考文献:#
原始参考文献:Solin & Sarkka, 2019。
概率编程语言中的 HSGP:Riutort-Mayol et al., 2020。
HSTP(学生 t 过程):Sellier & Dellaportas, 2023。
克罗内克 HSGP:Dan et al., 2022
PyMC 的
HSGPAPI
示例 1:基本 HSGP 用法#
我们将使用模拟数据来激发 HSGP 用法的概述。如果您对以下内容感兴趣,请参阅本节
查看
HSGP在操作中的简单示例。用更快的近似替换标准 GP,即
pm.gp.Latent,只要您使用的是更常见的协方差核之一,如ExpQuad、Matern52或Matern32。了解何时使用中心化或非中心化参数化。
加性 GP 的快速示例
import arviz as az
import matplotlib.pyplot as plt
import numpy as np
import preliz as pz
import pymc as pm
import pytensor.tensor as pt
# Sample on the CPU
%env CUDA_VISIBLE_DEVICES=''
# import jax
# import numpyro
# numpyro.set_host_device_count(6)
env: CUDA_VISIBLE_DEVICES=''
az.style.use("arviz-whitegrid")
plt.rcParams["figure.figsize"] = [12, 5]
%config InlineBackend.figure_format = 'retina'
seed = sum(map(ord, "hsgp"))
rng = np.random.default_rng(seed)
模拟数据#
def simulate_1d(x, ell_true, eta_true, sigma_true):
"""Given a domain x, the true values of the lengthscale ell, the
scale eta, and the noise sigma, simulate a one-dimensional GP
observed at the given x-locations.
"""
# Draw one sample from the underlying GP.
n = len(x)
cov_func = eta_true**2 * pm.gp.cov.Matern52(1, ell_true)
gp_true = pm.MvNormal.dist(mu=np.zeros(n), cov=cov_func(x[:, None]))
f_true = pm.draw(gp_true, draws=1, random_seed=rng)
# The observed data is the latent function plus a small amount
# of Gaussian distributed noise.
noise_dist = pm.Normal.dist(mu=0.0, sigma=sigma_true)
y_obs = f_true + pm.draw(noise_dist, draws=n, random_seed=rng)
return y_obs, f_true
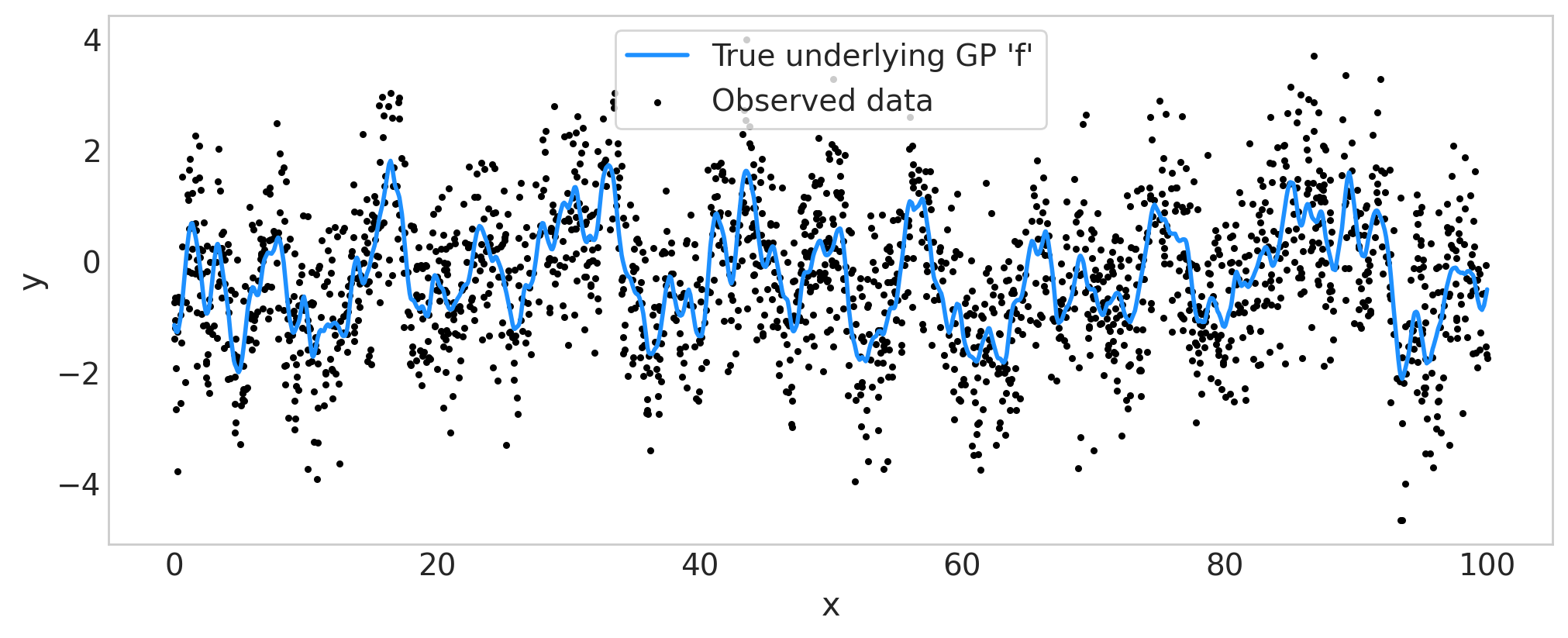
fig = plt.figure(figsize=(10, 4))
ax = fig.gca()
x = 100.0 * np.sort(np.random.rand(2000))
y_obs, f_true = simulate_1d(x=x, ell_true=1.0, eta_true=1.0, sigma_true=1.0)
ax.plot(x, f_true, color="dodgerblue", lw=2, label="True underlying GP 'f'")
ax.scatter(x, y_obs, marker="o", color="black", s=5, label="Observed data")
ax.set_xlabel("x")
ax.set_ylabel("y")
ax.legend(frameon=True)
ax.grid(False)
定义和拟合 HSGP 模型#
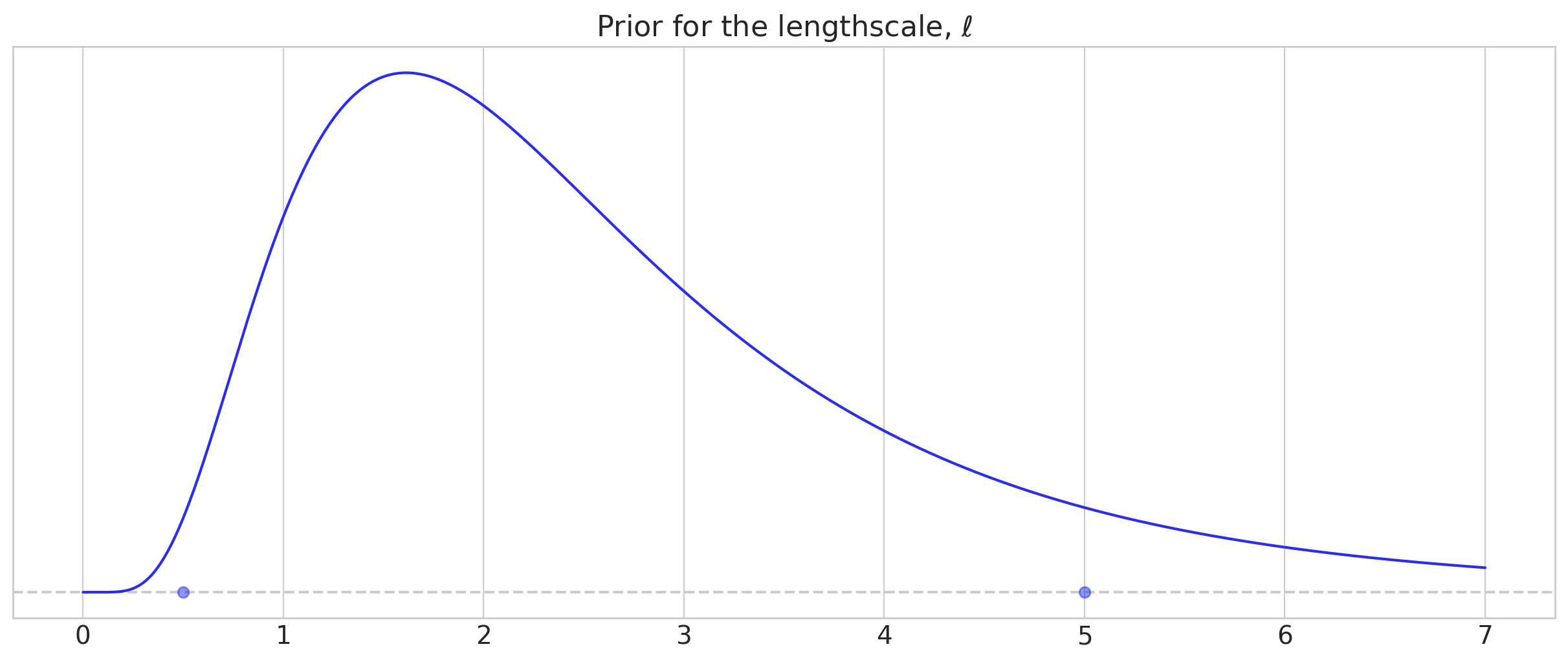
首先,我们使用 pz.maxent 为长度尺度参数选择先验,maxent 返回在指定区间 [lower, upper] 内具有指定 mass 的最大熵先验。
我们使用对数正态分布来惩罚非常小的长度尺度,同时具有较重的右尾。当来自 GP 的信号相对于噪声较高时,我们能够使用更具信息性的先验。
lower, upper = 0.5, 5.0
ell_dist, ax = pz.maxent(
pz.LogNormal(),
lower=lower,
upper=upper,
mass=0.9,
plot_kwargs={"support": (0, 7), "legend": None},
)
ax.set_title(r"Prior for the lengthscale, $\ell$")
Text(0.5, 1.0, 'Prior for the lengthscale, $\\ell$')
关于下面的模型代码,有几点需要注意
近似参数
m和c控制近似保真度与计算复杂度的权衡。我们将在后面的章节中看到如何选择这些值。简而言之,选择更大的m有助于改进对较小长度尺度和 GP 必须拟合的其他短距离变化的近似。选择更大的c有助于改进对更长和更慢变化的近似。我们选择
centered参数化是因为真实的底层 GP 强烈地受到数据的告知。您可以在 此处 和 此处 阅读有关中心化与非中心化的更多信息。在HSGP类中,默认值为non-centered,对于可以说更常见的情况,即底层 GP 受到观测数据的弱告知,效果更好。
with pm.Model(coords={"basis_coeffs": np.arange(200), "obs_id": np.arange(len(y_obs))}) as model:
ell = ell_dist.to_pymc("ell")
eta = pm.Exponential("eta", scale=1.0)
cov_func = eta**2 * pm.gp.cov.Matern52(input_dim=1, ls=ell)
# m and c control the fidelity of the approximation
m, c = 200, 1.5
parametrization = "centered"
gp = pm.gp.HSGP(m=[m], c=c, parametrization=parametrization, cov_func=cov_func)
# Compare to the code for the full, unapproximated GP:
# gp = pm.gp.Latent(cov_func=cov_func)
f = gp.prior("f", X=x[:, None], hsgp_coeffs_dims="basis_coeffs", gp_dims="obs_id")
sigma = pm.Exponential("sigma", scale=1.0)
pm.Normal("y_obs", mu=f, sigma=sigma, observed=y_obs, dims="obs_id")
idata = pm.sample()
idata.extend(pm.sample_posterior_predictive(idata, random_seed=rng))
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [ell, eta, f_hsgp_coeffs_, sigma]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 133 seconds.
Sampling: [y_obs]
az.summary(idata, var_names=["eta", "ell", "sigma"], round_to=2)
| 均值 | 标准差 | hdi_3% | hdi_97% | mcse_mean | mcse_sd | ess_bulk | ess_tail | r_hat | |
|---|---|---|---|---|---|---|---|---|---|
| eta | 0.91 | 0.08 | 0.77 | 1.07 | 0.0 | 0.0 | 3468.12 | 3062.29 | 1.0 |
| ell | 0.94 | 0.10 | 0.76 | 1.13 | 0.0 | 0.0 | 1476.96 | 2829.41 | 1.0 |
| sigma | 1.01 | 0.02 | 0.98 | 1.04 | 0.0 | 0.0 | 8891.85 | 3058.98 | 1.0 |
az.plot_trace(
idata,
var_names=["eta", "ell", "sigma"],
lines=[("eta", {}, [1]), ("ell", {}, [1]), ("sigma", {}, [1])],
);
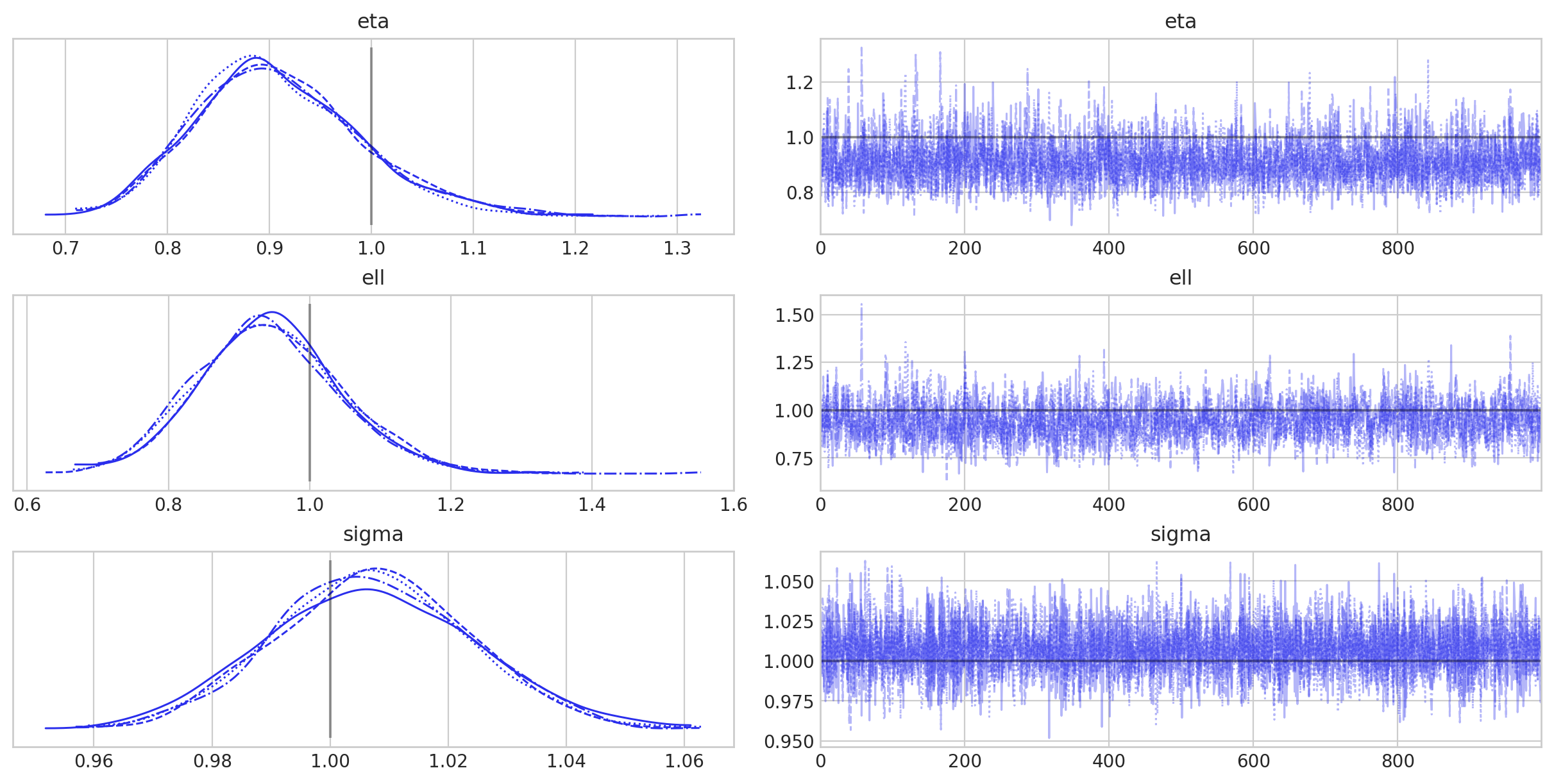
拟合一切顺利,因此我们可以继续绘制推断的 GP 以及后验预测样本。
后验预测图#
fig = plt.figure(figsize=(10, 4))
ax = fig.gca()
f = az.extract(idata.posterior.sel(draw=slice(None, None, 10)), var_names="f")
y_preds = az.extract(idata.posterior_predictive.sel(draw=slice(None, None, 10)), var_names="y_obs")
ax.plot(x, y_preds, color="#AAC4E6", alpha=0.02)
ax.plot(x, f, color="#70133A", alpha=0.1)
ax.scatter(x, y_obs, marker="o", color="grey", s=15, label="Observed data")
ax.plot(x, f_true, color="#FBE64D", lw=2, label="True underlying GP 'f'")
ax.set(title="The HSGP Fit", xlabel="x", ylabel="y")
ax.legend(frameon=True, fontsize=11, ncol=2);
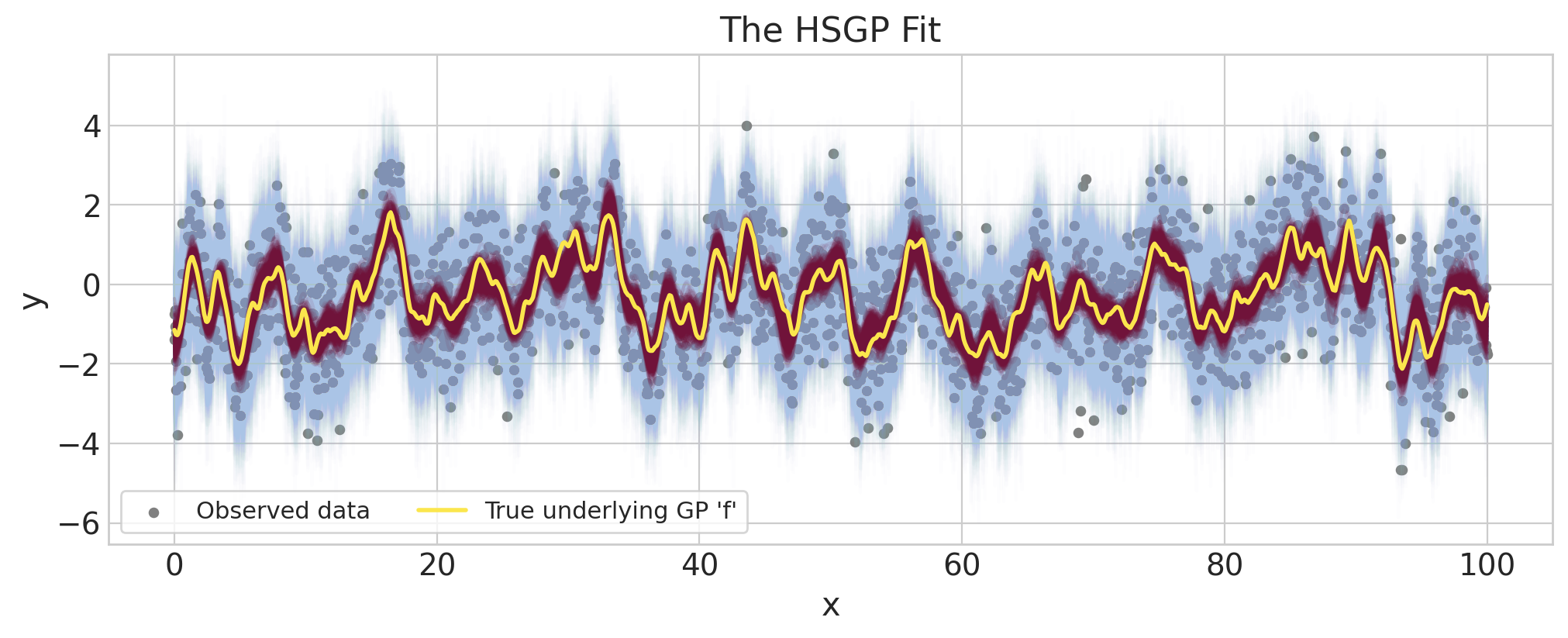
推断的底层 GP(波尔多色)准确地匹配了真实的底层 GP(黄色)。我们还看到后验预测样本(浅蓝色)非常适合观测数据。
加性 GP
HSGP 与加性协方差兼容,而不是定义两个完全独立的 HSGP。
不是构造然后直接相加它们,而是可以通过首先取它们的功率谱密度之和,然后从组合的功率谱密度创建一个 GP 来更有效地计算两个 HSGP 的和。这减少了未知参数的数量,因为两个 GP 可以共享相同的基集。
此代码看起来类似于
cov1 = eta1**2 * pm.gp.cov.ExpQuad(input_dim, ls=ell1)
cov2 = eta2**2 * pm.gp.cov.Matern32(input_dim, ls=ell2)
cov = cov1 + cov2
gp = pm.gp.HSGP(m=[m], c=c, cov_func=cov_func)
选择 HSGP 近似参数,m,L 和 c#
在使用 HSGP 拟合模型之前,您必须选择 m 和 c 或 L。m 是基向量的数量。回想一下,HSGP 近似的计算复杂度是 \(\mathcal{O}(mn + m)\),其中 \(n\) 是数据点的数量。
此选择是以下三个考虑因素之间的平衡
近似的准确性。
减少计算负担。
需要进行预测或预测的
X位置。
在本节末尾,我们将给出 Ruitort-Mayol et. al. 中给出的经验法则。理解如何选择这些参数的最佳方法是理解 m、c 和 L 彼此之间的关系,这需要更多地了解近似在底层是如何工作的。
L 和 c 如何影响基#
非技术性地讲,HSGP 将 GP 先验近似为正弦波的线性组合。线性组合的系数是 IID 正态随机变量,其标准差取决于 GP 超参数(对于 Matern 族,它是幅度和长度尺度)。
为了看到这一点,我们将绘制一些 \(m=3\) 和 \(m=5\) 基向量的图,并仔细注意它们在域边界处的行为。请注意,我们必须首先中心化 x 数据,然后相对于中心化数据选择 L。值得一提的是,我们绘制的基向量不依赖于协方差核的选择,也不依赖于协方差函数具有的任何未知参数。
# Our data goes from x=-5 to x=5
x = np.linspace(-5, 5, 1000)
# (plotting code)
fig, axs = plt.subplots(1, 3, figsize=(14, 4), sharey=True, constrained_layout=True)
ylim = 0.55
axs[0].set_ylim([-ylim, ylim])
axs[1].set_yticks([])
axs[1].set_xlabel("xs (mean subtracted x)")
axs[2].set_yticks([])
# change L as we create the basis vectors
L_options = [5.0, 6.0, 20.0]
m_options = [3, 3, 5]
for i, ax in enumerate(axs.flatten()):
L = L_options[i]
m = m_options[i]
eigvals = pm.gp.hsgp_approx.calc_eigenvalues(pt.as_tensor([L]), [m])
phi = pm.gp.hsgp_approx.calc_eigenvectors(
x[:, None],
pt.as_tensor([L]),
eigvals,
[m],
).eval()
for j in range(phi.shape[1]):
ax.plot(x, phi[:, j])
ax.set_xticks(np.arange(-5, 6, 5))
S = 5.0
c = L / S
ax.text(-4.9, -0.45, f"L = {L}\nc = {c}", fontsize=15)
ax.set(title=f"{m} basis functions")
plt.suptitle("The effect of changing $L$ on the HSGP basis vectors", fontsize=18);
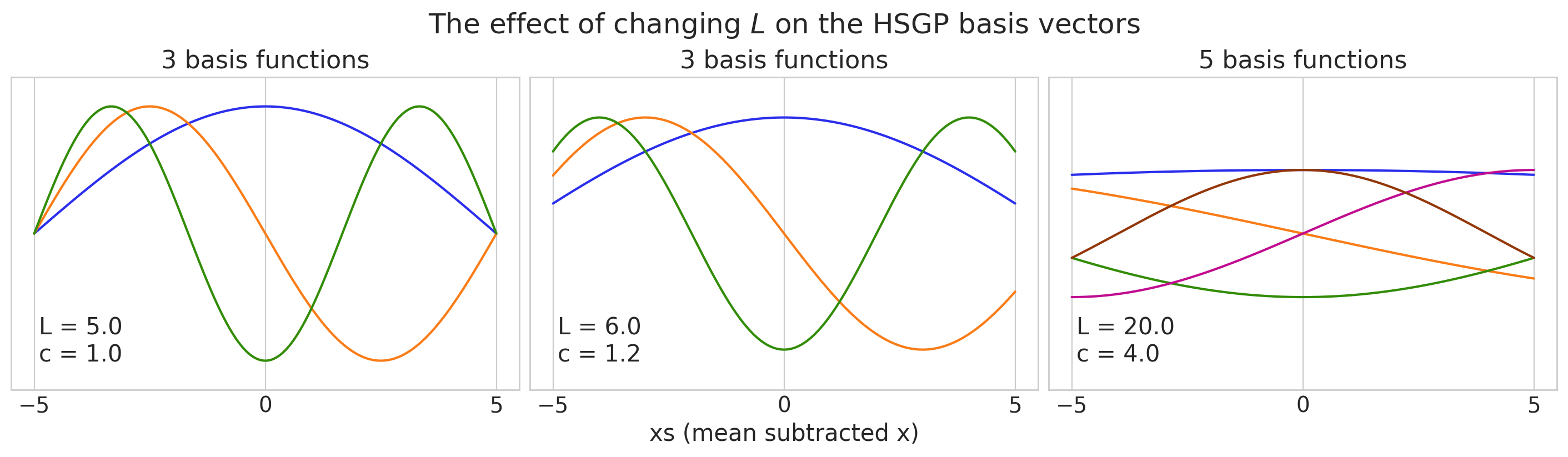
第一个和中间面板有 3 个基函数,最右边有 5 个。
请注意,L 和 m 都被指定为列表,以允许按输入维度设置 L 和 m。在本例中,这些都是单元素列表,因为我们的示例是在一维的、类似时间序列的上下文中。在继续之前,定义 \(S\) 作为中心化数据的半程范围,或从 \(x=0\) 的中点到边缘 \(x=5\) 的距离,这很有帮助。在本例中,每个绘图面板的 \(S=5\)。然后,我们可以定义 \(c\),使其将 \(S\) 与 \(L\) 相关联,
在最左边的图中,我们选择了 \(L=S=5\),这恰好位于我们 x 位置的边缘。对于任何 \(m\),所有基向量都被迫在边缘 \(x=-5\) 和 \(x=5\) 处收缩为零。这意味着当您接近 \(x=-5\) 和 \(x=5\) 时,HSGP 近似会变得很差。多快取决于长度尺度。较大的长度尺度需要较大的 \(L\) 和 \(c\) 值,而较小的长度尺度会减轻此问题。Ruitort-Mayol et. al. 建议使用 1.2 作为最小值。中心面板中显示了此选择对基向量的影响。特别是,我们现在可以看到基向量没有被迫收缩为零。
右侧面板显示了选择更大的 \(L\) 或设置 \(c=4\) 的效果。较大的 \(L\) 或 \(c\) 值使边界条件的问题减少,并且需要准确地近似具有较长长度尺度的 GP。您还需要考虑需要在哪里进行预测。除了观测到的 \(x\) 值的位置之外,新的 \(x\) 位置也需要远离边界条件引起的“收缩”。基函数的周期也随着我们增加 \(L\) 或 \(c\) 而增加。这意味着,如果我们希望近似具有较小长度尺度的 GP,则我们需要增加 \(m\),即基向量的数量,以进行补偿。
对于较大的 \(L\) 或 \(c\),第一个特征向量可能会变得非常平坦,以至于它与模型中的截距部分或完全无法识别。最右边的面板是这种情况的一个示例(请参见蓝色基向量)。在这些情况下,删除第一个特征向量对采样非常有益。PyMC 中的 HSGP 和 HSGPPeriodic 类都具有 drop_first 选项来执行此操作,或者如果您使用 .prior_linearized,则可以自己控制它。如果采样器遇到问题,请务必检查基向量。
总结一下
增加 \(m\) 有助于 HSGP 近似具有较小长度尺度的 GP,但代价是增加了计算复杂度。
增加 \(c\) 或 \(L\) 有助于 HSGP 近似具有较大长度尺度的 GP,但可能需要增加 \(m\) 以补偿较小长度尺度下保真度的损失。
在选择 \(m\)、\(c\) 或 \(L\) 时,重要的是要考虑您需要在哪里进行预测的位置,以便它们也不受边界条件的影响。
第一个基向量可能与截距无法识别,尤其是在 \(L\) 或 \(c\) 较大时。
选择 \(m\) 和 \(c\) 的启发法#
在实践中,您需要从数据中推断长度尺度,因此 HSGP 需要在代表您选择的先验的长度尺度范围内近似 GP。您需要选择足够大的 \(c\) 来处理您可能拟合的最大长度尺度,并且还需要选择足够大的 \(m\) 来容纳最小的长度尺度。
Ruitort-Mayol et. al. 给出了一些方便的启发法,用于给定 \(m\) 和 \(c\) 值的准确再现的长度尺度范围。下面,我们提供了一个函数,该函数使用他们的启发法来推荐最小的 \(m\) 和 \(c\) 值。请注意,这些建议基于一维 GP。
例如,如果您使用 Matern52 协方差,并且您的数据范围从 \(x=-5\) 到 \(x=95\),并且您的长度尺度先验的大部分介于 \(\ell=1\) 和 \(\ell=50\) 之间,那么最小的推荐值是 \(m=543\) 和 \(c=3.7\),如下所示
m, c = pm.gp.hsgp_approx.approx_hsgp_hyperparams(
x_range=[-5, 95], lengthscale_range=[1, 50], cov_func="matern52"
)
print("Recommended smallest number of basis vectors (m):", m)
print("Recommended smallest scaling factor (c):", np.round(c, 1))
Recommended smallest number of basis vectors (m): 543
Recommended smallest scaling factor (c): 4.1
HSGP 近似格拉姆矩阵#
您可能无法依赖这些启发法的原因有几个。您可能正在使用与 ExpQuad、Matern52 或 Matern32 不同的协方差函数。此外,它们仅针对一维 GP 定义。检查 HSGP 保真度的另一种方法是直接比较未近似的格拉姆矩阵(格拉姆矩阵是在输入 X 上计算协方差函数后获得的矩阵),\(\mathbf{K}\),与 HSGP 近似产生的格拉姆矩阵,
X 网格的示例。重要的是要注意,HSGP 近似要求我们中心化输入 X 数据,这通过在下面的代码中将 X 转换为 Xs 来完成。我们绘制不同 \(L\) 和 \(c\) 值的近似格拉姆矩阵,以查看对于给定的 X 位置和长度尺度选择,近似何时开始退化。
## Define the X locations and calculate the Gram matrix from a given covariance function
x1, x2 = np.meshgrid(np.linspace(0, 10, 50), np.linspace(0, 10, 4))
X = np.vstack((x2.flatten(), x1.flatten())).T
# X is two dimensional, so we set input_dim=2
chosen_ell = 3.0
cov_func = pm.gp.cov.ExpQuad(input_dim=2, ls=chosen_ell)
K = cov_func(X).eval()
## Calculate the HSGP approximate Gram matrix
# Center or "scale" X so we can work with Xs (important)
X_center = (np.max(X, axis=0) + np.min(X, axis=0)) / 2.0
Xs = X - X_center
# Calculate L given Xs and c
m, c = [20, 20], 2.0
L = pm.gp.hsgp_approx.set_boundary(Xs, c)
def calculate_Kapprox(Xs, L, m):
# Calculate Phi and the diagonal matrix of power spectral densities
eigvals = pm.gp.hsgp_approx.calc_eigenvalues(L, m)
phi = pm.gp.hsgp_approx.calc_eigenvectors(Xs, L, eigvals, m)
omega = pt.sqrt(eigvals)
psd = cov_func.power_spectral_density(omega)
return (phi @ pt.diag(psd) @ phi.T).eval()
fig, axs = plt.subplots(2, 4, figsize=(14, 7), sharey=True)
axs[0, 0].imshow(K, cmap="inferno", vmin=0, vmax=1)
axs[0, 0].set(xlabel="x1", ylabel="x2", title=f"True Gram matrix\nTrue $\\ell$ = {chosen_ell}")
axs[1, 0].axis("off")
im_kwargs = {
"cmap": "inferno",
"vmin": 0,
"vmax": 1,
"interpolation": "none",
}
## column 1
m, c = [30, 30], 5.0
L = pm.gp.hsgp_approx.set_boundary(Xs, c)
K_approx = calculate_Kapprox(Xs, L, m)
axs[0, 1].imshow(K_approx, **im_kwargs)
axs[0, 1].set_title(f"m = {m}, c = {c}")
m, c = [30, 30], 1.2
L = pm.gp.hsgp_approx.set_boundary(Xs, c)
K_approx = calculate_Kapprox(Xs, L, m)
axs[1, 1].imshow(K_approx, **im_kwargs)
axs[1, 1].set(xlabel="x1", ylabel="x2", title=f"m = {m}, c = {c}")
## column 2
m, c = [15, 15], 5.0
L = pm.gp.hsgp_approx.set_boundary(Xs, c)
K_approx = calculate_Kapprox(Xs, L, m)
axs[0, 2].imshow(K_approx, **im_kwargs)
axs[0, 2].set_title(f"m = {m}, c = {c}")
m, c = [15, 15], 1.2
L = pm.gp.hsgp_approx.set_boundary(Xs, c)
K_approx = calculate_Kapprox(Xs, L, m)
axs[1, 2].imshow(K_approx, **im_kwargs)
axs[1, 2].set_title(f"m = {m}, c = {c}")
## column 3
m, c = [2, 2], 5.0
L = pm.gp.hsgp_approx.set_boundary(Xs, c)
K_approx = calculate_Kapprox(Xs, L, m)
axs[0, 3].imshow(K_approx, **im_kwargs)
axs[0, 3].set_title(f"m = {m}, c = {c}")
m, c = [2, 2], 1.2
L = pm.gp.hsgp_approx.set_boundary(Xs, c)
K_approx = calculate_Kapprox(Xs, L, m)
axs[1, 3].imshow(K_approx, **im_kwargs)
axs[1, 3].set_title(f"m = {m}, c = {c}")
for ax in axs.flatten():
ax.grid(False)
plt.tight_layout();
/tmp/ipykernel_21840/3948114812.py:54: UserWarning: The figure layout has changed to tight
plt.tight_layout();
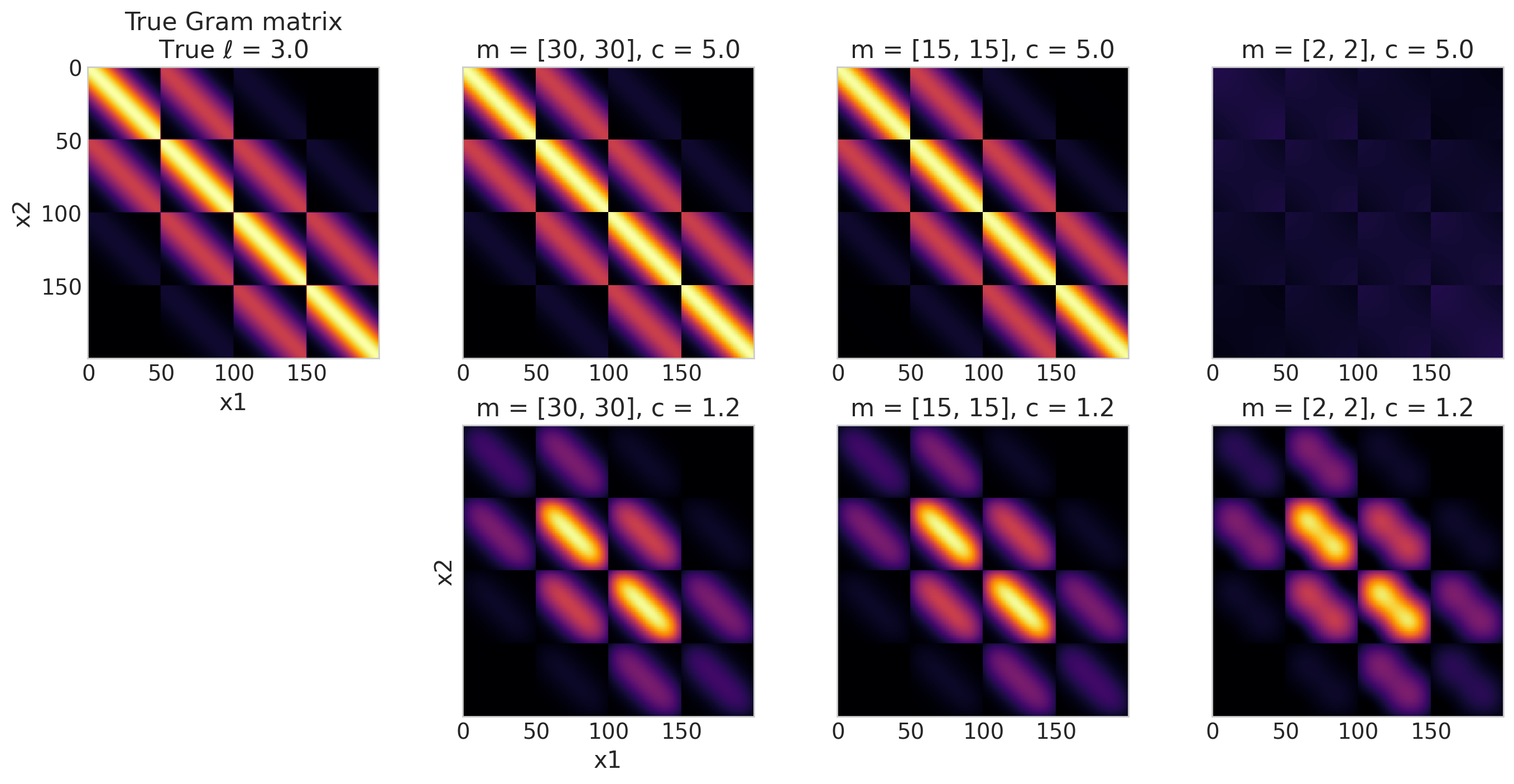
上面的图将近似格拉姆矩阵与左上角面板中未近似的格拉姆矩阵进行比较。目标是将近似的格拉姆矩阵与真实的格拉姆矩阵(左上角)进行比较。定性地,它们看起来越相似,近似效果越好。此外,这些结果仅与 X 和选择的长度尺度 \(\ell = 3\) 定义的特定域的上下文相关——仅仅因为它对于 \(\ell = 3\) 看起来不错并不意味着它对于例如 \(\ell = 10\) 看起来不错。
我们可以做一些观察
对于 \(m = 15\) 或 \(m = 30\) 以及 \(c=5.0\) 的两个面板,近似在视觉上看起来不错。其余的显示出与未近似的格拉姆矩阵的明显差异。
无论 \(m\) 如何,\(c=1.2\) 通常都太小。
也许令人惊讶的是,\(m=[2, 2]\),\(c=1.2\) 近似看起来比 \(m=[2, 2]\),\(c=5\) 近似更好。正如我们前面展示的那样,当我们“拉伸”特征向量基以填充比我们的
X更大的域(大 \(c\) 倍)时,我们可能会在较小的长度尺度上失去保真度。换句话说,在第二种情况下。\(m\) 对于 \(c\) 的值来说太小了。这就是为什么第一个选项看起来更好的原因。第二行 (\(c=1.2\)) 并没有随着 \(m\) 的增加而真正改善。那是因为 \(m\) 足够好以捕获较小的长度尺度,但是 \(c\) 总是太小而无法捕获较大的长度尺度。
另一方面,第一行显示 \(c=5\) 对于较大的长度尺度来说足够好,并且一旦我们达到 \(m=15\),我们也能够捕获较小的长度尺度。
对于您的特定情况,您将需要在您的长度尺度范围内进行实验,并量化多少近似误差是可以接受的。通常,在原型化模型时,您可以使用较低保真度的 HSGP 近似以加快采样速度。然后,一旦您了解了相关长度尺度的范围,就可以调整正确的 \(m\) 和 \(L\)(或 \(c\))值。
请注意,也可能遇到低保真度 HSGP 近似比高保真度 HSGP 近似给出更简约拟合的情况。低保真度 HSGP 近似仍然是某个未知函数的有效先验,即使有点牵强。这是否重要将取决于您的上下文。
示例 2:将 HSGP 用作参数线性模型#
HSGP 近似的主要优势之一是能够将其集成到现有模型中,特别是如果您需要在采样后在新 x 位置进行预测。与 PyMC 中的其他 GP 实现不同,您可以绕过 .prior 和 .conditional API,而是使用 HSGP.prior_linearized,这允许您使用 pm.Data 和 pm.set_data 进行预测。
如果您对以下内容感兴趣,请参阅本节
查看具有模型中其他预测变量的二维或空间 HSGP 示例。
在更大的 PyMC 模型中使用 HSGP 进行预测。
将您的 HSGP 近似转换为 HSTP 近似,或 TP 或学生 t 过程的近似。
数据生成#
def simulate_2d(
beta0_true,
beta1_true,
ell_true,
eta_true,
sigma_true,
):
# Create the 2d X locations
from scipy.stats import qmc
sampler = qmc.Sobol(d=2, scramble=False, optimization="lloyd")
X = 20 * sampler.random_base2(m=11) - 10.0
# add the fixed effect at specific intervals
ix = 1.0 * (np.abs(X[:, 0] // 5) == 1)
X = np.hstack((X, ix[:, None]))
# Draw one sample from the underlying GP
n = X.shape[0]
cov_func = eta_true**2 * pm.gp.cov.Matern52(3, ell_true, active_dims=[0, 1])
gp_true = pm.MvNormal.dist(mu=np.zeros(n), cov=cov_func(X))
f_true = pm.draw(gp_true, draws=1, random_seed=rng)
# Add the fixed effects
mu = beta0_true + beta1_true * X[:, 2] + f_true
# The observed data is the latent function plus a small amount
# of Gaussian distributed noise.
noise_dist = pm.Normal.dist(mu=0.0, sigma=sigma_true)
y_obs = mu + pm.draw(noise_dist, draws=n, random_seed=rng)
return y_obs, f_true, mu, X
y_obs, f_true, mu, X = simulate_2d(
beta0_true=3.0,
beta1_true=2.0,
ell_true=1.0,
eta_true=1.0,
sigma_true=0.75,
)
# Split into train and test sets
ix_tr = (X[:, 1] < 2) | (X[:, 1] > 4)
ix_te = (X[:, 1] > 2) & (X[:, 1] < 4)
X_tr, X_te = X[ix_tr, :], X[ix_te, :]
y_tr, y_te = y_obs[ix_tr], y_obs[ix_te]
fig = plt.figure(figsize=(13, 4))
plt.subplots_adjust(wspace=0.02)
ax1 = plt.subplot(131)
ax1.scatter(X_tr[:, 0], X_tr[:, 1], c=mu[ix_tr] - f_true[ix_tr])
ax1.set_title("$\\beta_0 + \\beta_1 X$")
ax1.set_ylabel("$x_1$", rotation=0)
ax2 = plt.subplot(132)
ax2.scatter(X_tr[:, 0], X_tr[:, 1], c=f_true[ix_tr])
ax2.set_title("The spatial GP, $f$")
ax2.set_yticks([])
ax2.set_xlabel("$x_0$")
ax3 = plt.subplot(133)
im = ax3.scatter(X_tr[:, 0], X_tr[:, 1], c=y_obs[ix_tr])
ax3.set_title("The observed data, $y$")
ax3.set_yticks([])
fig.colorbar(im, ax=[ax1, ax2, ax3]);
/tmp/ipykernel_21840/2813859405.py:2: UserWarning: This figure was using a layout engine that is incompatible with subplots_adjust and/or tight_layout; not calling subplots_adjust.
plt.subplots_adjust(wspace=0.02)

正如预期的那样,我们清楚地看到测试集位于 \(2 < x1 < 4\) 的区域中。
这是与我们的生成场景相对应的模型结构。下面我们描述其主要组成部分。
模型结构#
with pm.Model() as model:
# Set mutable data
X_gp = pm.Data("X_gp", X_tr[:, :2])
X_fe = pm.Data("X_fe", X_tr[:, 2])
# Priors on regression coefficients
beta = pm.Normal("beta", mu=0.0, sigma=10.0, shape=2)
# Prior on the HSGP
eta = pm.Exponential("eta", scale=2.0)
ell_params = pm.find_constrained_prior(
pm.Lognormal, lower=0.5, upper=5.0, mass=0.9, init_guess={"mu": 1.0, "sigma": 1.0}
)
ell = pm.Lognormal("ell", **ell_params)
cov_func = eta**2 * pm.gp.cov.Matern52(input_dim=2, ls=ell)
# m and c control the fidelity of the approximation
m0, m1, c = 30, 30, 2.0
gp = pm.gp.HSGP(m=[m0, m1], c=c, cov_func=cov_func)
phi, sqrt_psd = gp.prior_linearized(X=X_gp)
basis_coeffs = pm.Normal("basis_coeffs", size=gp.n_basis_vectors)
f = pm.Deterministic("f", phi @ (basis_coeffs * sqrt_psd))
mu = pm.Deterministic("mu", beta[0] + beta[1] * X_fe + f)
sigma = pm.Exponential("sigma", scale=2.0)
pm.Normal("y_obs", mu=mu, sigma=sigma, observed=y_tr, shape=X_gp.shape[0])
idata = pm.sample_prior_predictive(random_seed=rng)
pm.model_to_graphviz(model)
Sampling: [basis_coeffs, beta, ell, eta, sigma, y_obs]
在采样并查看结果之前,模型上方有几件事需要注意。
设置系数,中心化和非中心化#
首先,prior_linearized 返回特征向量基,phi,以及特征值处功率谱的平方根,sqrt_psd。您必须从中构建 HSGP 近似。以下是相关的代码行,显示了中心化和非中心化参数化。
phi, sqrt_psd = gp.prior_linearized(X=X)
## non-centered
basis_coeffs= pm.Normal("basis_coeffs", size=gp.n_basis_vectors)
f = pm.Deterministic("f", phi @ (beta * sqrt_psd))
## centered
basis_coeffs= pm.Normal("basis_coeffs", sigma=sqrt_psd, size=gp.n_basis_vectors)
f = pm.Deterministic("f", phi @ beta)
务必使用 HSGP 对象的 n_basis_vectors 属性(或 phi 的列数)设置 basis_coeffs 的大小,\(m^* = \prod_i m_i\)。在上面的示例中,\(m^* = 30 \cdot 30 = 900\),并且是近似中使用的基向量总数。
近似 TP 而不是 GP#
我们可以稍微修改上面的代码以获得 Student-t 过程,
nu = pm.Gamma("nu", alpha=2, beta=0.1)
basis_coeffs= pm.StudentT("basis_coeffs", nu=nu, size=gp.n_basis_vectors)
f = pm.Deterministic("f", phi @ (beta * sqrt_psd))
其中我们对 \(\nu\) 使用 \(\text{Gamma}(\alpha=2, \beta=0.1)\) 先验,这使得大约 50% 的概率 \(\nu > 30\),此时 Student-T 与高斯分布大致无法区分。有关更多信息,请参见此链接。
结果#
现在,让我们对模型进行采样并快速检查结果
with model:
idata.extend(pm.sample(nuts_sampler="numpyro", random_seed=rng))
2024-08-17 10:55:34.390278: E external/xla/xla/service/slow_operation_alarm.cc:65] Constant folding an instruction is taking > 1s:
%reduce = f64[4,1000,900,1]{3,2,1,0} reduce(f64[4,1000,1,900,1]{4,3,2,1,0} %broadcast.8, f64[] %constant.14), dimensions={2}, to_apply=%region_1.45, metadata={op_name="jit(process_fn)/jit(main)/reduce_prod[axes=(2,)]" source_file="/tmp/tmp7cn2oyvr" source_line=33}
This isn't necessarily a bug; constant-folding is inherently a trade-off between compilation time and speed at runtime. XLA has some guards that attempt to keep constant folding from taking too long, but fundamentally you'll always be able to come up with an input program that takes a long time.
If you'd like to file a bug, run with envvar XLA_FLAGS=--xla_dump_to=/tmp/foo and attach the results.
2024-08-17 10:55:36.431977: E external/xla/xla/service/slow_operation_alarm.cc:133] The operation took 3.041937496s
Constant folding an instruction is taking > 1s:
%reduce = f64[4,1000,900,1]{3,2,1,0} reduce(f64[4,1000,1,900,1]{4,3,2,1,0} %broadcast.8, f64[] %constant.14), dimensions={2}, to_apply=%region_1.45, metadata={op_name="jit(process_fn)/jit(main)/reduce_prod[axes=(2,)]" source_file="/tmp/tmp7cn2oyvr" source_line=33}
This isn't necessarily a bug; constant-folding is inherently a trade-off between compilation time and speed at runtime. XLA has some guards that attempt to keep constant folding from taking too long, but fundamentally you'll always be able to come up with an input program that takes a long time.
If you'd like to file a bug, run with envvar XLA_FLAGS=--xla_dump_to=/tmp/foo and attach the results.
idata.sample_stats.diverging.sum().data
array(0)
var_names = [var.name for var in model.free_RVs if var.size.eval() <= 2]
az.summary(idata, var_names=var_names, round_to=2)
| 均值 | 标准差 | hdi_3% | hdi_97% | mcse_mean | mcse_sd | ess_bulk | ess_tail | r_hat | |
|---|---|---|---|---|---|---|---|---|---|
| beta[0] | 2.97 | 0.12 | 2.76 | 3.19 | 0.0 | 0.0 | 6895.09 | 2705.11 | 1.0 |
| beta[1] | 1.97 | 0.11 | 1.76 | 2.17 | 0.0 | 0.0 | 8498.54 | 2687.87 | 1.0 |
| eta | 1.06 | 0.06 | 0.95 | 1.17 | 0.0 | 0.0 | 1601.09 | 2599.12 | 1.0 |
| ell | 0.80 | 0.09 | 0.64 | 0.97 | 0.0 | 0.0 | 2089.67 | 2878.43 | 1.0 |
| sigma | 0.81 | 0.01 | 0.79 | 0.84 | 0.0 | 0.0 | 5583.59 | 3112.06 | 1.0 |
az.plot_trace(
idata,
var_names=var_names,
lines=[("beta", {}, [3, 2]), ("ell", {}, [1]), ("eta", {}, [1]), ("sigma", {}, [0.75])],
);
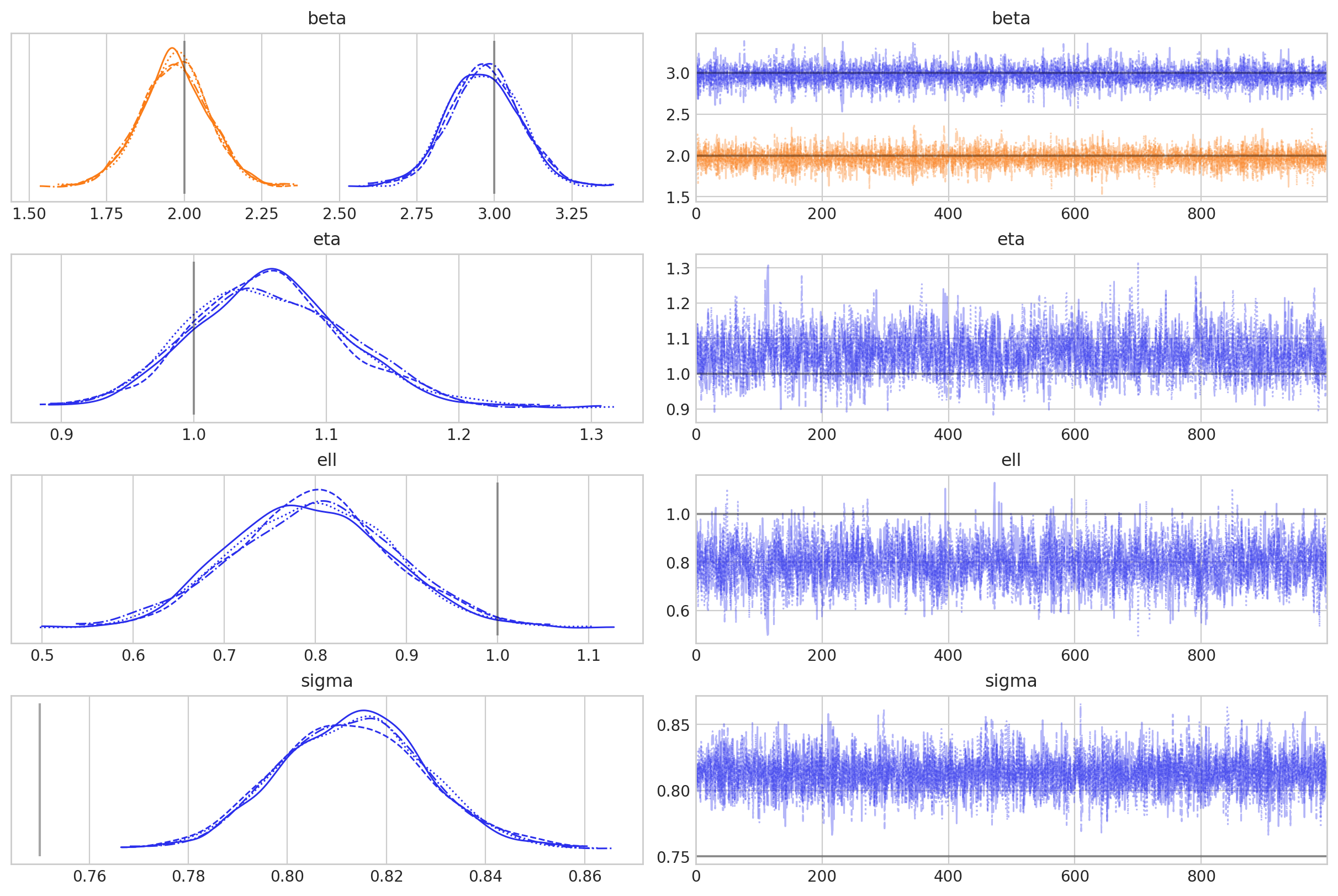
采样进行得很顺利,但有趣的是,我们似乎在 sigma 的后验中存在偏差。 这不是本笔记本的重点,但在实际用例中深入研究它会很有趣。
样本外预测#
然后,我们可以只使用 pm.set_data 在新点进行预测。我们将在下面的图中一起显示拟合和预测。
with model:
pm.set_data({"X_gp": X[:, :2], "X_fe": X[:, 2]})
idata_thinned = idata.sel(draw=slice(None, None, 10))
idata.extend(
pm.sample_posterior_predictive(idata_thinned, var_names=["f", "mu"], random_seed=rng)
)
Sampling: []
pm.model_to_graphviz(model)
fig = plt.figure(figsize=(13, 4))
plt.subplots_adjust(wspace=0.02)
ax1 = plt.subplot(131)
ax1.scatter(X[:, 0], X[:, 1], c=f_true)
ax1.set_title("True underlying GP")
ax1.set_xlabel("$x_0$")
ax1.set_ylabel("$x_1$", rotation=0)
ax2 = plt.subplot(132)
f_sd = az.extract(idata.posterior_predictive, var_names="f").std(dim="sample")
ax2.scatter(X[:, 0], X[:, 1], c=f_sd)
ax2.set_title("Std. dev. of the inferred GP")
ax2.set_yticks([])
ax2.set_xlabel("$x_0$")
ax3 = plt.subplot(133)
f_mu = az.extract(idata.posterior_predictive, var_names="f").mean(dim="sample")
im = ax3.scatter(X[:, 0], X[:, 1], c=f_mu)
ax3.set_title("Mean of the inferred GP")
ax3.set_yticks([])
ax3.set_xlabel("$x_0$")
fig.colorbar(im, ax=[ax1, ax2, ax3]);
/tmp/ipykernel_21840/3852779046.py:2: UserWarning: This figure was using a layout engine that is incompatible with subplots_adjust and/or tight_layout; not calling subplots_adjust.
plt.subplots_adjust(wspace=0.02)
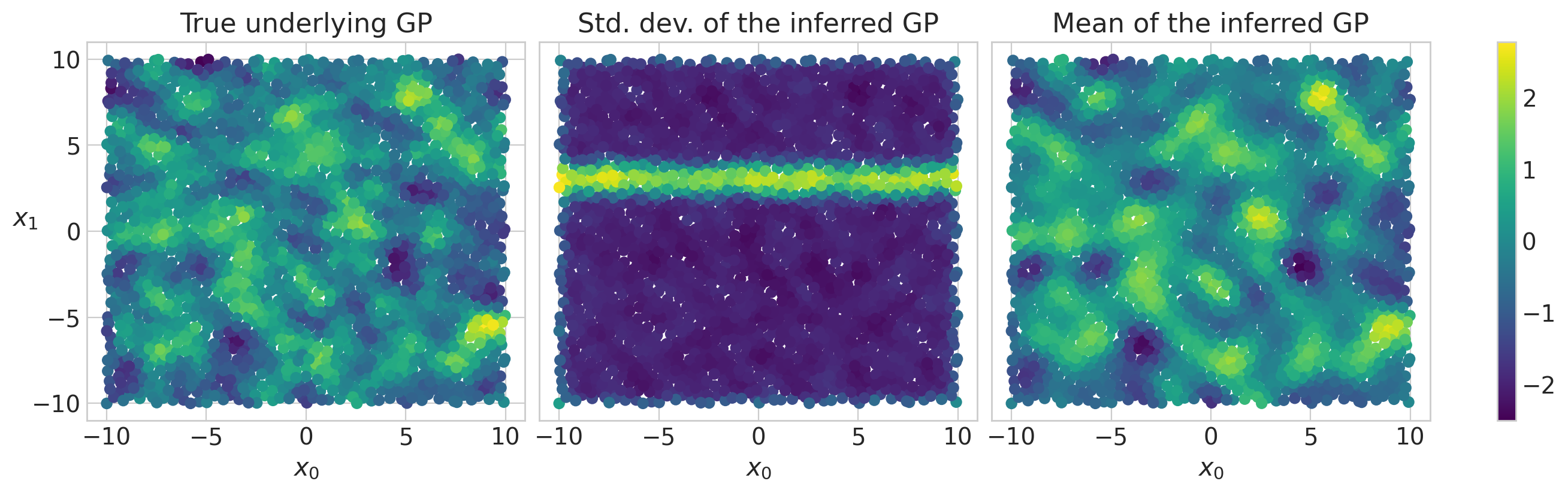
采样诊断看起来都很好,我们可以看到底层 GP 被很好地推断出来。我们还可以看到训练数据之外的不确定性增加,表现为中间面板中的水平条纹,显示此处推断的 GP 的标准偏差增加。
水印#
%load_ext watermark
%watermark -n -u -v -iv -w -p xarray
Last updated: Sat Aug 17 2024
Python implementation: CPython
Python version : 3.11.5
IPython version : 8.16.1
xarray: 2023.10.1
pytensor : 2.25.2
matplotlib: 3.8.4
pymc : 5.16.2+20.g747fda319
numpy : 1.24.4
preliz : 0.9.0
arviz : 0.19.0.dev0
Watermark: 2.4.3
许可声明#
此示例库中的所有笔记本均根据 MIT 许可证 提供,该许可证允许修改和再分发以用于任何用途,前提是版权和许可声明得到保留。
引用 PyMC 示例#
要引用此笔记本,请使用 Zenodo 为 pymc-examples 存储库提供的 DOI。
重要提示
许多笔记本改编自其他来源:博客、书籍…… 在这种情况下,您也应该引用原始来源。
另请记住引用您的代码使用的相关库。
这是一个 bibtex 中的引用模板
@incollection{citekey,
author = "<notebook authors, see above>",
title = "<notebook title>",
editor = "PyMC Team",
booktitle = "PyMC examples",
doi = "10.5281/zenodo.5654871"
}
一旦渲染,它可能看起来像