使用标记的对数高斯 Cox 过程对空间点模式进行建模#
简介#
对数高斯 Cox 过程 (LGCP) 是一种空间或时间中典型观测到的点模式的概率模型。它有两个主要组成部分。首先,使用指数变换的高斯过程在整个域 \(X\) 上对正实数值的潜在强度场 \(\lambda(s)\) 进行建模,这约束了 \(\lambda\) 为正值。然后,此强度场用于参数化泊松点过程,该过程表示在空间中放置点的随机机制。适用于此表示的现象包括一个县的癌症病例发生率,或一个城市中犯罪事件的时空位置。空间和时间维度都可以在此框架内等效处理,尽管本教程仅涉及二维空间中的数据。
更正式地说,如果我们有一个空间 \(X\) 和 \(A\subseteq X\),则子集 \(A\) 内发生的点数 \(Y_A\) 的分布由下式给出
观察到的点模式是否意味着空间强度发生统计显著的偏移?
具有相同统计属性的随机采样模式会是什么样子?
点事件的频率和幅度之间是否存在统计相关性?
在本笔记本中,我们将使用基于网格的近似方法,通过 PyMC 拟合模型并分析其后验摘要。我们还将探讨标记泊松过程的用法,这是此模型的扩展,用于解释与每个数据点关联的标记的分布。
数据#
我们的观测数据涉及 231 个海葵,记录了它们在法国海岸的大小和位置。此数据取自 R 中的 spatstat 空间建模包,该包旨在处理像 LGCP 及其后续改进的模型。此数据的原始来源是 Upton 和 Fingleton (1985) 的教科书Spatial data analysis by example,可以在那里找到对数据的更详细描述。
import warnings
from itertools import product
import arviz as az
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
import pymc as pm
from matplotlib import MatplotlibDeprecationWarning
from numpy.random import default_rng
warnings.filterwarnings(action="ignore", category=MatplotlibDeprecationWarning)
%config InlineBackend.figure_format = 'retina'
az.style.use("arviz-darkgrid")
data = pd.read_csv(pm.get_data("anemones.csv"))
n = data.shape[0]
此数据集具有每个海葵的坐标和离散标记值。虽然这些标记是整数,但为了简单起见,我们将在稍后的步骤中将这些值建模为连续值。
data.head(3)
| x | y | 标记 | |
|---|---|---|---|
| 0 | 27 | 7 | 6 |
| 1 | 197 | 5 | 4 |
| 2 | 74 | 15 | 4 |
让我们在 2D 空间中查看此数据
plt.scatter(data["x"], data["y"], c=data["marks"])
plt.colorbar(label="Anemone size")
plt.axis("equal");
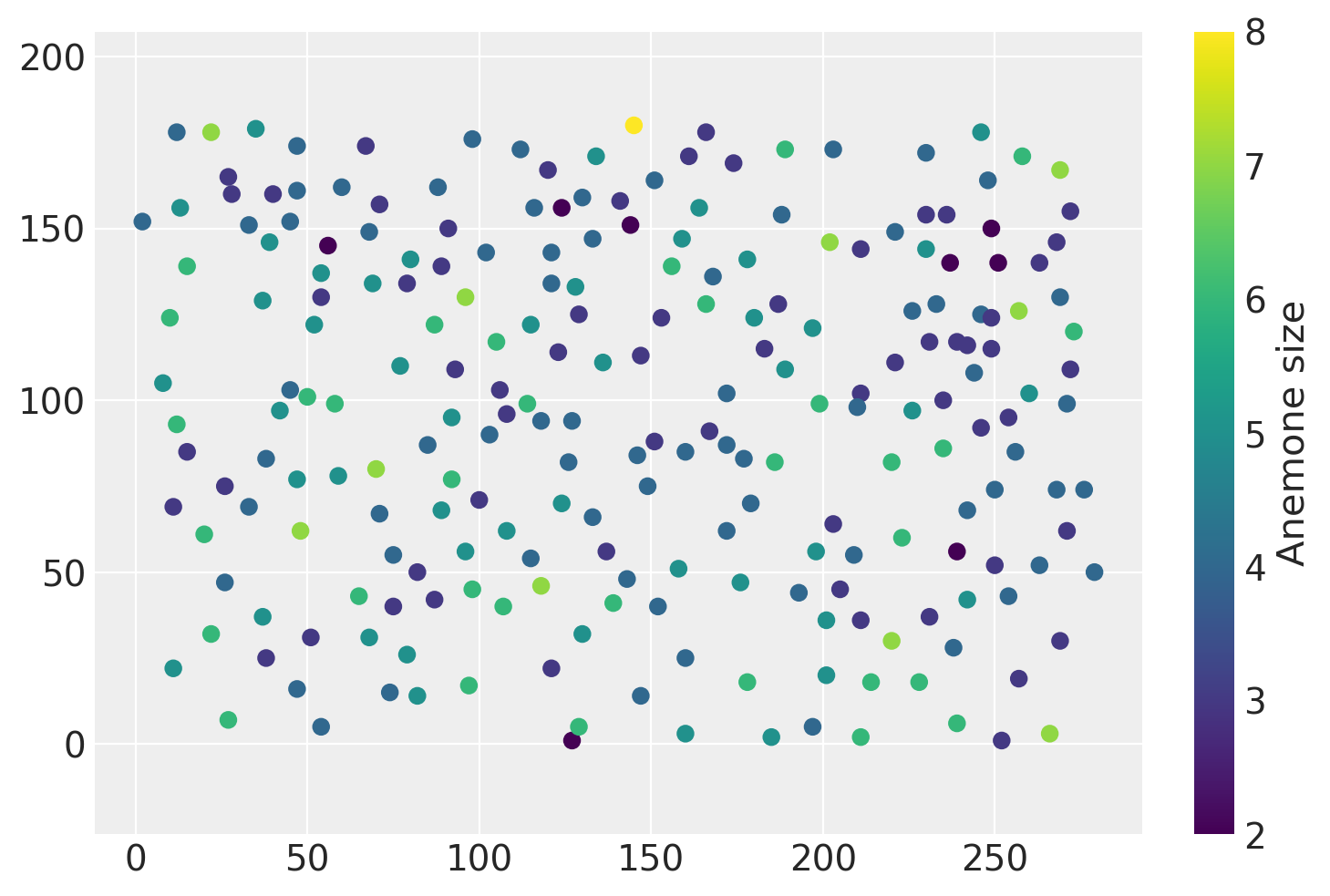
“标记”列指示每个海葵的大小。如果我们要对标记和点的空间分布都进行建模,我们将对标记泊松点过程进行建模。扩展基本点模式模型以包含此特征是本笔记本的第二部分。
虽然有多种进行推断的方法,但也许最简单的方法是将我们的域 \(X\) 切割成许多小块 \(A_1, A_2,...,A_M\),并将强度场固定为在每个子集中恒定。然后,我们将每个 \(A_j\) 内的点数视为泊松随机变量,使得 \(Y_j \sim Poisson(\lambda_j)\)。并且我们还将 \(\log{\lambda_1}...,\log{\lambda_M}\) 变量视为来自高斯过程的单个抽样。
下面的代码将域拆分为网格单元,计算每个单元内的点数,并识别其质心。
xy = data[["x", "y"]].values
# Jitter the data slightly so that none of the points fall exactly
# on cell boundaries
eps = 1e-3
rng = default_rng()
xy = xy.astype("float") + rng.standard_normal(xy.shape) * eps
resolution = 20
# Rescaling the unit of area so that our parameter estimates
# are easier to read
area_per_cell = resolution**2 / 100
cells_x = int(280 / resolution)
cells_y = int(180 / resolution)
# Creating bin edges for a 2D histogram
quadrat_x = np.linspace(0, 280, cells_x + 1)
quadrat_y = np.linspace(0, 180, cells_y + 1)
# Identifying the midpoints of each grid cell
centroids = np.asarray(list(product(quadrat_x[:-1] + 10, quadrat_y[:-1] + 10)))
cell_counts, _, _ = np.histogram2d(xy[:, 0], xy[:, 1], [quadrat_x, quadrat_y])
cell_counts = cell_counts.ravel().astype(int)
将点拆分为不同的单元格并计算单元格质心后,我们可以绘制新的网格化数据集,如下所示。
line_kwargs = {"color": "k", "linewidth": 1, "alpha": 0.5}
plt.figure(figsize=(6, 4.5))
[plt.axhline(y, **line_kwargs) for y in quadrat_y]
[plt.axvline(x, **line_kwargs) for x in quadrat_x]
plt.scatter(data["x"], data["y"], c=data["marks"], s=6)
for i, row in enumerate(centroids):
shifted_row = row - 2
plt.annotate(cell_counts[i], shifted_row, alpha=0.75)
plt.title("Anemone counts per grid cell"), plt.colorbar(label="Anemone size");
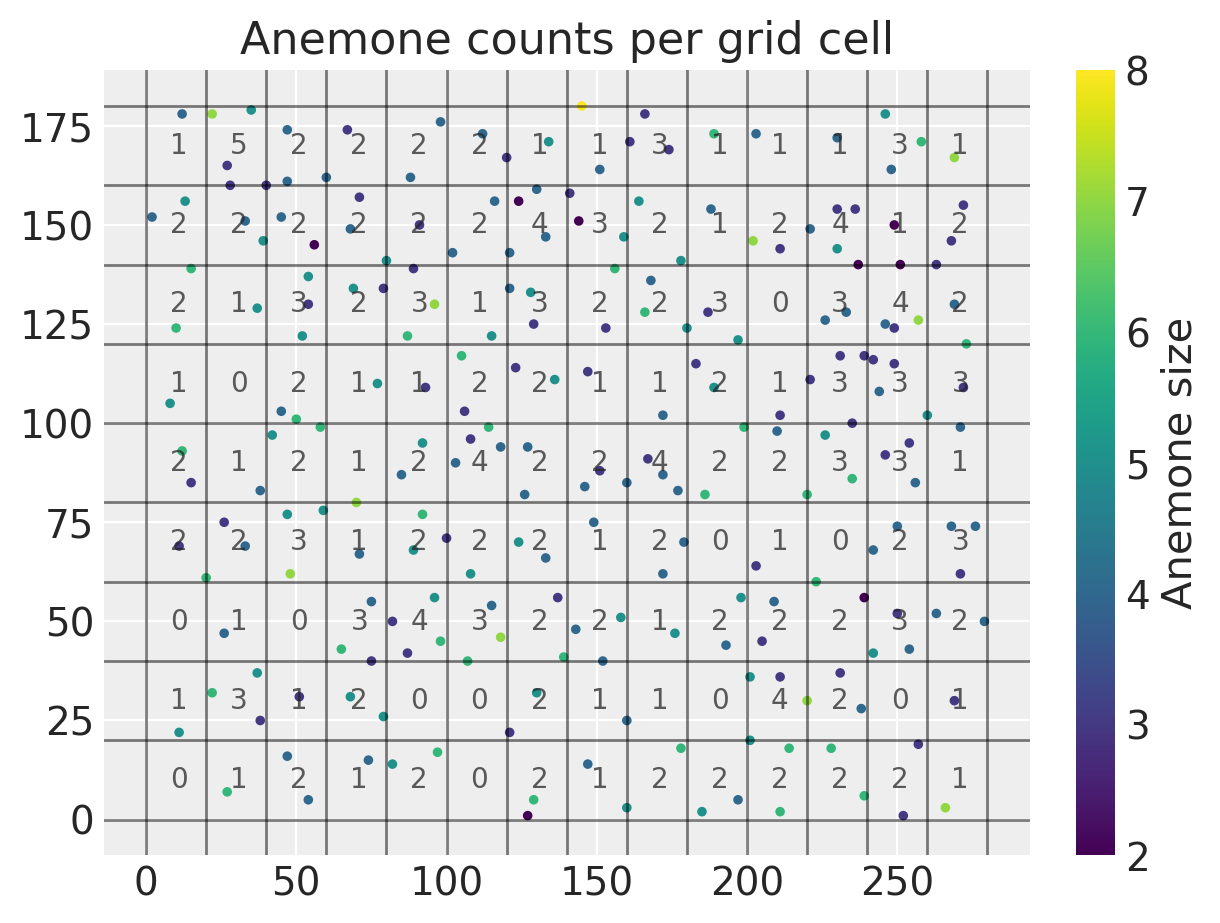
我们可以看到所有计数都相当低,范围从零到五。准备好所有数据后,我们可以继续开始在 PyMC 中编写我们的概率模型。我们将上面的每个单元格计数 \(Y_1,...Y_M\) 视为泊松随机变量。
推断#
我们的第一步是为高斯过程的高级参数设置先验分布。这包括协方差函数的长度尺度 \(\rho\) 和 GP 的常数均值 \(\mu\)。
with pm.Model() as lgcp_model:
mu = pm.Normal("mu", sigma=3)
rho = pm.Uniform("rho", lower=25, upper=300)
variance = pm.InverseGamma("variance", alpha=1, beta=1)
cov_func = variance * pm.gp.cov.Matern52(2, ls=rho)
mean_func = pm.gp.mean.Constant(mu)
接下来,我们通过 pm.math.exp 将高斯过程转换为正值过程,并使用每个单元格的面积将强度函数 \(\lambda(s)\) 转换为速率 \(\lambda_i\),从而参数化单元格 \(i\) 内计数的泊松似然。
with lgcp_model:
gp = pm.gp.Latent(mean_func=mean_func, cov_func=cov_func)
log_intensity = gp.prior("log_intensity", X=centroids)
intensity = pm.math.exp(log_intensity)
rates = intensity * area_per_cell
counts = pm.Poisson("counts", mu=rates, observed=cell_counts)
完全指定模型后,我们可以开始使用默认的 NUTS 采样器从后验进行采样。我还会调整目标接受率以减少散度数量。
with lgcp_model:
trace = pm.sample(1000, tune=2000, target_accept=0.95)
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [mu, rho, variance, log_intensity_rotated_]
Sampling 4 chains for 2_000 tune and 1_000 draw iterations (8_000 + 4_000 draws total) took 8125 seconds.
There were 2 divergences after tuning. Increase `target_accept` or reparameterize.
There was 1 divergence after tuning. Increase `target_accept` or reparameterize.
解释结果#
对 length_scale 参数进行后验推断有助于理解数据中是否存在长程相关性。我们还可以检查对数强度场的均值,但由于它在对数尺度上,因此很难直接解释。
az.summary(trace, var_names=["mu", "rho"])
| 均值 | 标准差 | hdi_3% | hdi_97% | mcse_均值 | mcse_标准差 | ess_bulk | ess_tail | r_hat | |
|---|---|---|---|---|---|---|---|---|---|
| mu | -0.929 | 0.642 | -2.186 | 0.293 | 0.012 | 0.009 | 2825.0 | 2025.0 | 1.0 |
| rho | 243.612 | 44.685 | 161.203 | 299.971 | 0.658 | 0.470 | 4083.0 | 2785.0 | 1.0 |
我们还有兴趣查看空间中大量新点的强度场值。我们可以通过在模型中包含一个新的随机变量来适应这一点,该变量用于在更密集的一组点上评估潜在高斯过程。使用 sample_posterior_predictive,我们生成变量 intensity_new 中包含的新数据点的后验预测。
x_new = np.linspace(5, 275, 20)
y_new = np.linspace(5, 175, 20)
xs, ys = np.asarray(np.meshgrid(x_new, y_new))
xy_new = np.asarray([xs.ravel(), ys.ravel()]).T
with lgcp_model:
intensity_new = gp.conditional("log_intensity_new", Xnew=xy_new)
spp_trace = pm.sample_posterior_predictive(
trace, var_names=["log_intensity_new"], keep_size=True
)
trace.extend(spp_trace)
intensity_samples = np.exp(trace.posterior_predictive["log_intensity_new"])
让我们看一下 \(\lambda(s)\) 的几个实现。由于样本在对数尺度上,我们需要将它们取幂以获得 2D 泊松过程的空间强度场。在下面的图中,叠加了观察到的点模式。
fig, axes = plt.subplots(2, 3, figsize=(8, 5), constrained_layout=True)
axes = axes.ravel()
field_kwargs = {"marker": "o", "edgecolor": "None", "alpha": 0.5, "s": 80}
for i in range(6):
field_handle = axes[i].scatter(
xy_new[:, 0], xy_new[:, 1], c=intensity_samples.sel(chain=0, draw=i), **field_kwargs
)
obs_handle = axes[i].scatter(data["x"], data["y"], s=10, color="k")
axes[i].axis("off")
axes[i].set_title(f"Sample {i}")
plt.figlegend(
(obs_handle, field_handle),
("Observed data", r"Posterior draws of $\lambda(s)$"),
ncol=2,
loc=(0.2, -0.01),
fontsize=14,
frameon=False,
);
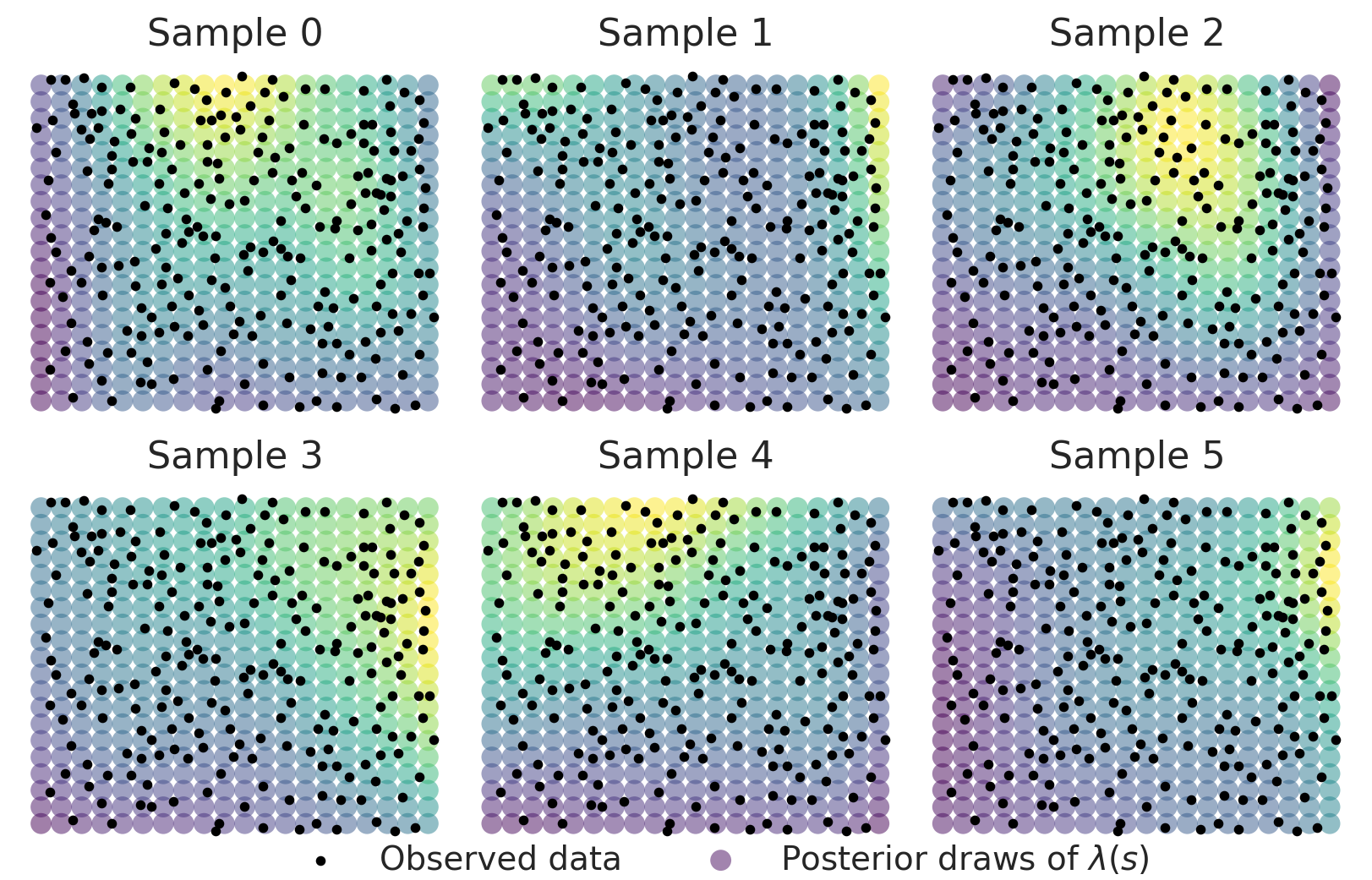
虽然这些表面显示的模式存在一些异质性,但我们获得了后验均值表面,该表面具有非常清晰定义的空间表面,右上角强度较高,左下角强度较低。
fig = plt.figure(figsize=(5, 4))
plt.scatter(
xy_new[:, 0],
xy_new[:, 1],
c=intensity_samples.mean(("chain", "draw")),
marker="o",
alpha=0.75,
s=100,
edgecolor=None,
)
plt.title("$E[\\lambda(s) \\vert Y]$")
plt.colorbar(label="Posterior mean");
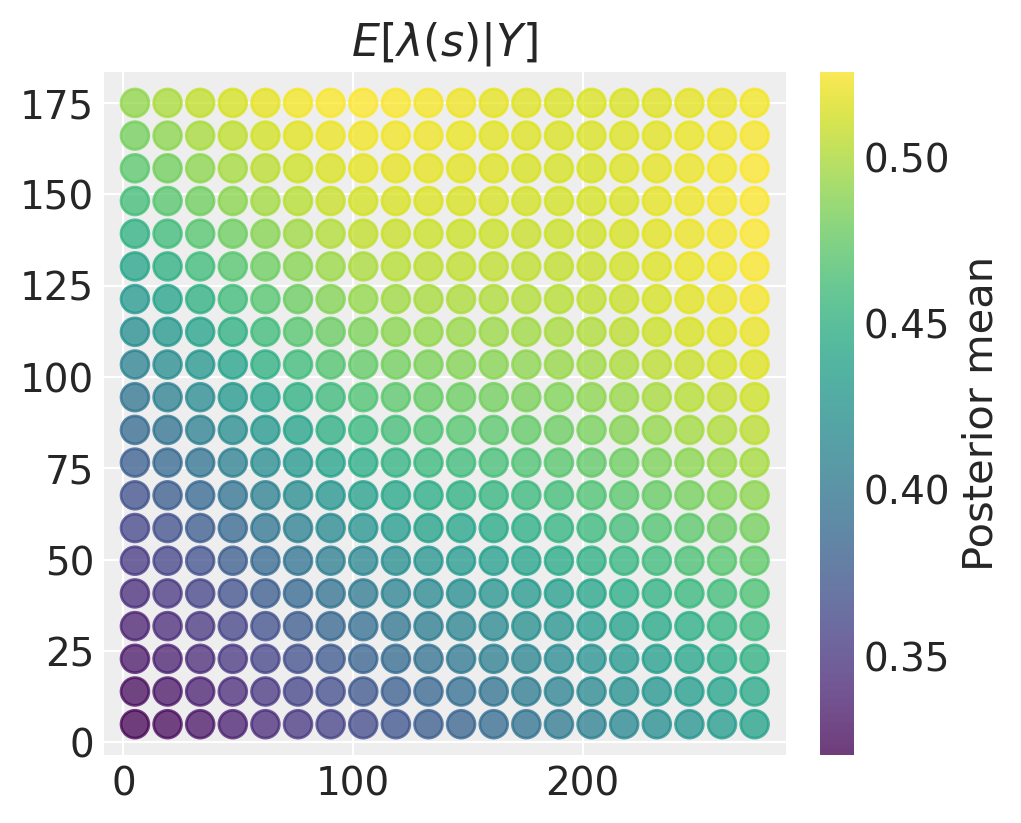
如果存在很多不确定性,那么我们对强度场估计的空间变化可能没有太大意义。在这种情况下,我们可以绘制类似的后验方差(或标准差)图
fig = plt.figure(figsize=(5, 4))
plt.scatter(
xy_new[:, 0],
xy_new[:, 1],
c=intensity_samples.var(("chain", "draw")),
marker="o",
alpha=0.75,
s=100,
edgecolor=None,
)
plt.title("$Var[\\lambda(s) \\vert Y]$"), plt.colorbar();
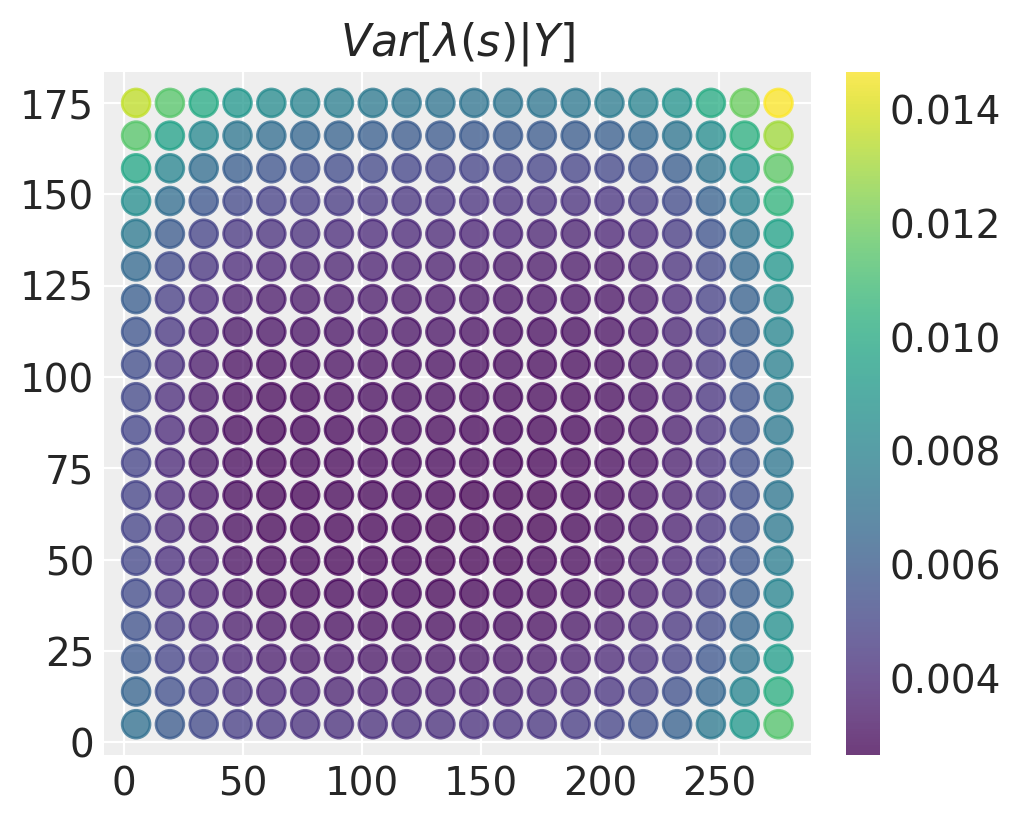
后验方差在域的中间最低,在角落和边缘最大。这是有道理的 - 在数据较多的位置,我们对强度场的值可能是什么有更准确的估计。
水印#
%load_ext watermark
%watermark -n -u -v -iv -w
Last updated: Wed Jun 01 2022
Python implementation: CPython
Python version : 3.9.10
IPython version : 8.1.1
matplotlib: 3.5.1
arviz : 0.12.0
numpy : 1.22.2
pymc : 4.0.0b6
pandas : 1.4.1
sys : 3.9.10 | packaged by conda-forge | (main, Feb 1 2022, 21:24:11)
[GCC 9.4.0]
Watermark: 2.3.0
许可声明#
本示例 галерея 中的所有笔记本均根据 MIT 许可证 提供,该许可证允许修改和再分发以用于任何用途,前提是保留版权和许可声明。
引用 PyMC 示例#
要引用此笔记本,请使用 Zenodo 为 pymc-examples 存储库提供的 DOI。
重要提示
许多笔记本都改编自其他来源:博客、书籍... 在这种情况下,您也应该引用原始来源。
另请记住引用您的代码使用的相关库。
这是一个 bibtex 中的引用模板
@incollection{citekey,
author = "<notebook authors, see above>",
title = "<notebook title>",
editor = "PyMC Team",
booktitle = "PyMC examples",
doi = "10.5281/zenodo.5654871"
}
渲染后可能看起来像