冈比亚疟疾流行率#
导入#
import arviz as az
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
import pymc as pm
import pytensor.tensor as pt
注意
此 notebook 使用的库不是 PyMC 的依赖项,因此需要专门安装才能运行此 notebook。 打开下面的下拉菜单以获取更多指导。
额外的依赖项安装说明
为了运行此 notebook(本地或在 binder 上),您不仅需要安装可用的 PyMC 以及所有可选依赖项,还需要安装一些额外的依赖项。 有关安装 PyMC 本身的建议,请参阅安装
您可以使用您喜欢的包管理器安装这些依赖项,我们提供以下 pip 和 conda 命令作为示例。
$ pip install folium geopandas mapclassify pyproj rasterio
请注意,如果您想(或需要)从 notebook 内部而不是命令行安装软件包,您可以通过运行 pip 命令的变体来安装软件包
import sys
!{sys.executable} -m pip install folium geopandas mapclassify pyproj rasterio
您不应运行 !pip install ,因为它可能会将软件包安装在不同的环境中,即使已安装,也无法从 Jupyter notebook 中使用。
另一种选择是使用 conda 代替
$ conda install folium geopandas mapclassify pyproj rasterio
当使用 conda 安装科学 python 软件包时,我们建议使用 conda forge
# These dependencies need to be installed separately from PyMC
import folium
import geopandas as gpd
import mapclassify
import rasterio as rio
from pyproj import Transformer
简介#
通常,我们发现自己拥有空间相关的连续测量样本(地统计数据),我们的目标是确定未采样周围区域中该测量的估计值。 在以下案例研究中,我们查看了冈比亚地区 65 个村庄样本中疟疾检测呈阳性的人数,并继续估计 65 个采样村庄周围区域内的疟疾流行率(总阳性人数/总检测人数)。
数据处理#
# load the tabular data
try:
gambia = pd.read_csv("../data/gambia_dataset.csv")
except FileNotFoundError:
gambia = pd.read_csv(pm.get_data("gambia_dataset.csv"))
gambia.head()
| Unnamed: 0 | x | y | pos | 年龄 | netuse | treated | green | phc | |
|---|---|---|---|---|---|---|---|---|---|
| 0 | 1850 | 349631.3 | 1458055 | 1 | 1783 | 0 | 0 | 40.85 | 1 |
| 1 | 1851 | 349631.3 | 1458055 | 0 | 404 | 1 | 0 | 40.85 | 1 |
| 2 | 1852 | 349631.3 | 1458055 | 0 | 452 | 1 | 0 | 40.85 | 1 |
| 3 | 1853 | 349631.3 | 1458055 | 1 | 566 | 1 | 0 | 40.85 | 1 |
| 4 | 1854 | 349631.3 | 1458055 | 0 | 598 | 1 | 0 | 40.85 | 1 |
数据目前处于个人层面,但为了我们的目的,我们需要将其置于村庄层面。 我们将按村庄汇总数据,以计算检测总人数、检测呈阳性的人数以及样本流行率; 样本流行率将通过将总检测阳性人数除以总检测人数来计算。
# For each village compute the total tested, total positive, and the prevalence
gambia_agg = (
gambia.groupby(["x", "y"])
.agg(total=("x", "size"), positive=("pos", "sum"))
.eval("prev = positive / total")
.reset_index()
)
gambia_agg.head()
| x | y | total | positive | prev | |
|---|---|---|---|---|---|
| 0 | 349631.3 | 1458055 | 33 | 17 | 0.515152 |
| 1 | 358543.1 | 1460112 | 63 | 19 | 0.301587 |
| 2 | 360308.1 | 1460026 | 17 | 7 | 0.411765 |
| 3 | 363795.7 | 1496919 | 24 | 8 | 0.333333 |
| 4 | 366400.5 | 1460248 | 26 | 10 | 0.384615 |
我们需要将我们的 dataframe 转换为 geodataframe。 为了做到这一点,我们需要知道使用什么坐标参考系统 (CRS),地理坐标系统 (GCS) 或投影坐标系统 (PCS)。 GCS 告诉您您的数据在地球上的位置,而 PCS 告诉您如何在二维平面上绘制您的数据。 存在许多不同的 GCS/PCS,因为每个 GCS/PCS 都是地球表面的模型。 然而,地球表面从一个位置到另一个位置是可变的。 因此,不同的 GCS/PCS 版本将根据您的分析所基于的地理位置而更准确。 由于我们的分析在冈比亚,我们将使用 PCS EPSG 32628 和 GCS EPSG 4326 在地球仪上绘图时。 其中 EPSG 代表欧洲石油调查组,该组织维护坐标系统的测地参数。
# Create a GeoDataframe and set coordinate reference system to EPSG 4326
gambia_gpdf = gpd.GeoDataFrame(
gambia_agg, geometry=gpd.points_from_xy(gambia_agg["x"], gambia_agg["y"]), crs="EPSG:32628"
).drop(["x", "y"], axis=1)
gambia_gpdf_4326 = gambia_gpdf.to_crs(crs="EPSG:4326")
# Get an interactive plot of the data with a cmap on the prevalence values
gambia_gpdf_4326.round(2).explore(column="prev")
我们希望在地图上包含冈比亚境内的海拔高度。 为此,我们提取存储在栅格文件中的海拔值,并将其叠加在地图上。 颜色较深的红色区域表示海拔较高。
# Overlay the raster image of elevations in the Gambia on top of the map
m = gambia_gpdf_4326.round(2).explore(column="prev")
## Load the elevation rasterfile
in_path = "../data/GMB_elv_msk.tif"
dst_crs = "EPSG:4326"
with rio.open(in_path) as src:
img = src.read()
src_crs = str(src.crs)
min_lon, min_lat, max_lon, max_lat = src.bounds
xs = gambia_gpdf_4326["geometry"].x
ys = gambia_gpdf_4326["geometry"].y
rows, cols = rio.transform.rowcol(src.transform, xs, ys)
## Conversion of elevation locations from UTM to WGS84 CRS
bounds_orig = [[min_lat, min_lon], [max_lat, max_lon]]
bounds_fin = []
for item in bounds_orig:
# converting to lat/lon
lat = item[0]
lon = item[1]
proj = Transformer.from_crs(
int(src_crs.split(":")[1]), int(dst_crs.split(":")[1]), always_xy=True
)
lon_n, lat_n = proj.transform(lon, lat)
bounds_fin.append([lat_n, lon_n])
# Finding the center of latitude & longitude
centre_lon = bounds_fin[0][1] + (bounds_fin[1][1] - bounds_fin[0][1]) / 2
centre_lat = bounds_fin[0][0] + (bounds_fin[1][0] - bounds_fin[0][0]) / 2
# Overlay raster
m.add_child(
folium.raster_layers.ImageOverlay(
img.transpose(1, 2, 0),
opacity=0.7,
bounds=bounds_fin,
overlay=True,
control=True,
cross_origin=False,
zindex=1,
colormap=lambda x: (1, 0, 0, x),
)
)
m
/opt/miniconda3/envs/pymc_spatial/lib/python3.12/site-packages/branca/utilities.py:320: RuntimeWarning: invalid value encountered in cast
array = array.astype("uint8")
我们希望将海拔高度作为我们模型中的协变量。 因此,我们需要从栅格图像中提取值并将其存储到 dataframe 中。
# Pull the elevation values from the raster file and put them into a dataframe
path = "../data/GMB_elv_msk.tif"
with rio.open(path) as f:
arr = f.read(1)
mask = arr != f.nodata
elev = arr[mask]
col, row = np.where(mask)
x, y = f.xy(col, row)
uid = np.arange(f.height * f.width).reshape((f.height, f.width))[mask]
result = np.rec.fromarrays([uid, x, y, elev], names=["id", "x", "y", "elev"])
elevations = pd.DataFrame(result)
elevations = gpd.GeoDataFrame(
elevations, geometry=gpd.points_from_xy(elevations["x"], elevations["y"], crs="EPSG:4326")
)
在提取海拔值后,我们需要对汇总的数据集与流行率执行空间连接。 空间连接是一种特殊的连接,它基于地理信息连接数据。 至关重要的是,当您执行此类连接时,您使用的投影坐标系统对于您的地理位置是准确的。
# Set coordinate system to EPSG 32628 and spatially join our prevalence dataframe to our elevations dataframe
elevations = elevations.to_crs(epsg="32628")
gambia_gpdf = gambia_gpdf.sjoin_nearest(elevations, how="inner")
# Set CRS to EPSG 4326 for plotting
gambia_gpdf_4326 = gambia_gpdf.to_crs(crs="EPSG:4326")
gambia_gpdf_4326.head()
| total | positive | prev | geometry | index_right | id | x | y | elev | |
|---|---|---|---|---|---|---|---|---|---|
| 0 | 33 | 17 | 0.515152 | POINT (-16.38755 13.18541) | 12390 | 39649 | -16.387500 | 13.187500 | 15.0 |
| 1 | 63 | 19 | 0.301587 | POINT (-16.30543 13.20444) | 12166 | 38843 | -16.304167 | 13.204167 | 33.0 |
| 2 | 17 | 7 | 0.411765 | POINT (-16.28914 13.20374) | 12168 | 38845 | -16.287500 | 13.204167 | 32.0 |
| 3 | 24 | 8 | 0.333333 | POINT (-16.25869 13.53742) | 3429 | 22528 | -16.262500 | 13.537500 | 20.0 |
| 4 | 26 | 10 | 0.384615 | POINT (-16.23294 13.20603) | 12175 | 38852 | -16.229167 | 13.204167 | 29.0 |
# Get relevant measures for modeling
elev = gambia_gpdf["elev"].values
pos = gambia_gpdf["positive"].values
n = gambia_gpdf["total"].values
lonlat = gambia_gpdf[["y", "x"]].values
# Set a seed for reproducibility of results
seed: int = sum(map(ord, "spatialmalaria"))
rng: np.random.Generator = np.random.default_rng(seed=seed)
模型规范#
我们指定以下模型
其中 \(n_{i}\) 代表接受疟疾检测的个体,\(P(x_{i})\) 是位置 \(x_{i}\) 的疟疾流行率,\(\beta_{0}\) 是截距,\(\beta_{1}\) 是海拔协变量的系数,\(S(x_{i})\) 是零均值场高斯过程,具有 Matérn 协方差函数 \(\nu=\frac{3}{2}\),我们将使用希尔伯特空间高斯过程 (HSGP) 进行近似
为了使用 HSGP 近似高斯过程,我们需要选择参数 m 和 c。 要了解有关如何设置这些参数的更多信息,请参阅这个精彩的(示例)关于如何设置这些参数。
with pm.Model() as hsgp_model:
_X = pm.Data("X", lonlat)
_elev = pm.Data("elevation", elev_std)
ls = pm.Gamma("ls", mu=20, sigma=5)
cov_func = pm.gp.cov.Matern32(2, ls=ls)
m0, m1, c = 40, 40, 2.5
gp = pm.gp.HSGP(m=[m0, m1], c=c, cov_func=cov_func)
s = gp.prior("s", X=_X)
beta_0 = pm.Normal("beta_0", 0, 1)
beta_1 = pm.Normal("beta_1", 0, 1)
p_logit = pm.Deterministic("p_logit", beta_0 + beta_1 * _elev + s)
p = pm.Deterministic("p", pm.math.invlogit(p_logit))
pm.Binomial("likelihood", n=n, logit_p=p_logit, observed=pos)
hsgp_model.to_graphviz()
2024-11-19 18:24:12.156137: E external/xla/xla/service/slow_operation_alarm.cc:65] Constant folding an instruction is taking > 1s:
%reduce = f64[4,1000,1600,1]{3,2,1,0} reduce(f64[4,1000,1,1600,1]{4,3,2,1,0} %broadcast.9, f64[] %constant.15), dimensions={2}, to_apply=%region_1.42, metadata={op_name="jit(process_fn)/jit(main)/reduce_prod[axes=(2,)]" source_file="/var/folders/qv/63yqp4p50630y7pqfgcbgtq80000gn/T/tmpf1zosxas" source_line=25}
This isn't necessarily a bug; constant-folding is inherently a trade-off between compilation time and speed at runtime. XLA has some guards that attempt to keep constant folding from taking too long, but fundamentally you'll always be able to come up with an input program that takes a long time.
If you'd like to file a bug, run with envvar XLA_FLAGS=--xla_dump_to=/tmp/foo and attach the results.
2024-11-19 18:24:33.243058: E external/xla/xla/service/slow_operation_alarm.cc:133] The operation took 22.092144s
Constant folding an instruction is taking > 1s:
%reduce = f64[4,1000,1600,1]{3,2,1,0} reduce(f64[4,1000,1,1600,1]{4,3,2,1,0} %broadcast.9, f64[] %constant.15), dimensions={2}, to_apply=%region_1.42, metadata={op_name="jit(process_fn)/jit(main)/reduce_prod[axes=(2,)]" source_file="/var/folders/qv/63yqp4p50630y7pqfgcbgtq80000gn/T/tmpf1zosxas" source_line=25}
This isn't necessarily a bug; constant-folding is inherently a trade-off between compilation time and speed at runtime. XLA has some guards that attempt to keep constant folding from taking too long, but fundamentally you'll always be able to come up with an input program that takes a long time.
If you'd like to file a bug, run with envvar XLA_FLAGS=--xla_dump_to=/tmp/foo and attach the results.
长度尺度的后验均值为 0.2(如下所示)。 因此,我们可以预期,当我们从地图上的任何采样点移动 0.2 度时,高斯均值将衰减为 0(因为我们设置了 0 均值函数)。 虽然这不是一个硬性截止值,因为长度尺度不受观测数据的约束,但能够直观地了解长度尺度如何影响估计仍然很有用。
az.summary(hsgp_trace, var_names=["ls"], kind="stats")
| 均值 | 标准差 | hdi_3% | hdi_97% | |
|---|---|---|---|---|
| ls | 0.197 | 0.123 | 0.102 | 0.28 |
后验预测检查#
我们需要验证我们的模型规范是否正确表示了观测数据。 我们可以推出流行率的后验预测,并在坐标系上绘制它们,以检查它们是否与我们样本中的观测流行率相似
with hsgp_model:
ppc = pm.sample_posterior_predictive(hsgp_trace, random_seed=rng)
Sampling: [likelihood]
posterior_prevalence = hsgp_trace["posterior"]["p"]
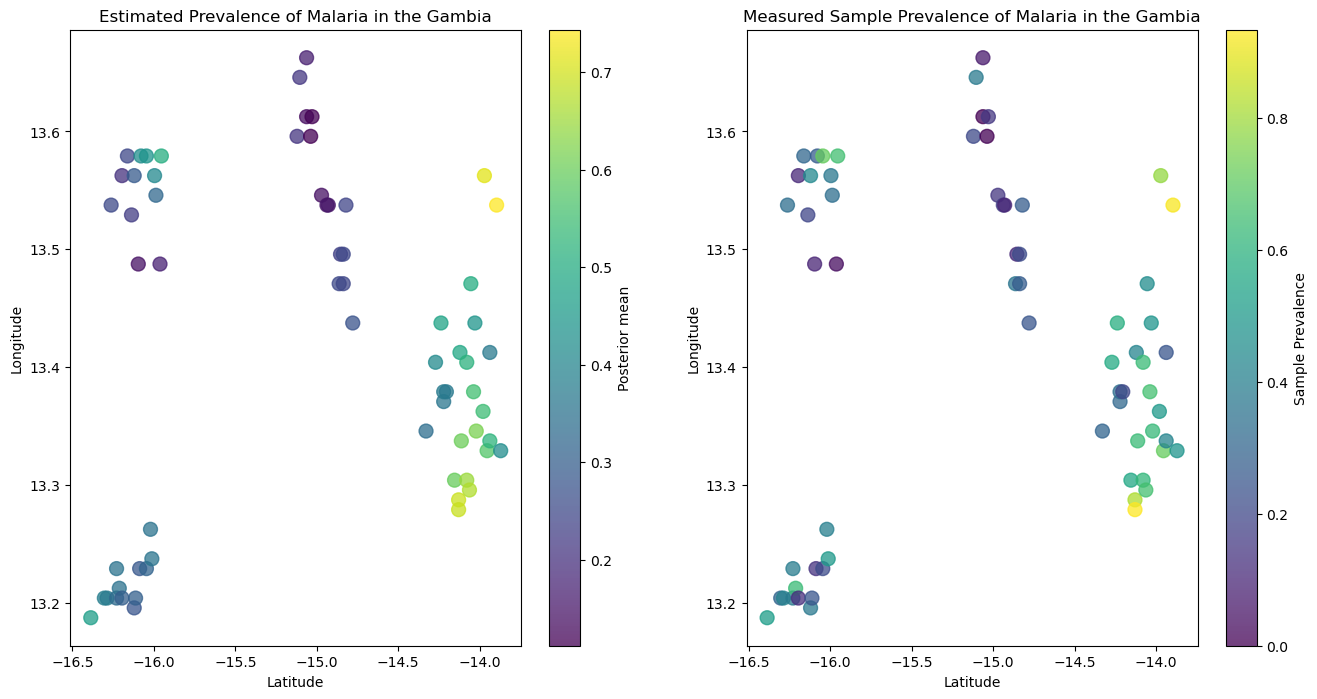
我们可以看到,左下图中的后验预测与右图所示的观测样本一致。
plt.subplots(nrows=1, ncols=2, figsize=(16, 8))
plt.subplot(1, 2, 1)
plt.scatter(
lonlat[:, 1],
lonlat[:, 0],
c=posterior_prevalence.mean(("chain", "draw")),
marker="o",
alpha=0.75,
s=100,
edgecolor=None,
)
plt.xlabel("Latitude")
plt.ylabel("Longitude")
plt.title("Estimated Prevalence of Malaria in the Gambia")
plt.colorbar(label="Posterior mean")
plt.subplot(1, 2, 2)
plt.scatter(
lonlat[:, 1],
lonlat[:, 0],
c=gambia_gpdf_4326["prev"],
marker="o",
alpha=0.75,
s=100,
edgecolor=None,
)
plt.xlabel("Latitude")
plt.ylabel("Longitude")
plt.title("Measured Sample Prevalence of Malaria in the Gambia")
plt.colorbar(label="Sample Prevalence");
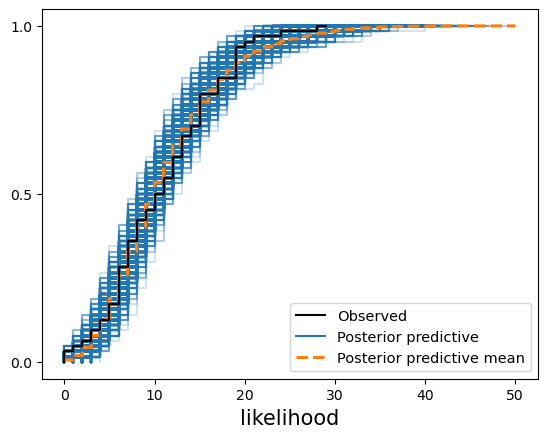
我们还可以检查似然性(疟疾检测呈阳性的人数)是否与观测数据一致。 正如下图中所示,我们的后验预测样本代表了观测样本。
az.plot_ppc(ppc, kind="cumulative");
样本外后验预测#
既然我们已经验证了我们有一个收敛的代表性模型,我们想要估计我们观察到数据点周围区域的疟疾流行率。 我们的新数据集将包括冈比亚境内每个经度和纬度位置,我们在这些位置有海拔高度的测量值。
with hsgp_model:
pm.set_data(new_data={"X": new_lonlat, "elevation": new_elev_std})
pp = pm.sample_posterior_predictive(hsgp_trace, var_names=["p"], random_seed=rng)
Sampling: []
posterior_predictive_prevalence = pp["posterior_predictive"]["p"]
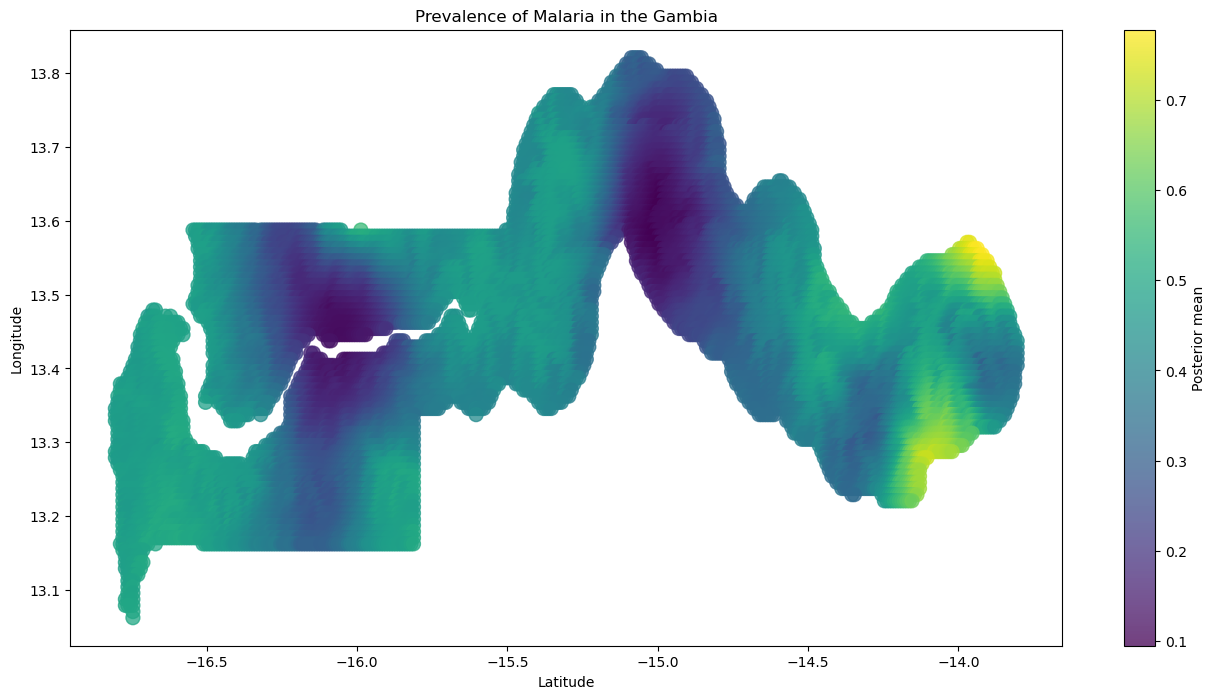
我们可以绘制我们的样本外后验预测,以可视化冈比亚各地估计的疟疾流行率。 在下图中,您会注意到,在我们观察到数据的区域周围,流行率平稳过渡,较近的区域具有更相似的流行率,并且随着您远离,您会接近零(高斯过程的均值)。
fig = plt.figure(figsize=(16, 8))
plt.scatter(
new_lonlat[:, 1],
new_lonlat[:, 0],
c=posterior_predictive_prevalence.mean(("chain", "draw")),
marker="o",
alpha=0.75,
s=100,
edgecolor=None,
)
plt.xlabel("Latitude")
plt.ylabel("Longitude")
plt.title("Prevalence of Malaria in the Gambia")
plt.colorbar(label="Posterior mean");
基于超限概率做出决策#
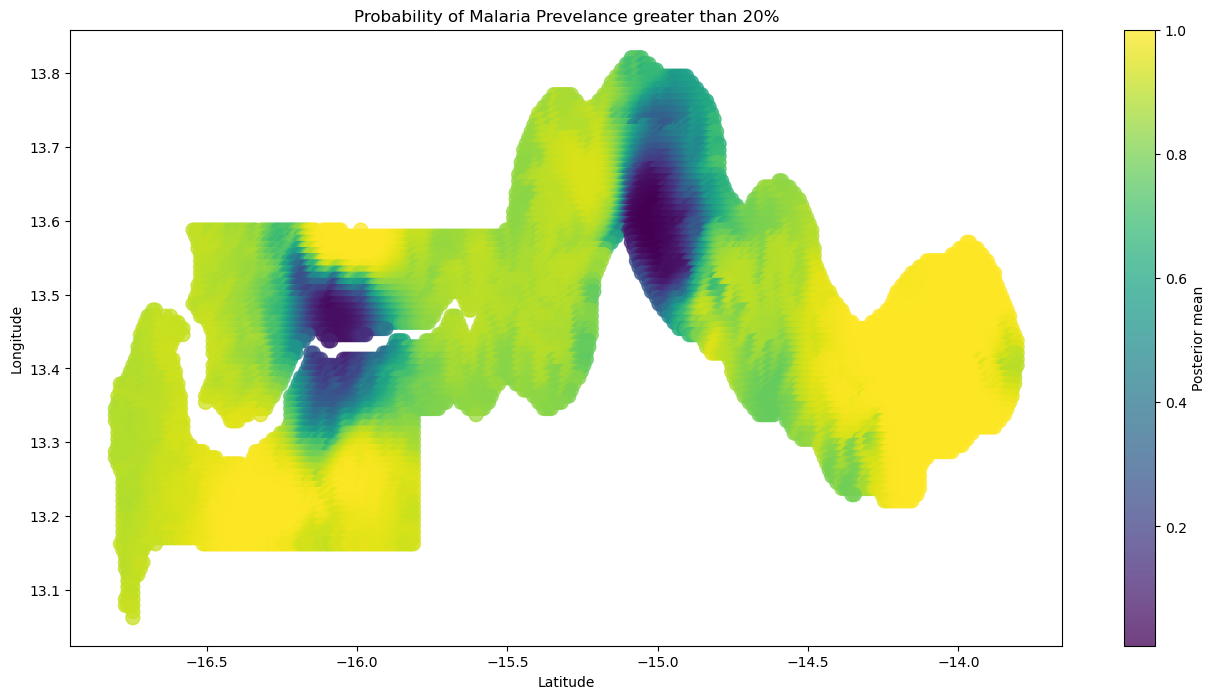
确定我们可能决定应用干预措施的一种方法是查看选定的疟疾流行率阈值的超限概率。 这些超限概率将使我们能够将我们对估计流行率的不确定性纳入考虑,而不是仅仅考虑后验分布的均值。 对于我们的用例,我们决定将流行率的超限阈值设置为 20%。
prob_prev_gt_20percent = 1 - (posterior_predictive_prevalence <= 0.2).mean(("chain", "draw"))
我们可以使用下图获得的见解,向我们最有信心的疟疾流行率超过 20% 的地区发送援助。
fig = plt.figure(figsize=(16, 8))
plt.scatter(
new_lonlat[:, 1],
new_lonlat[:, 0],
c=prob_prev_gt_20percent,
marker="o",
alpha=0.75,
s=100,
edgecolor=None,
)
plt.xlabel("Latitude")
plt.ylabel("Longitude")
plt.title("Probability of Malaria Prevalence greater than 20%")
plt.colorbar(label="Posterior mean");
不同的协方差函数#
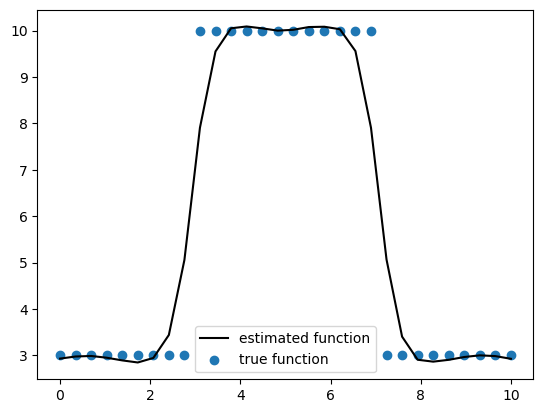
在结束之前,让我们简要讨论一下为什么我们决定使用 Matérn 协方差函数族而不是指数二次函数。 Matérn 协方差族是指数二次函数的推广。 当 Matérn 的平滑参数 \(\nu \to \infty\) 时,我们得到指数二次协方差函数。 随着平滑参数的增加,您正在估计的函数变得更平滑。 \(\nu\) 的几个常用值是 \(\frac{1}{2}\)、\(\frac{3}{2}\) 和 \(\frac{5}{2}\)。 通常,当估计具有空间依赖性的度量时,我们不希望函数过于平滑,因为这会阻止我们的估计捕捉到我们正在估计的测量值的突变。 下面我们模拟一些数据来展示 Matérn 如何能够捕捉这些突变,而指数二次函数则过于平滑。 为了简单起见,我们将在一维中工作,但这些概念适用于二维数据。
# simulate 1-dimensional data
x = np.linspace(0, 10, 30)
y = list()
for v in x:
# introduce abrupt changes
if v > 3 and v < 7:
y.append(np.array(10.0))
else:
y.append(np.array(3.0))
y = np.array(y).ravel()
# Fit a GP to model the simulated data
with pm.Model() as matern_model:
eta = pm.Exponential("eta", scale=10.0)
ls = pm.Lognormal("ls", mu=0.5, sigma=0.75)
cov_func = eta**2 * pm.gp.cov.Matern32(input_dim=1, ls=ls)
gp = pm.gp.Latent(cov_func=cov_func)
s = gp.prior("s", X=x[:, None])
measurement_error = pm.Exponential("measurement_error", scale=5.0)
pm.Normal("likelihood", mu=s, sigma=measurement_error, observed=y)
matern_idata = pm.sample(
draws=2000, tune=2000, nuts_sampler="numpyro", target_accept=0.98, random_seed=rng
)
There were 13 divergences after tuning. Increase `target_accept` or reparameterize.
with matern_model:
ppc = pm.sample_posterior_predictive(matern_idata, random_seed=rng)
Sampling: [likelihood]
y_mean_ppc = ppc["posterior_predictive"]["likelihood"].mean(("chain", "draw"))
# Plot the estimated function against the true function
plt.plot(x, y_mean_ppc, c="k", label="estimated function")
plt.scatter(x, y, label="true function")
plt.legend(loc="best")
<matplotlib.legend.Legend at 0x347d54710>
# Fit a GP to model the simulated data
with pm.Model() as expquad_model:
eta = pm.Exponential("eta", scale=10.0)
ls = pm.Lognormal("ls", mu=0.5, sigma=0.75)
cov_func = eta**2 * pm.gp.cov.ExpQuad(input_dim=1, ls=ls)
gp = pm.gp.Latent(cov_func=cov_func)
s = gp.prior("s", X=x[:, None])
measurement_error = pm.Exponential("measurement_error", scale=5.0)
pm.Normal("likelihood", mu=s, sigma=measurement_error, observed=y)
expquad_idata = pm.sample(
draws=2000, tune=2000, nuts_sampler="numpyro", target_accept=0.98, random_seed=rng
)
There were 5 divergences after tuning. Increase `target_accept` or reparameterize.
with expquad_model:
ppc = pm.sample_posterior_predictive(expquad_idata, random_seed=rng)
Sampling: [likelihood]
y_mean_ppc = ppc["posterior_predictive"]["likelihood"].mean(("chain", "draw"))
# Plot the estimated function against the true function
plt.plot(x, y_mean_ppc, c="k", label="estimated function")
plt.scatter(x, y, label="true function")
plt.legend(loc="best")
<matplotlib.legend.Legend at 0x399a4e3c0>

正如您从上图中看到的那样。 指数二次协方差函数太慢而无法捕捉突变,但由于过于平滑,也过冲了变化。
结论#
该案例研究向我们介绍了如何利用 HSGP 将空间信息纳入我们的估计中。 具体来说,我们看到了如何验证我们的模型规范、生成样本外估计,以及如何使用整个后验分布来做出决策。
参考文献#
水印#
%load_ext watermark
%watermark -n -u -v -iv -w -p xarray
Last updated: Tue Nov 19 2024
Python implementation: CPython
Python version : 3.12.2
IPython version : 8.29.0
xarray: 2024.10.0
matplotlib : 3.9.2
geopandas : 1.0.1
mapclassify: 2.8.1
rasterio : 1.4.2
arviz : 0.20.0
numpy : 1.26.4
pytensor : 2.26.3
pyproj : 3.7.0
pandas : 2.2.3
pymc : 5.18.1
folium : 0.18.0
Watermark: 2.5.0
许可声明#
此示例库中的所有 notebook 均根据 MIT 许可证提供,该许可证允许修改和再分发以用于任何用途,前提是保留版权和许可声明。
引用 PyMC 示例#
要引用此 notebook,请使用 Zenodo 为 pymc-examples 存储库提供的 DOI。
重要提示
许多 notebook 改编自其他来源:博客、书籍... 在这种情况下,您也应该引用原始来源。
另请记住引用您的代码使用的相关库。
这是一个 bibtex 中的引用模板
@incollection{citekey,
author = "<notebook authors, see above>",
title = "<notebook title>",
editor = "PyMC Team",
booktitle = "PyMC examples",
doi = "10.5281/zenodo.5654871"
}
渲染后可能看起来像