Weibull 加速失效时间模型重新参数化#
import arviz as az
import numpy as np
import pymc as pm
import pytensor.tensor as pt
print(f"Running on PyMC v{pm.__version__}")
Running on PyMC v5.18.2
注意
此 notebook 使用的库不是 PyMC 的依赖项,因此需要专门安装才能运行此 notebook。打开下面的下拉菜单以获取更多指导。
额外的依赖项安装说明
为了运行此 notebook(在本地或 Binder 上),您不仅需要安装可用的 PyMC 以及所有可选依赖项,还需要安装一些额外的依赖项。有关安装 PyMC 本身的建议,请参阅 安装
您可以使用首选的包管理器安装这些依赖项,我们以 pip 和 conda 命令为例提供如下。
$ pip install statsmodels
请注意,如果您想(或需要)从 notebook 内部而不是命令行安装软件包,您可以通过运行 pip 命令的变体来安装软件包
import sys
!{sys.executable} -m pip install statsmodels
您不应运行 !pip install,因为它可能会将软件包安装在不同的环境中,即使已安装,也无法从 Jupyter notebook 中使用。
另一种选择是使用 conda
$ conda install statsmodels
当使用 conda 安装科学 Python 包时,我们建议使用 conda-forge
# These dependencies need to be installed separately from PyMC
import statsmodels.api as sm
%config InlineBackend.figure_format = 'retina'
# These seeds are for sampling data observations
RANDOM_SEED = 8927
np.random.seed(RANDOM_SEED)
# Set a seed for reproducibility of posterior results
seed: int = sum(map(ord, "aft_weibull"))
rng: np.random.Generator = np.random.default_rng(seed=seed)
az.style.use("arviz-darkgrid")
数据集#
之前的关于贝叶斯参数生存分析的示例 notebook 介绍了两种不同的加速失效时间 (AFT) 模型:Weibull 和对数线性模型。在本 notebook 中,我们介绍了 Weibull AFT 模型的三个不同的参数化。
我们将使用的数据集是 flchain R 数据集,它来自一项医学研究,该研究调查了血清游离轻链 (FLC) 对寿命的影响。通过运行以下代码阅读数据的完整文档
print(sm.datasets.get_rdataset(package='survival', dataname='flchain').__doc__).
# Fetch and clean data
data = (
sm.datasets.get_rdataset(package="survival", dataname="flchain")
.data.sample(500) # Limit ourselves to 500 observations
.reset_index(drop=True)
)
y = data.futime.values
censored = ~data["death"].values.astype(bool)
y[:5]
array([ 975, 2272, 138, 4262, 4928])
censored[:5]
array([False, True, False, True, True])
使用 pm.Potential#
在建模删失数据时,我们遇到了一个独特的问题。严格来说,对于删失值,我们没有任何数据:我们只知道删失值的数量。我们如何将此信息包含在我们的模型中?
一种方法是使用 pm.Potential。PyMC2 文档 非常好地解释了它的用法。本质上,声明 pm.Potential('x', logp) 将把 logp 添加到模型的对数似然中。
但是,pm.Potential 仅影响基于概率的抽样,这排除了使用 pm.sample_prior_predictice 和 pm.sample_posterior_predictive。我们可以通过使用 pm.Censored 来克服这些限制。我们可以通过定义 pm.Censored 的 upper 参数来建模我们的右删失数据。
参数化 1#
此参数化是 Weibull 生存函数的直观、直接的参数化。这可能是人们首先想到的参数化。
# normalize the event time between 0 and 1
y_norm = y / np.max(y)
# If censored then observed event time else maximum time
right_censored = [x if x > 0 else np.max(y_norm) for x in y_norm * censored]
with pm.Model() as model_1:
alpha_sd = 1.0
mu = pm.Normal("mu", mu=0, sigma=1)
alpha_raw = pm.Normal("a0", mu=0, sigma=0.1)
alpha = pm.Deterministic("alpha", pt.exp(alpha_sd * alpha_raw))
beta = pm.Deterministic("beta", pt.exp(mu / alpha))
beta_backtransformed = pm.Deterministic("beta_backtransformed", beta * np.max(y))
latent = pm.Weibull.dist(alpha=alpha, beta=beta)
y_obs = pm.Censored("Censored_likelihood", latent, upper=right_censored, observed=y_norm)
az.plot_trace(idata_param1, var_names=["alpha", "beta"])
array([[<Axes: title={'center': 'alpha'}>,
<Axes: title={'center': 'alpha'}>],
[<Axes: title={'center': 'beta'}>,
<Axes: title={'center': 'beta'}>]], dtype=object)
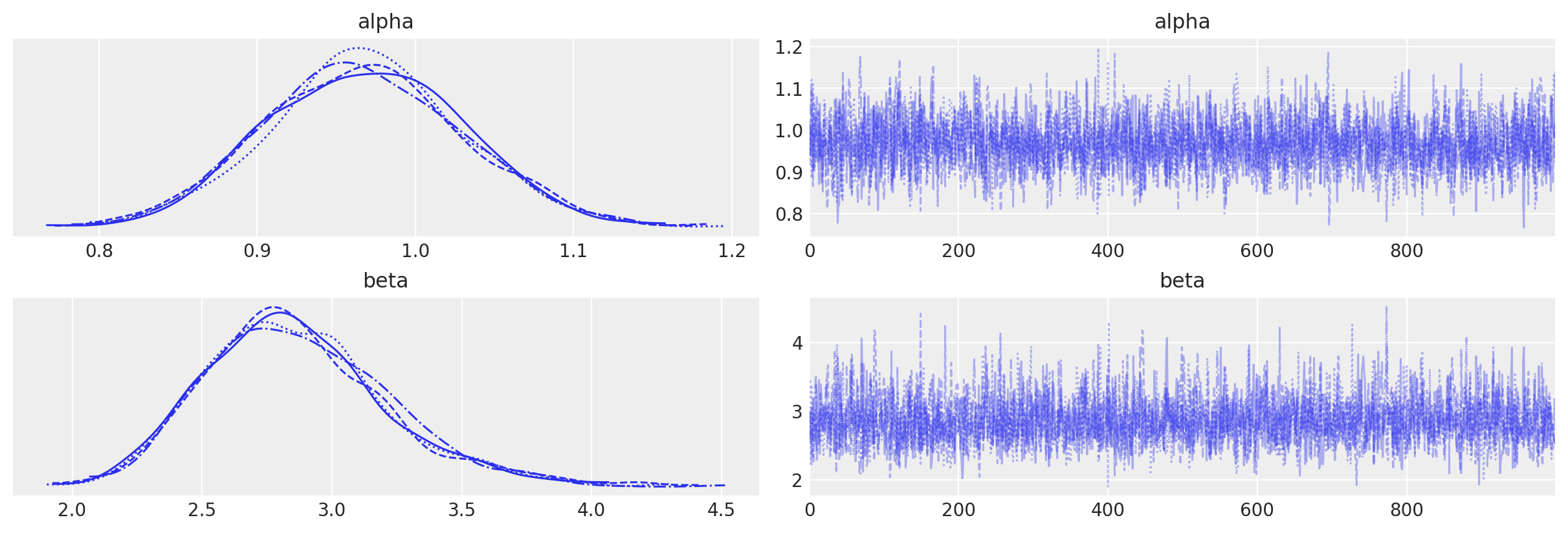
az.summary(idata_param1, var_names=["alpha", "beta", "beta_backtransformed"], round_to=2)
| 平均值 | 标准差 | hdi_3% | hdi_97% | mcse_mean | mcse_sd | ess_bulk | ess_tail | r_hat | |
|---|---|---|---|---|---|---|---|---|---|
| alpha | 0.97 | 0.06 | 0.85 | 1.08 | 0.00 | 0.0 | 2752.10 | 2282.64 | 1.0 |
| beta | 2.86 | 0.35 | 2.23 | 3.54 | 0.01 | 0.0 | 2677.53 | 2519.18 | 1.0 |
| beta_backtransformed | 14694.41 | 1806.79 | 11450.50 | 18165.12 | 34.64 | 24.5 | 2677.53 | 2519.18 | 1.0 |
参数化 2#
请注意,令人困惑的是,alpha 现在称为 r,而 alpha 表示先验;我们保留此符号是为了忠实于 Stan 中的原始实现。在此参数化中,我们仍然对相同的参数 alpha(现在是 r)和 beta 进行建模。
有关更多信息,请参阅 此 Stan 示例模型 和 相应的文档。
with pm.Model() as model_2:
alpha = pm.Normal("alpha", mu=0, sigma=1)
r = pm.Gamma("r", alpha=2, beta=1)
beta = pm.Deterministic("beta", pt.exp(-alpha / r))
beta_backtransformed = pm.Deterministic("beta_backtransformed", beta * np.max(y))
latent = pm.Weibull.dist(alpha=r, beta=beta)
y_obs = pm.Censored("Censored_likelihood", latent, upper=right_censored, observed=y_norm)
az.plot_trace(idata_param2, var_names=["r", "beta"])
array([[<Axes: title={'center': 'r'}>, <Axes: title={'center': 'r'}>],
[<Axes: title={'center': 'beta'}>,
<Axes: title={'center': 'beta'}>]], dtype=object)
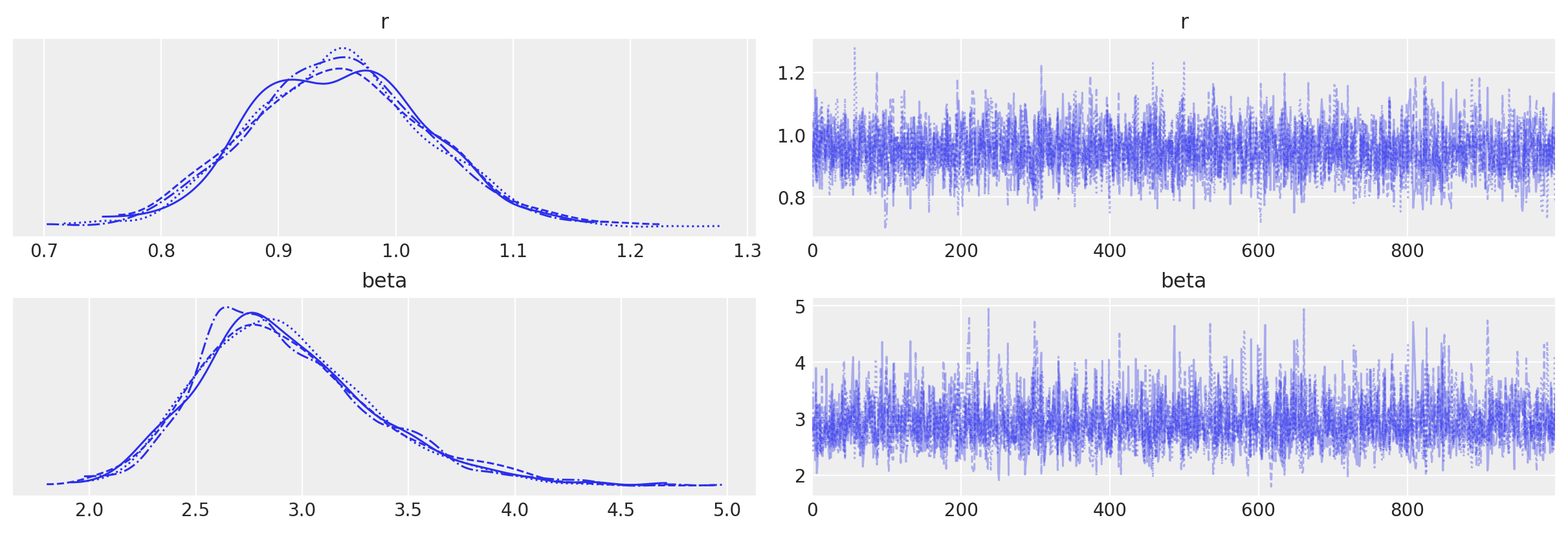
az.summary(idata_param2, var_names=["r", "beta", "beta_backtransformed"], round_to=2)
| 平均值 | 标准差 | hdi_3% | hdi_97% | mcse_mean | mcse_sd | ess_bulk | ess_tail | r_hat | |
|---|---|---|---|---|---|---|---|---|---|
| r | 0.95 | 0.08 | 0.80 | 1.09 | 0.00 | 0.00 | 2907.17 | 2504.73 | 1.0 |
| beta | 2.93 | 0.43 | 2.23 | 3.81 | 0.01 | 0.01 | 2661.47 | 2456.59 | 1.0 |
| beta_backtransformed | 15060.96 | 2201.68 | 11442.10 | 19549.83 | 42.35 | 29.96 | 2661.47 | 2456.59 | 1.0 |
参数化 3#
在此参数化中,我们使用 Gumbel 分布对数线性误差分布进行建模,而不是直接对生存函数进行建模。有关更多信息,请参阅 此博客文章。
logtime = np.log(y)
# If censored then observed event time else maximum time
right_censored = [x if x > 0 else np.max(logtime) for x in logtime * censored]
with pm.Model() as model_3:
s = pm.HalfNormal("s", tau=3.0)
gamma = pm.Normal("gamma", mu=0, sigma=5)
latent = pm.Gumbel.dist(mu=gamma, beta=s)
y_obs = pm.Censored("Censored_likelihood", latent, upper=right_censored, observed=logtime)
az.plot_trace(idata_param3)
array([[<Axes: title={'center': 'gamma'}>,
<Axes: title={'center': 'gamma'}>],
[<Axes: title={'center': 's'}>, <Axes: title={'center': 's'}>]],
dtype=object)
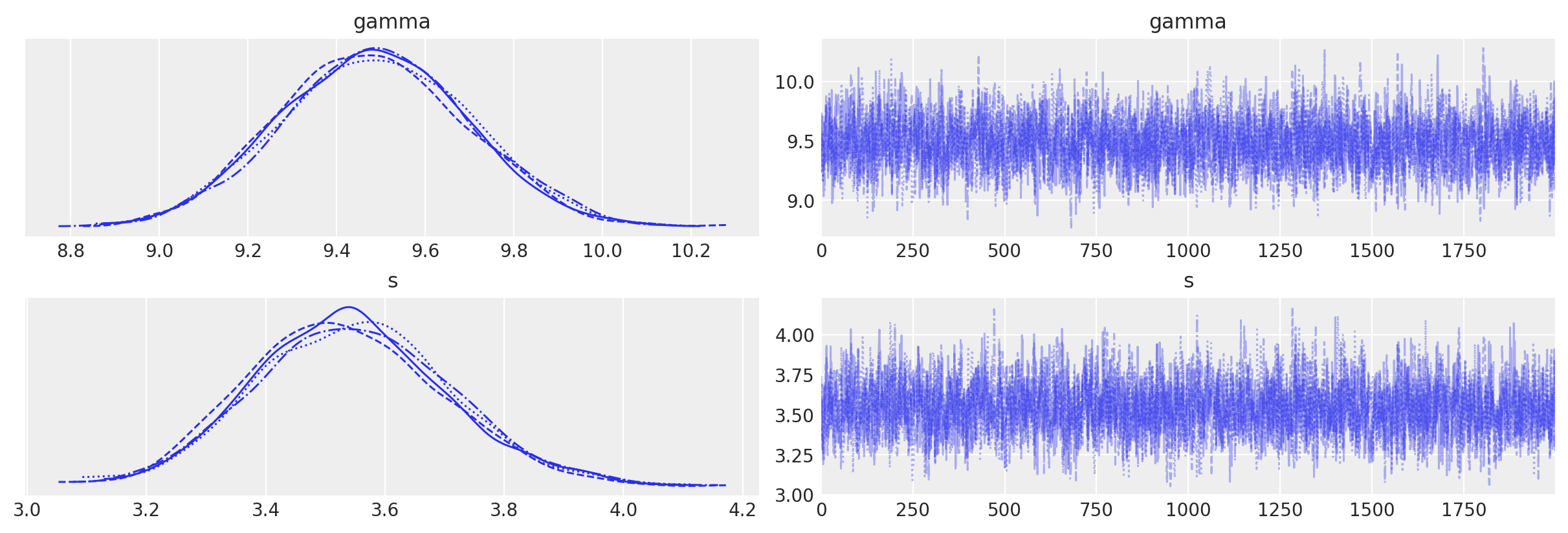
az.summary(idata_param3, round_to=2)
| 平均值 | 标准差 | hdi_3% | hdi_97% | mcse_mean | mcse_sd | ess_bulk | ess_tail | r_hat | |
|---|---|---|---|---|---|---|---|---|---|
| gamma | 9.49 | 0.21 | 9.10 | 9.89 | 0.0 | 0.0 | 2727.89 | 3337.16 | 1.0 |
| s | 3.54 | 0.16 | 3.24 | 3.85 | 0.0 | 0.0 | 2701.50 | 4170.84 | 1.0 |
许可声明#
此示例库中的所有 notebook 均根据 MIT 许可证 提供,该许可证允许修改和再分发用于任何用途,前提是版权和许可声明得到保留。
引用 PyMC 示例#
要引用此 notebook,请使用 Zenodo 为 pymc-examples 存储库提供的 DOI。
重要提示
许多 notebook 都改编自其他来源:博客、书籍... 在这种情况下,您也应该引用原始来源。
另请记住引用您的代码使用的相关库。
这是一个 bibtex 中的引用模板
@incollection{citekey,
author = "<notebook authors, see above>",
title = "<notebook title>",
editor = "PyMC Team",
booktitle = "PyMC examples",
doi = "10.5281/zenodo.5654871"
}
渲染后可能如下所示