从生成图导出的时间序列模型#
在本笔记本中,我们将展示如何从生成图开始建模和拟合时间序列模型。 特别是,我们将解释如何在 PyMC 模型中高效地使用 scan 进行循环。
动机
为什么我们要这样做,而不是直接使用 AR 呢? 有什么好处?
PyMC 中预构建的时间序列模型非常有用且易于使用。 然而,它们不够灵活,无法对更复杂的时间序列模型进行建模。 通过使用生成图,我们可以对我们想要的任何时间序列模型进行建模,只要我们可以用生成图来定义它。 例如
具有不同噪声分布的自回归模型(例如,
StudentT噪声)。指数平滑模型。
ARIMA-GARCH 模型。
在此示例中,我们考虑自回归模型 AR(2)。 回顾一下,AR(2) 模型定义为
也就是说,我们有一个关于时间序列前两个滞后项的递归线性模型。
显示代码单元格源
import arviz as az
import matplotlib.pyplot as plt
import numpy as np
import pymc as pm
import pytensor
import pytensor.tensor as pt
import statsmodels.api as sm
from pymc.pytensorf import collect_default_updates
az.style.use("arviz-darkgrid")
plt.rcParams["figure.figsize"] = [12, 7]
plt.rcParams["figure.dpi"] = 100
plt.rcParams["figure.facecolor"] = "white"
%config InlineBackend.figure_format = "retina"
rng = np.random.default_rng(42)
WARNING (pytensor.tensor.blas): Using NumPy C-API based implementation for BLAS functions.
定义 AR(2) 过程#
我们首先将 AR(2) 模型的生成图编码为一个函数 ar_dist。 策略是将此函数作为自定义分布通过 PyMC 模型内部的 CustomDist 传递。
我们需要指定初始状态 (ar_init)、自回归系数 (rho) 和噪声的标准差 (sigma)。 给定这些参数,我们可以使用 scan 操作定义 AR(2) 模型的生成图。
所有这些函数是什么?
起初,看到所有这些函数可能会让人感到有些不知所措。 然而,它们只是定义 AR(2) 模型生成图的辅助函数。
collect_default_updates()告诉 PyMC,生成图中的随机变量 (RV) 应在循环的每次迭代中更新。 如果我们不这样做,随机状态将不会在时间步之间更新,我们将一次又一次地对相同的创新进行采样。scan是在 PyMC 模型内部循环的有效方法。 它类似于 Python 中的for循环,但针对pytensor进行了优化。 我们需要指定以下参数fn: 定义过渡步长的函数。outputs_info: 这是变量或字典的列表,描述了循环计算的输出的初始状态。 此字典的一个常见键是taps,它指定要跟踪的先前时间步的数量。 在这种情况下,我们跟踪最后两个时间步(滞后)。non_sequences: 传递给每个步骤的fn的参数列表。 在这种情况下,是 AR(2) 模型的自回归系数和噪声标准差。n_steps: 循环的步数。strict: 如果为True,则fn中使用的所有共享变量都必须作为non_sequences或sequences的一部分提供(在本例中,我们不使用参数sequences,它是描述scan必须迭代的序列的变量或字典列表。 在这种情况下,我们可以简单地循环遍历时间步)。
让我们看看具体的实现
lags = 2 # Number of lags
timeseries_length = 100 # Time series length
# This is the transition function for the AR(2) model.
# We take as inputs previous steps and then specify the autoregressive relationship.
# Finally, we add Gaussian noise to the model.
def ar_step(x_tm2, x_tm1, rho, sigma):
mu = x_tm1 * rho[0] + x_tm2 * rho[1]
x = mu + pm.Normal.dist(sigma=sigma)
return x, collect_default_updates([x])
# Here we actually "loop" over the time series.
def ar_dist(ar_init, rho, sigma, size):
ar_steps, _ = pytensor.scan(
fn=ar_step,
outputs_info=[{"initial": ar_init, "taps": range(-lags, 0)}],
non_sequences=[rho, sigma],
n_steps=timeseries_length - lags,
strict=True,
)
return ar_steps
生成 AR(2) 图#
现在我们已经实现了 AR(2) 步骤,我们可以为参数 rho、sigma 和初始状态 ar_init 分配先验。
coords = {
"lags": range(-lags, 0),
"steps": range(timeseries_length - lags),
"timeseries_length": range(timeseries_length),
}
with pm.Model(coords=coords, check_bounds=False) as model:
rho = pm.Normal(name="rho", mu=0, sigma=0.2, dims=("lags",))
sigma = pm.HalfNormal(name="sigma", sigma=0.2)
ar_init = pm.Normal(name="ar_init", sigma=0.5, dims=("lags",))
ar_steps = pm.CustomDist(
"ar_steps",
ar_init,
rho,
sigma,
dist=ar_dist,
dims=("steps",),
)
ar = pm.Deterministic(
name="ar", var=pt.concatenate([ar_init, ar_steps], axis=-1), dims=("timeseries_length",)
)
pm.model_to_graphviz(model)
先验#
让我们从先验分布中采样,看看 AR(2) 模型的行为。
with model:
prior = pm.sample_prior_predictive(samples=500, random_seed=rng)
Sampling: [ar_init, ar_steps, rho, sigma]
_, ax = plt.subplots()
for i, hdi_prob in enumerate((0.94, 0.64), 1):
hdi = az.hdi(prior.prior["ar"], hdi_prob=hdi_prob)["ar"]
lower = hdi.sel(hdi="lower")
upper = hdi.sel(hdi="higher")
ax.fill_between(
x=np.arange(timeseries_length),
y1=lower,
y2=upper,
alpha=(i - 0.2) * 0.2,
color="C0",
label=f"{hdi_prob:.0%} HDI",
)
ax.plot(prior.prior["ar"].mean(("chain", "draw")), color="C0", label="Mean")
ax.legend(loc="upper right")
ax.set_xlabel("time")
ax.set_title("AR(2) Prior Samples", fontsize=18, fontweight="bold");
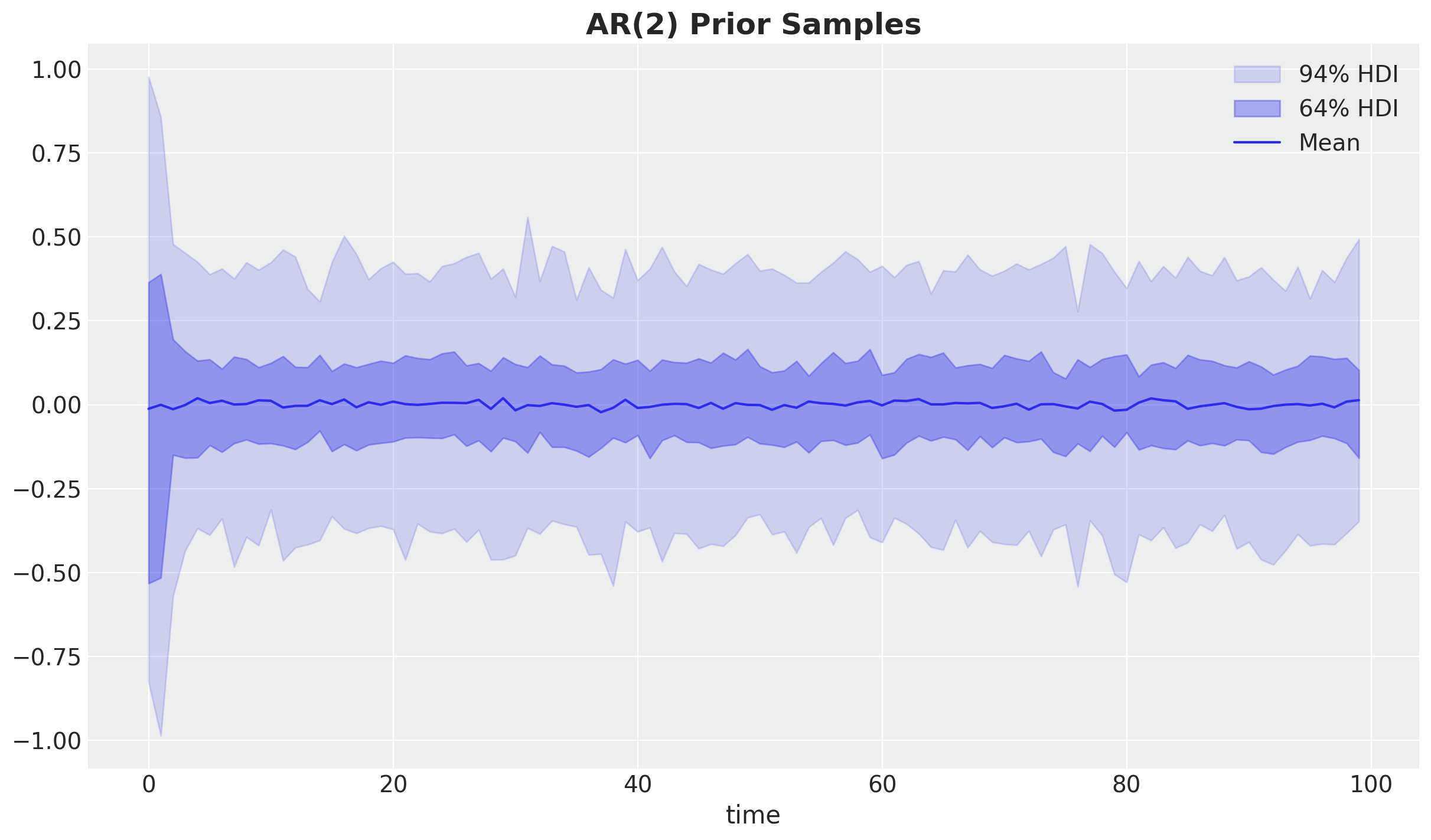
鉴于我们对 rho 参数的先验是弱信息性的且以零为中心,因此先验分布是零附近的平稳过程并不令人惊讶。
让我们看看单个样本,以感受生成序列的异质性
fig, ax = plt.subplots(
nrows=5, ncols=1, figsize=(12, 12), sharex=True, sharey=True, layout="constrained"
)
chosen_draw = 2
for i, axi in enumerate(ax, start=chosen_draw):
axi.plot(
prior.prior["ar"].isel(draw=i, chain=0),
color="C0" if i == chosen_draw else "black",
)
axi.set_title(f"Sample {i}", fontsize=18, fontweight="bold")
ax[-1].set_xlabel("time");
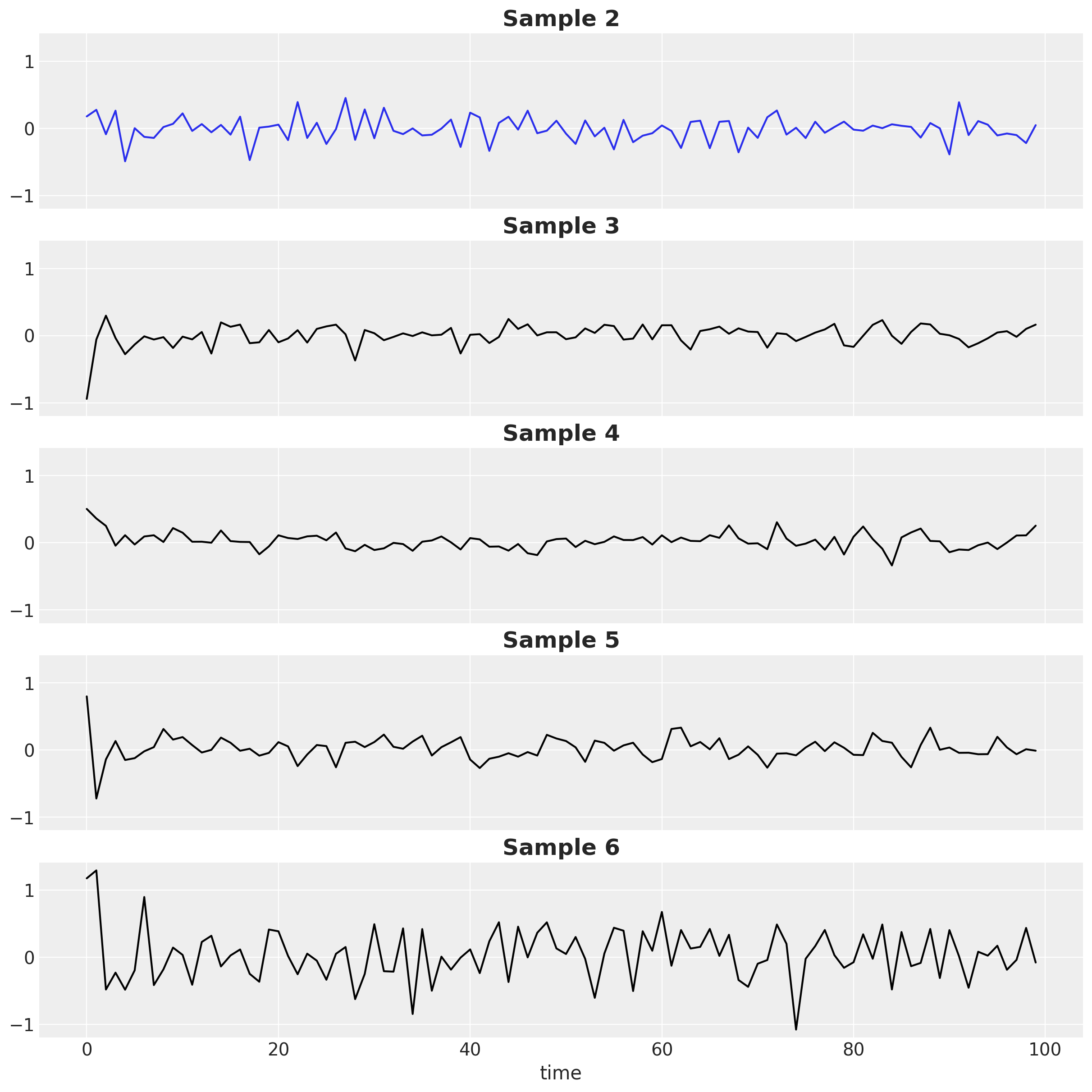
后验#
接下来,我们想将 AR(2) 模型以一些观测数据为条件,以便我们可以进行参数恢复分析。 我们使用 observe() 操作。
# select a random draw from the prior
prior_draw = prior.prior.isel(chain=0, draw=chosen_draw)
test_data = prior_draw["ar_steps"].to_numpy()
with pm.observe(model, {"ar_steps": test_data}) as observed_model:
trace = pm.sample(chains=4, random_seed=rng)
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [rho, sigma, ar_init]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 43 seconds.
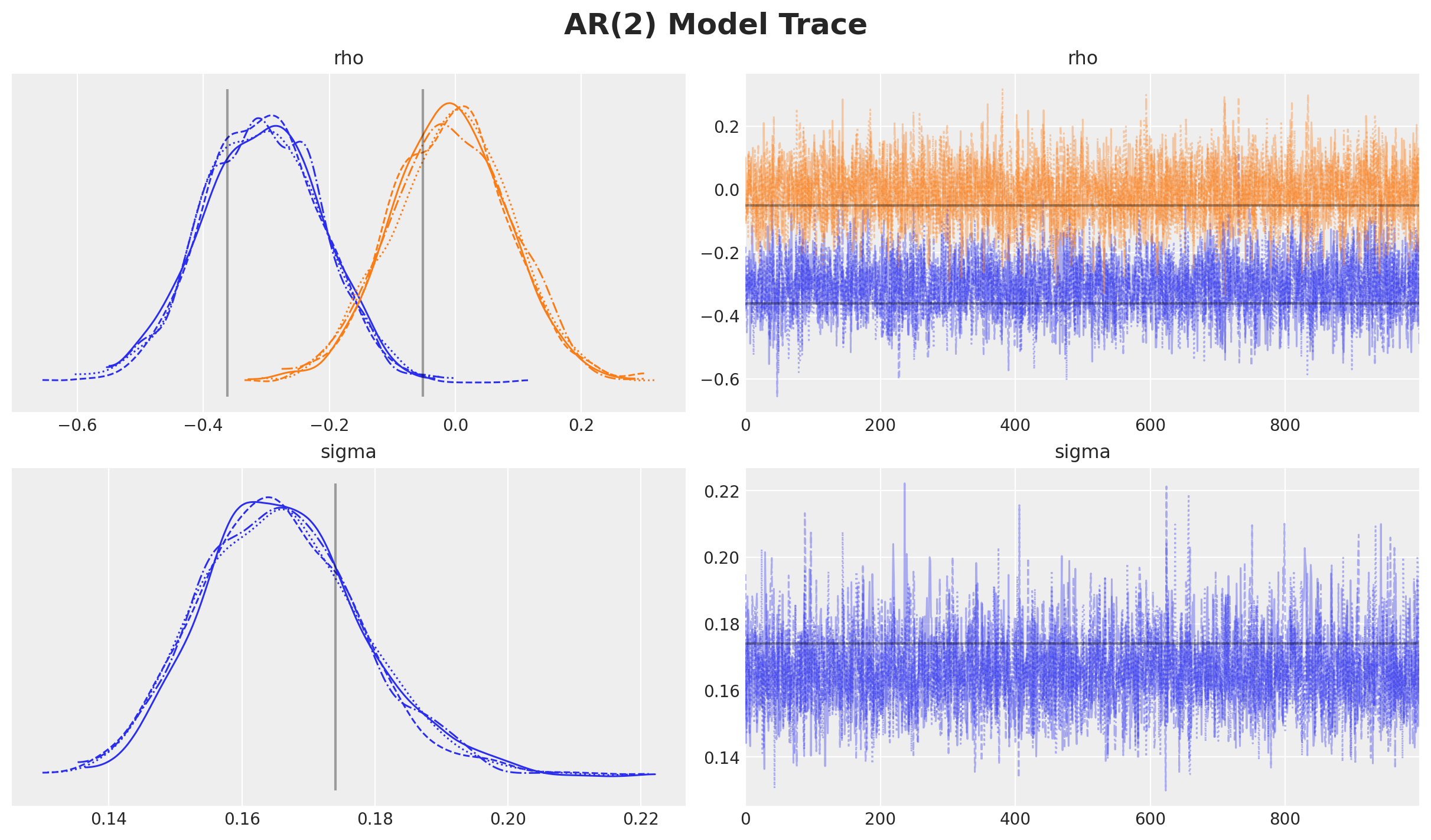
让我们绘制参数的迹线和后验分布。
# Get the true values of the parameters from the prior draw
rho_true = prior_draw["rho"].to_numpy()
sigma_true = prior_draw["sigma"].to_numpy()
ar_obs = prior_draw["ar"].to_numpy()
axes = az.plot_trace(
data=trace,
var_names=["rho", "sigma"],
compact=True,
lines=[
("rho", {}, rho_true),
("sigma", {}, sigma_true),
],
backend_kwargs={"figsize": (12, 7), "layout": "constrained"},
)
plt.gcf().suptitle("AR(2) Model Trace", fontsize=18, fontweight="bold");
axes = az.plot_posterior(
trace, var_names=["rho", "sigma"], ref_val=[*rho_true, sigma_true], figsize=(15, 5)
)
plt.gcf().suptitle("AR(2) Model Parameters Posterior", fontsize=18, fontweight="bold");
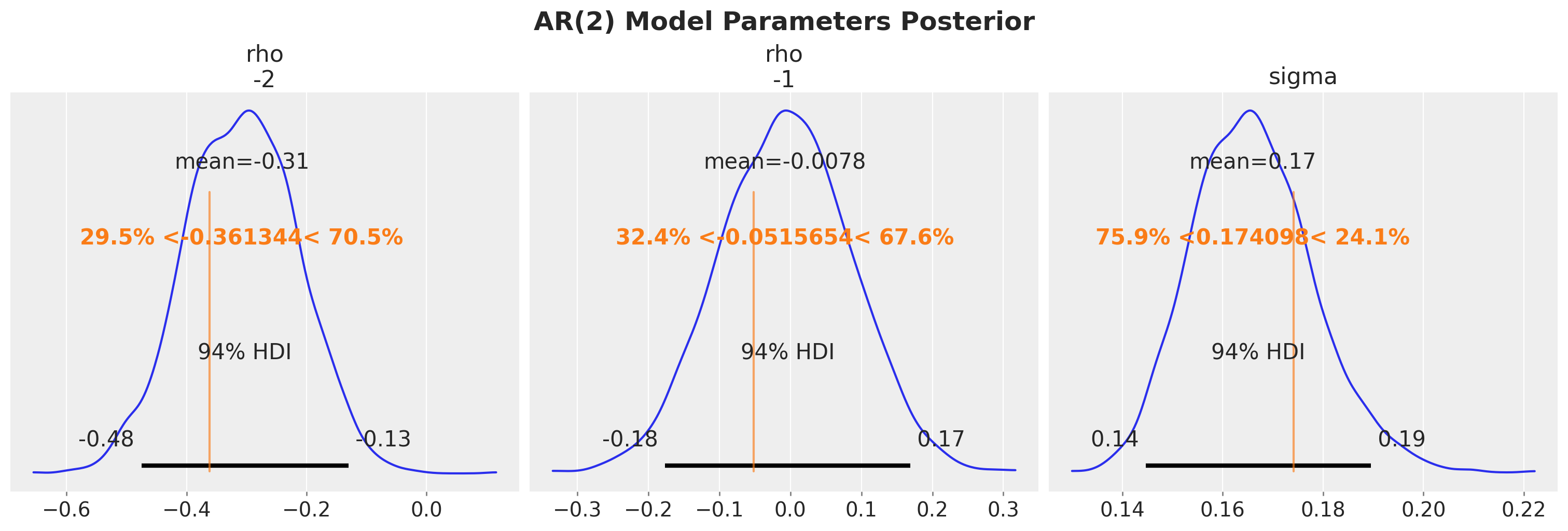
我们看到我们已成功恢复模型的真实参数。
后验预测#
最后,我们可以使用后验样本从 AR(2) 模型生成新数据。 然后,我们可以将生成的数据与观测数据进行比较,以检查模型的拟合优度。
with observed_model:
post_pred = pm.sample_posterior_predictive(trace, random_seed=rng)
Sampling: [ar_steps]
post_pred_ar = post_pred.posterior_predictive["ar"]
_, ax = plt.subplots()
for i, hdi_prob in enumerate((0.94, 0.64), 1):
hdi = az.hdi(post_pred_ar, hdi_prob=hdi_prob)["ar"]
lower = hdi.sel(hdi="lower")
upper = hdi.sel(hdi="higher")
ax.fill_between(
x=post_pred_ar["timeseries_length"],
y1=lower,
y2=upper,
alpha=(i - 0.2) * 0.2,
color="C0",
label=f"{hdi_prob:.0%} HDI",
)
ax.plot(
post_pred_ar["timeseries_length"],
post_pred_ar.mean(("chain", "draw")),
color="C0",
label="Mean",
)
ax.plot(ar_obs, color="black", label="Observed")
ax.legend(loc="upper right")
ax.set_xlabel("time")
ax.set_title("AR(2) Posterior Predictive Samples", fontsize=18, fontweight="bold");
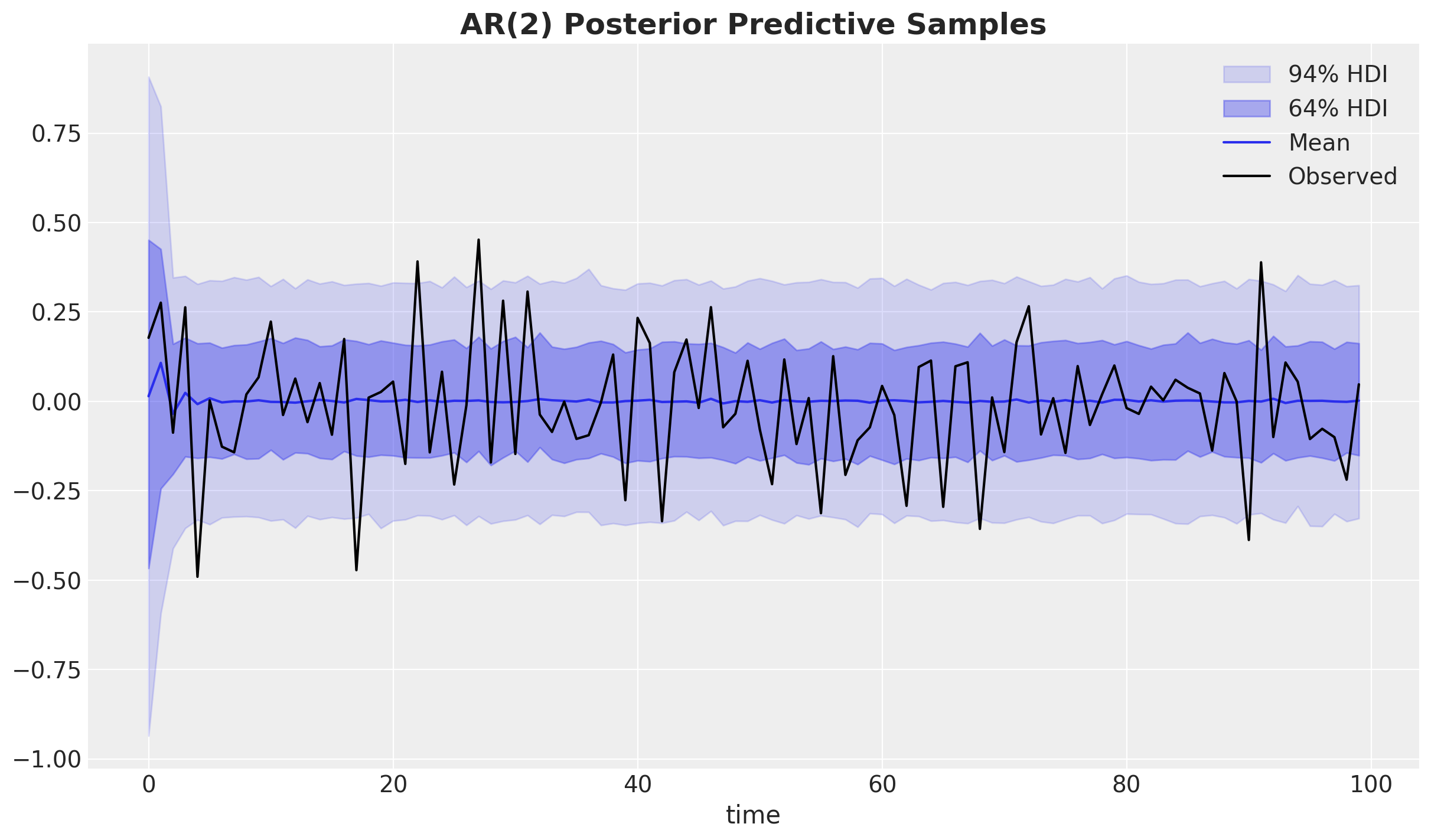
总的来说,我们看到该模型正在捕捉时间序列的全局动态。 为了更好地了解模型,我们可以绘制后验样本的子集,并将它们与观测数据进行比较。
fig, ax = plt.subplots(
nrows=5, ncols=1, figsize=(12, 12), sharex=True, sharey=True, layout="constrained"
)
for i, axi in enumerate(ax):
axi.plot(post_pred.posterior_predictive["ar"].isel(draw=i, chain=0), color="C0")
axi.plot(ar_obs, color="black", label="Observed")
axi.legend(loc="upper right")
axi.set_title(f"Sample {i}")
ax[-1].set_xlabel("time")
fig.suptitle("AR(2) Posterior Predictive Samples", fontsize=18, fontweight="bold", y=1.05);
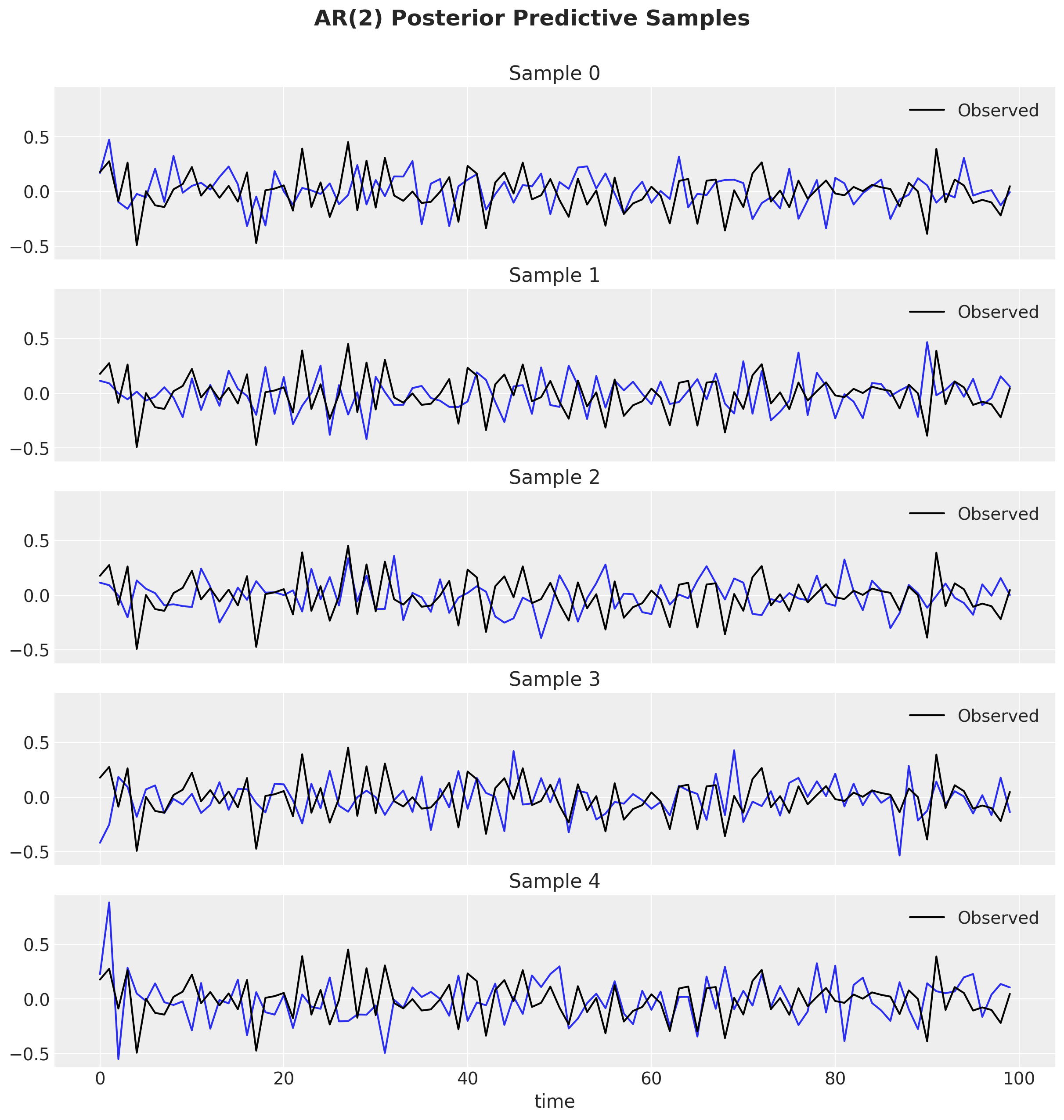
条件后验和非条件后验
许多用户会对这个后验感到惊讶,因为他们习惯于看到条件单步预测,即
(你在 statsmodels/stata/everything 中得到的),它更紧密,更贴近数据。
让我们看看如何在 PyMC 中做到这一点! 关键的观察是,我们需要将观测数据显式地传递到生成图中的“for 循环”中。 也就是说,我们需要将其传递到 scan 函数中。
def conditional_ar_dist(y_data, rho, sigma, size):
# Here we condition on the observed data by passing it through the `sequences` argument.
ar_steps, _ = pytensor.scan(
fn=ar_step,
sequences=[{"input": y_data, "taps": list(range(-lags, 0))}],
non_sequences=[rho, sigma],
n_steps=timeseries_length - lags,
strict=True,
)
return ar_steps
然后我们可以简单地从后验预测分布中生成样本。 请注意,我们需要“重写”生成图以包含条件过渡步骤。 当你调用 sample_posterior_predictive() 时,PyMC 将尝试将活动模型上下文中的随机变量名称与提供的 idata.posterior 中的名称进行匹配。 如果找到匹配项,则指定的模型先验将被忽略,并替换为从后验中抽取的样本。 这意味着我们可以对这些参数设置任何我们想要的先验,因为它将被忽略。 我们选择 Flat 是因为你无法从中采样。 这样,如果 PyMC 没有找到我们先验的匹配项,我们将收到一个错误,告知我们某些地方不对劲。 有关这些类型的跨模型预测的详细说明,请参阅精彩的博客文章 Out of model predictions with PyMC。
警告
我们需要将坐标 steps 向前移动一位! 原因是(例如)step=1 处的数据用于创建 step=2 的预测。 如果不进行移动,则 step=0 预测将被错误地标记为 step=0,并且模型看起来会比实际更好。
coords = {
"lags": range(-lags, 0),
"steps": range(-1, timeseries_length - lags - 1), # <- Coordinate shift!
"timeseries_length": range(1, timeseries_length + 1), # <- Coordinate shift!
}
with pm.Model(coords=coords, check_bounds=False) as conditional_model:
y_data = pm.Data("y_data", ar_obs)
rho = pm.Flat(name="rho", dims=("lags",))
sigma = pm.Flat(name="sigma")
ar_steps = pm.CustomDist(
"ar_steps",
y_data,
rho,
sigma,
dist=conditional_ar_dist,
dims=("steps",),
)
ar = pm.Deterministic(
name="ar", var=pt.concatenate([ar_init, ar_steps], axis=-1), dims=("timeseries_length",)
)
post_pred_conditional = pm.sample_posterior_predictive(trace, var_names=["ar"], random_seed=rng)
Sampling: [ar_steps]
让我们可视化条件后验预测分布,并将其与 statsmodels 结果进行比较
# PyMC conditional posterior predictive samples
conditional_post_pred_ar = post_pred_conditional.posterior_predictive["ar"]
# Statsmodels AR(2) model
mod = sm.tsa.ARIMA(ar_obs, order=(2, 0, 0))
res = mod.fit()
_, ax = plt.subplots()
for i, hdi_prob in enumerate((0.94, 0.64), 1):
hdi = az.hdi(conditional_post_pred_ar, hdi_prob=hdi_prob)["ar"]
lower = hdi.sel(hdi="lower")
upper = hdi.sel(hdi="higher")
ax.fill_between(
x=conditional_post_pred_ar["timeseries_length"],
y1=lower,
y2=upper,
alpha=(i - 0.2) * 0.2,
color="C1",
label=f"{hdi_prob:.0%} HDI",
)
ax.plot(
conditional_post_pred_ar["timeseries_length"],
conditional_post_pred_ar.mean(("chain", "draw")),
color="C1",
label="Mean",
)
ax.plot(ar_obs, color="black", label="Observed")
ax.plot(
conditional_post_pred_ar["timeseries_length"],
res.fittedvalues,
color="C2",
label="statsmodels",
)
ax.legend(loc="lower center", bbox_to_anchor=(0.5, -0.2), ncol=5)
ax.set_xlabel("time")
ax.set_title("AR(2) Conditional Posterior Predictive Samples", fontsize=18, fontweight="bold");
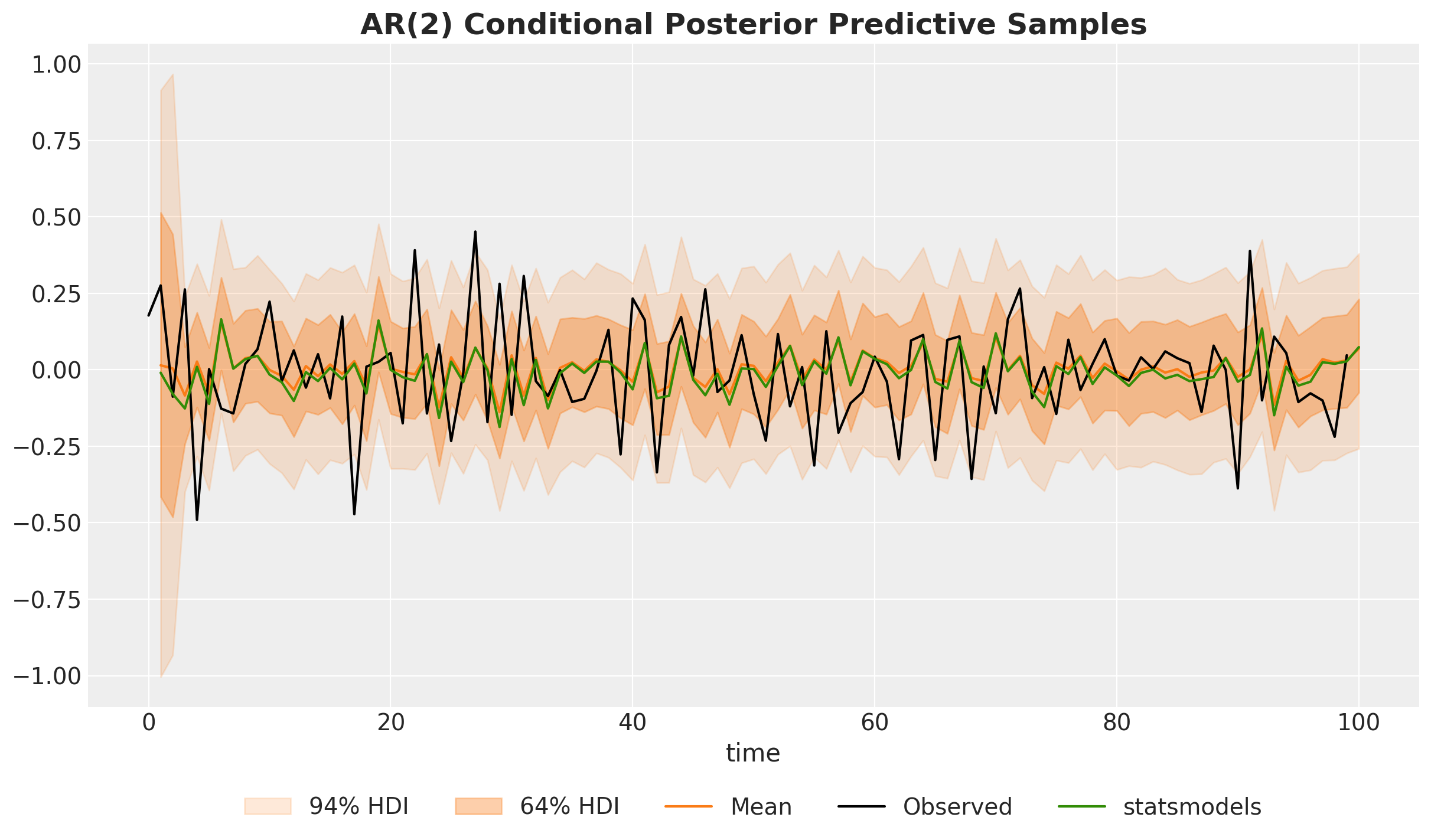
我们确实看到这些可信区间比非条件区间更窄。
以下是一些补充说明
没有对 \(y_0\) 的预测,因为我们没有观察到 \(y_{t - 1}\)。
预测似乎在“追逐”数据,因为这正是我们正在做的事情。 在每个步骤中,我们都重置为观测数据并进行一次预测。
注意
相对于 statsmodels 参考,我们在初始化方面只是略有不同。 这是有道理的,因为他们做了一些花哨的 MLE 初始化技巧,我们将它估计为一个参数。 当我们迭代序列时,差异应该会消失,我们看到确实如此。
样本外预测#
在最后一节中,我们将描述如何生成样本外预测。
# Specify the number of steps to forecast
forecast_steps = 10
我们的想法是使用后验样本和最新的两个可用数据点(因为我们有一个 AR(2) 模型)来生成预测
coords = {
"lags": range(-lags, 0),
"steps": range(timeseries_length, timeseries_length + forecast_steps),
}
with pm.Model(coords=coords, check_bounds=False) as forecasting_model:
forecast_initial_state = pm.Data("forecast_initial_state", ar_obs[-lags:], dims=("lags",))
rho = pm.Flat(name="rho", dims=("lags",))
sigma = pm.Flat(name="sigma")
def ar_dist_forecasting(forecast_initial_state, rho, sigma, size):
ar_steps, _ = pytensor.scan(
fn=ar_step,
outputs_info=[{"initial": forecast_initial_state, "taps": range(-lags, 0)}],
non_sequences=[rho, sigma],
n_steps=forecast_steps,
strict=True,
)
return ar_steps
ar_steps = pm.CustomDist(
"ar_steps",
forecast_initial_state,
rho,
sigma,
dist=ar_dist_forecasting,
dims=("steps",),
)
post_pred_forecast = pm.sample_posterior_predictive(
trace, var_names=["ar_steps"], random_seed=rng
)
Sampling: [ar_steps]
我们可以可视化样本外预测,并将这些结果与来自 statsmodels 的结果进行比较。
forecast_post_pred_ar = post_pred_forecast.posterior_predictive["ar_steps"]
_, ax = plt.subplots()
for i, hdi_prob in enumerate((0.94, 0.64), 1):
hdi = az.hdi(conditional_post_pred_ar, hdi_prob=hdi_prob)["ar"]
lower = hdi.sel(hdi="lower")
upper = hdi.sel(hdi="higher")
ax.fill_between(
x=conditional_post_pred_ar["timeseries_length"],
y1=lower,
y2=upper,
alpha=(i - 0.2) * 0.2,
color="C1",
label=f"{hdi_prob:.0%} HDI",
)
ax.plot(
conditional_post_pred_ar["timeseries_length"],
conditional_post_pred_ar.mean(("chain", "draw")),
color="C1",
label="Mean",
)
for i, hdi_prob in enumerate((0.94, 0.64), 1):
hdi = az.hdi(forecast_post_pred_ar, hdi_prob=hdi_prob)["ar_steps"]
lower = hdi.sel(hdi="lower")
upper = hdi.sel(hdi="higher")
ax.fill_between(
x=forecast_post_pred_ar["steps"],
y1=lower,
y2=upper,
alpha=(i - 0.2) * 0.2,
color="C3",
label=f"{hdi_prob:.0%} HDI forecast",
)
ax.plot(
forecast_post_pred_ar["steps"],
forecast_post_pred_ar.mean(("chain", "draw")),
color="C3",
label="Mean Forecast",
)
ax.plot(ar_obs, color="black", label="Observed")
ax.plot(
conditional_post_pred_ar["timeseries_length"],
res.fittedvalues,
color="C2",
label="statsmodels",
)
ax.plot(
forecast_post_pred_ar["steps"],
res.forecast(forecast_steps),
color="C2",
label="statsmodels forecast",
)
ax.legend(loc="upper center", bbox_to_anchor=(0.5, -0.1), ncol=4)
ax.set_xlabel("time")
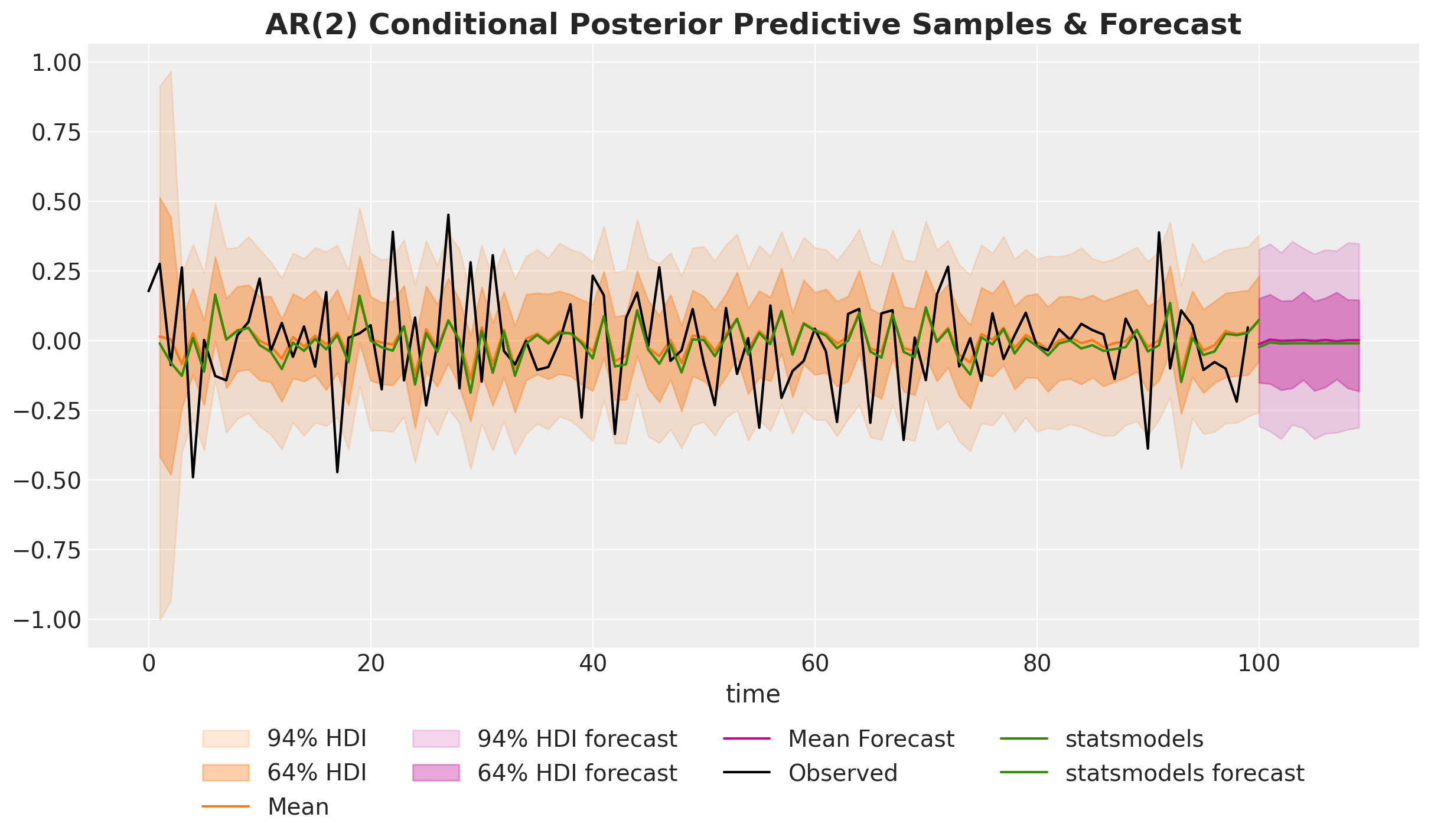
ax.set_title(
"AR(2) Conditional Posterior Predictive Samples & Forecast",
fontsize=18,
fontweight="bold",
);
水印#
%load_ext watermark
%watermark -n -u -v -iv -w -p pytensor
Last updated: Fri Jan 17 2025
Python implementation: CPython
Python version : 3.12.8
IPython version : 8.31.0
pytensor: 2.26.4
numpy : 1.26.4
arviz : 0.20.0
pymc : 5.20.0
statsmodels: 0.14.4
matplotlib : 3.10.0
pytensor : 2.26.4
Watermark: 2.5.0
许可声明#
此示例库中的所有笔记本均在 MIT 许可证下提供,该许可证允许修改和重新分发以用于任何用途,前提是保留版权和许可声明。
引用 PyMC 示例#
要引用此笔记本,请使用 Zenodo 为 pymc-examples 存储库提供的 DOI。
重要提示
许多笔记本都改编自其他来源:博客、书籍…… 在这种情况下,您也应该引用原始来源。
另请记住引用代码中使用的相关库。
这是一个 bibtex 中的引用模板
@incollection{citekey,
author = "<notebook authors, see above>",
title = "<notebook title>",
editor = "PyMC Team",
booktitle = "PyMC examples",
doi = "10.5281/zenodo.5654871"
}
渲染后可能如下所示