使用数据容器#
import warnings
import arviz as az
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
import pymc as pm
from numpy.random import default_rng
plt.rcParams["figure.constrained_layout.use"] = True
print(f"Running on PyMC v{pm.__version__}")
Running on PyMC v5.16.2
%matplotlib inline
%config InlineBackend.figure_format = 'retina'
RANDOM_SEED = sum(map(ord, "Data Containers in PyMC"))
rng = default_rng(RANDOM_SEED)
az.style.use("arviz-darkgrid")
简介#
在构建您梦想中的统计模型之后,您将需要向其输入一些数据。数据通常以两种方式引入 PyMC 模型。一些数据用作外生输入,在线性回归模型中称为 X,其中 mu = X @ beta。其他数据是模型内生输出的“观察”示例,在回归模型中称为 y,并用作模型隐含的似然函数的输入。这些数据,无论是外生的还是内生的,都可以以各种数据类型包含在您的模型中,包括 numpy ndarrays、pandas Series 和 DataFrame,甚至 pytensor TensorVariables。
虽然您可以将这些“原始”数据类型传递给您的 PyMC 模型,但将数据引入模型的最佳方式是使用 pymc.Data 容器。这些容器使在 PyMC 模型中使用数据变得非常容易。它们提供了一系列好处,包括
将数据可视化为概率图的组件
访问带标签的维度以提高可读性和可访问性
支持交换数据以进行样本外预测、插值/外推、预测等。
所有数据都将存储在您的
arviz.InferenceData中,这对于绘图和可重复的工作流程非常有用。
本笔记本将依次说明这些好处,并向您展示将数据集成到 PyMC 建模工作流程中的最佳方法。
重要提示
在 PyMC 的过去版本中,有两种类型的数据容器 pymc.MutableData() 和 pymc.ConstantData()。这些已被弃用,因为所有数据容器现在都是可变的。
使用数据容器提高可读性和可重复性#
该示例展示了使用数据容器和“原始”数据之间的一些差异。第一个模型展示了如何将原始数据(在本例中为 numpy 数组)直接提供给 PyMC 模型。
true_beta = 3
true_std = 5
n_obs = 100
x = rng.normal(size=n_obs)
y = rng.normal(loc=true_beta * x, scale=true_std, size=n_obs)
with pm.Model() as no_data_model:
beta = pm.Normal("beta")
mu = pm.Deterministic("mu", beta * x)
sigma = pm.Exponential("sigma", 1)
obs = pm.Normal("obs", mu=mu, sigma=sigma, observed=y)
idata = pm.sample(random_seed=RANDOM_SEED)
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [beta, sigma]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 1 seconds.
查看生成的计算图,obs 节点呈灰色阴影,表示它具有观察到的数据,在本例中为 y。但数据本身未显示在图上,因此没有任何关于观察到什么数据的提示。此外,x 数据未在图中的任何位置显示,因此不明显该模型使用了外生数据作为输入。
pm.model_to_graphviz(no_data_model)
此外,在 idata 内部,PyMC 已自动保存了观察到的(内生)y 数据,但未保存外生 x 数据。如果我们想保存此推断数据以供重用,或使其可用作可重复研究包的一部分,我们将必须确保单独包含 x 数据。
idata.observed_data
<xarray.Dataset> Size: 2kB
Dimensions: (obs_dim_0: 100)
Coordinates:
* obs_dim_0 (obs_dim_0) int64 800B 0 1 2 3 4 5 6 7 ... 93 94 95 96 97 98 99
Data variables:
obs (obs_dim_0) float64 800B -0.3966 -3.337 -7.844 ... -6.549 -0.8598
Attributes:
created_at: 2024-07-28T11:34:21.141826+00:00
arviz_version: 0.19.0
inference_library: pymc
inference_library_version: 5.16.2在下一个模型中,我们创建一个 pymc.Data 容器来保存观测值,并将此容器传递给 observed。我们还创建一个 pymc.Data 容器来保存 x 数据
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [beta, sigma]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 1 seconds.
因为我们使用了 pymc.Data 容器,所以数据现在出现在我们的概率图中。它位于 obs 的下游(因为 obs 变量“导致”数据),以灰色阴影显示(因为它已被观察到),并且具有特殊的圆形正方形形状,以强调它是数据。我们还看到 x_data 已添加到图中。
pm.model_to_graphviz(no_data_model)
最后,我们可以检查推断数据,以查看 x 数据也已存储在那里。它现在是重现我们模型所需的所有信息的完整摘要。
idata.constant_data
<xarray.Dataset> Size: 3kB
Dimensions: (x_data_dim_0: 100, y_data_dim_0: 100)
Coordinates:
* x_data_dim_0 (x_data_dim_0) int64 800B 0 1 2 3 4 5 6 ... 94 95 96 97 98 99
* y_data_dim_0 (y_data_dim_0) int64 800B 0 1 2 3 4 5 6 ... 94 95 96 97 98 99
Data variables:
x_data (x_data_dim_0) float64 800B -1.383 -0.2725 ... -1.745 -0.5087
y_data (y_data_dim_0) float64 800B -0.3966 -3.337 ... -6.549 -0.8598
Attributes:
created_at: 2024-07-28T11:34:24.969470+00:00
arviz_version: 0.19.0
inference_library: pymc
inference_library_version: 5.16.2使用数据容器的命名维度#
命名维度是使用数据容器的另一个强大优势。数据容器允许用户跟踪多维数据的维度(如日期或城市)和坐标(如实际日期时间或城市名称)。两者都允许您指定随机变量的维度名称和坐标,而不是将这些随机变量的形状指定为数字。请注意,在之前的概率图中,所有节点 x_data、mu、obs 和 y_data 都在一个带有数字 100 的框中。读者自然会问一个问题:“100 什么?”。维度和坐标通过准确回答这个问题来帮助组织模型、变量和数据。
在下一个示例中,我们将生成一个由 3 个城市在 2 个月内的温度组成的人工数据集。然后,我们将使用命名维度和坐标来提高模型代码的可读性和可视化的质量。
df_data = pd.DataFrame(columns=["date"]).set_index("date")
dates = pd.date_range(start="2020-05-01", end="2020-07-01")
for city, mu in {"Berlin": 15, "San Marino": 18, "Paris": 16}.items():
df_data[city] = rng.normal(loc=mu, size=len(dates))
df_data.index = dates
df_data.index.name = "date"
df_data.head()
| 柏林 | 圣马力诺 | 巴黎 | |
|---|---|---|---|
| 日期 | |||
| 2020-05-01 | 15.401536 | 18.817801 | 16.836690 |
| 2020-05-02 | 13.575241 | 17.441153 | 14.407089 |
| 2020-05-03 | 14.808934 | 19.890369 | 15.616649 |
| 2020-05-04 | 16.071487 | 18.407539 | 15.396678 |
| 2020-05-05 | 15.505263 | 17.621143 | 16.723544 |
如上所述,pymc.Data 使您能够为数据的维度提供命名标签。这通过将 dimension: coordinate 键值对字典传递给创建模型时 pymc.Model 的 coords 参数来完成。
有关维度、坐标及其巨大优势的更多解释,我们鼓励您查看 ArviZ 文档。
这有很多解释——让我们看看它是如何完成的!我们将使用分层模型:它假设欧洲大陆的平均温度,并对每个城市相对于大陆平均温度进行建模
# The data has two dimensions: date and city
# The "coordinates" are the unique values that these dimensions can take
coords = {"date": df_data.index, "city": df_data.columns}
with pm.Model(coords=coords) as model:
data = pm.Data("observed_temp", df_data, dims=("date", "city"))
europe_mean = pm.Normal("europe_mean_temp", mu=15.0, sigma=3.0)
city_offset = pm.Normal("city_offset", mu=0.0, sigma=3.0, dims="city")
city_temperature = pm.Deterministic(
"expected_city_temp", europe_mean + city_offset, dims="city"
)
sigma = pm.Exponential("sigma", 1)
pm.Normal("temperature", mu=city_temperature, sigma=sigma, observed=data, dims=("date", "city"))
idata = pm.sample(
random_seed=RANDOM_SEED,
)
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [europe_mean_temp, city_offset, sigma]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 6 seconds.
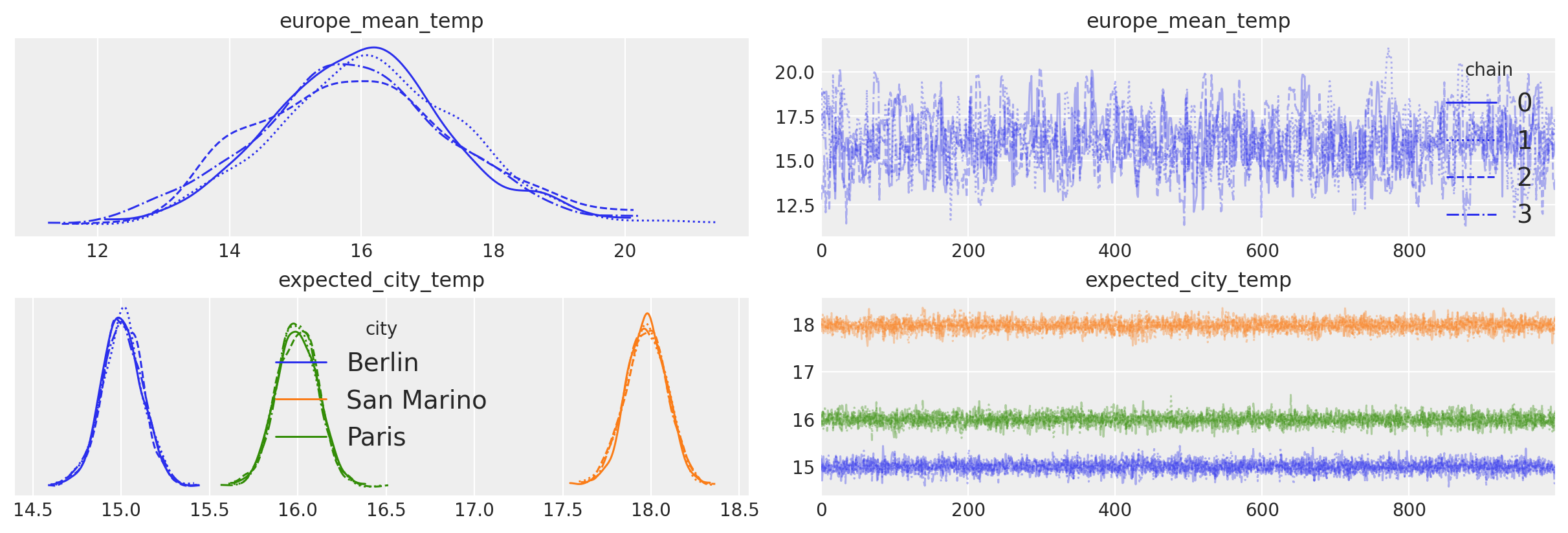
再一次,我们可以查看模型隐含的概率图。和以前一样,相似的节点(或节点组)被分组在盘子内部。盘子用每个节点的维度标记。之前,我们有标签 100 并问:“100 什么?”。现在我们看到在我们的模型中,有 3 个城市和 62 个日期。3 个城市中的每个城市都有自己的偏移量,这与组平均值一起决定了估计温度。最后,我们看到我们的数据实际上是一个 2d 矩阵。添加带标签的维度大大改进了我们概率模型的呈现。
pm.model_to_graphviz(model)
我们看到模型确实记住了我们给它的坐标
for k, v in model.coords.items():
print(f"{k}: {v[:20]}")
date: (Timestamp('2020-05-01 00:00:00'), Timestamp('2020-05-02 00:00:00'), Timestamp('2020-05-03 00:00:00'), Timestamp('2020-05-04 00:00:00'), Timestamp('2020-05-05 00:00:00'), Timestamp('2020-05-06 00:00:00'), Timestamp('2020-05-07 00:00:00'), Timestamp('2020-05-08 00:00:00'), Timestamp('2020-05-09 00:00:00'), Timestamp('2020-05-10 00:00:00'), Timestamp('2020-05-11 00:00:00'), Timestamp('2020-05-12 00:00:00'), Timestamp('2020-05-13 00:00:00'), Timestamp('2020-05-14 00:00:00'), Timestamp('2020-05-15 00:00:00'), Timestamp('2020-05-16 00:00:00'), Timestamp('2020-05-17 00:00:00'), Timestamp('2020-05-18 00:00:00'), Timestamp('2020-05-19 00:00:00'), Timestamp('2020-05-20 00:00:00'))
city: ('Berlin', 'San Marino', 'Paris')
坐标会自动存储到 arviz.InferenceData 对象中
idata.posterior.coords
Coordinates:
* chain (chain) int64 32B 0 1 2 3
* draw (draw) int64 8kB 0 1 2 3 4 5 6 7 ... 993 994 995 996 997 998 999
* city (city) <U10 120B 'Berlin' 'San Marino' 'Paris'
坐标也由 arviz 在制作图表时使用。这里我们传递 legend=True 到 az.plot_trace 以自动标记我们的迹线图中的三个城市。
axes = az.plot_trace(idata, var_names=["europe_mean_temp", "expected_city_temp"], legend=True);
当我们使用 pymc.Data 时,数据在内部表示为 pytensor pytensor.tensor.sharedvar.TensorSharedVariable。
type(data)
pytensor.tensor.sharedvar.TensorSharedVariable
可以使用 pytensor.graph.Variable.eval() 方法检查所有 pytensor 张量的值。要访问数据,我们可以使用 pymc.Model 对象。可以通过使用变量名称索引模型对象本身来访问所有模型变量,包括数据容器。由于这行代码
data = pm.Data("observed_temp", df_data, dims=("date", "city"))
将名称“observed_temp”赋予数据,我们可以按如下方式访问它
model["observed_temp"].eval()[:15]
array([[15.40153619, 18.81780064, 16.83668986],
[13.57524128, 17.4411525 , 14.40708945],
[14.80893441, 19.89036942, 15.61664911],
[16.07148689, 18.40753876, 15.39667778],
[15.50526329, 17.62114292, 16.72354378],
[14.86774578, 16.91297071, 16.64398094],
[15.39861564, 18.06957647, 16.8952898 ],
[14.82344668, 17.8925678 , 18.13033425],
[14.29595165, 18.18139386, 14.87228836],
[13.7296701 , 18.6225866 , 15.67262894],
[14.68800267, 17.48605691, 15.26520975],
[14.62891834, 19.16219466, 16.71921201],
[14.58253668, 17.09701213, 17.10149473],
[14.39255946, 17.66357467, 15.68899804],
[15.8653625 , 16.66259542, 16.69857121]])
使用数据容器来改变数据#
在许多情况下,您需要能够在模型运行之间切换数据。当您想要将模型拟合到多个数据集、您对样本外预测感兴趣以及在许多其他应用中,都会出现这种情况。
使用数据容器变量将同一模型拟合到多个数据集#
我们可以在 PyMC 中使用 Data 容器变量将同一模型拟合到多个数据集,而无需每次都重新创建模型(如果数据集数量很大,这可能会很耗时)。但请注意,模型仍然会在每次重新编译。
在下一个示例中,我们将生成 10 个具有单个参数 mu 的模型。每个模型将有一个数据集,其中包含不同数量的观测值,从 10 到 100。我们将使用此设置来探索数据量对参数恢复的影响。
# We generate 10 datasets
n_models = 10
obs_multiplier = 10
true_mu = [rng.random() for _ in range(n_models)]
observed_data = [mu + rng.normal(size=(i + 1) * obs_multiplier) for i, mu in enumerate(true_mu)]
with pm.Model() as model:
data = pm.Data("data", observed_data[0])
mu = pm.Normal("mu", 0, 10)
pm.Normal("y", mu=mu, sigma=1, observed=data)
正如我们之前展示的那样,我们可以使用 eval 方法来检查我们的 Data 容器值
model["data"].eval()
array([-0.10562792, 0.62102496, 1.48658991, 0.92295128, 0.43538261,
0.73235925, -0.11983016, 0.89501808, -0.39242869, 1.4783441 ])
虽然 eval 可用于检查值,但 pymc.set_data() 用于更改它们。让我们将 Data 与 pymc.set_data 一起使用,以将同一模型重复拟合到多个数据集。请注意,每个数据集的大小不同无关紧要!
# Generate one trace for each dataset
idatas = []
for data_vals in observed_data:
with model:
# Switch out the observed dataset
pm.set_data({"data": data_vals})
idatas.append(pm.sample(random_seed=RANDOM_SEED))
显示代码单元格输出
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [mu]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 1 seconds.
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [mu]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 1 seconds.
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [mu]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 1 seconds.
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [mu]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 1 seconds.
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [mu]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 1 seconds.
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [mu]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 1 seconds.
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [mu]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 1 seconds.
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [mu]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 1 seconds.
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [mu]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 1 seconds.
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [mu]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 1 seconds.
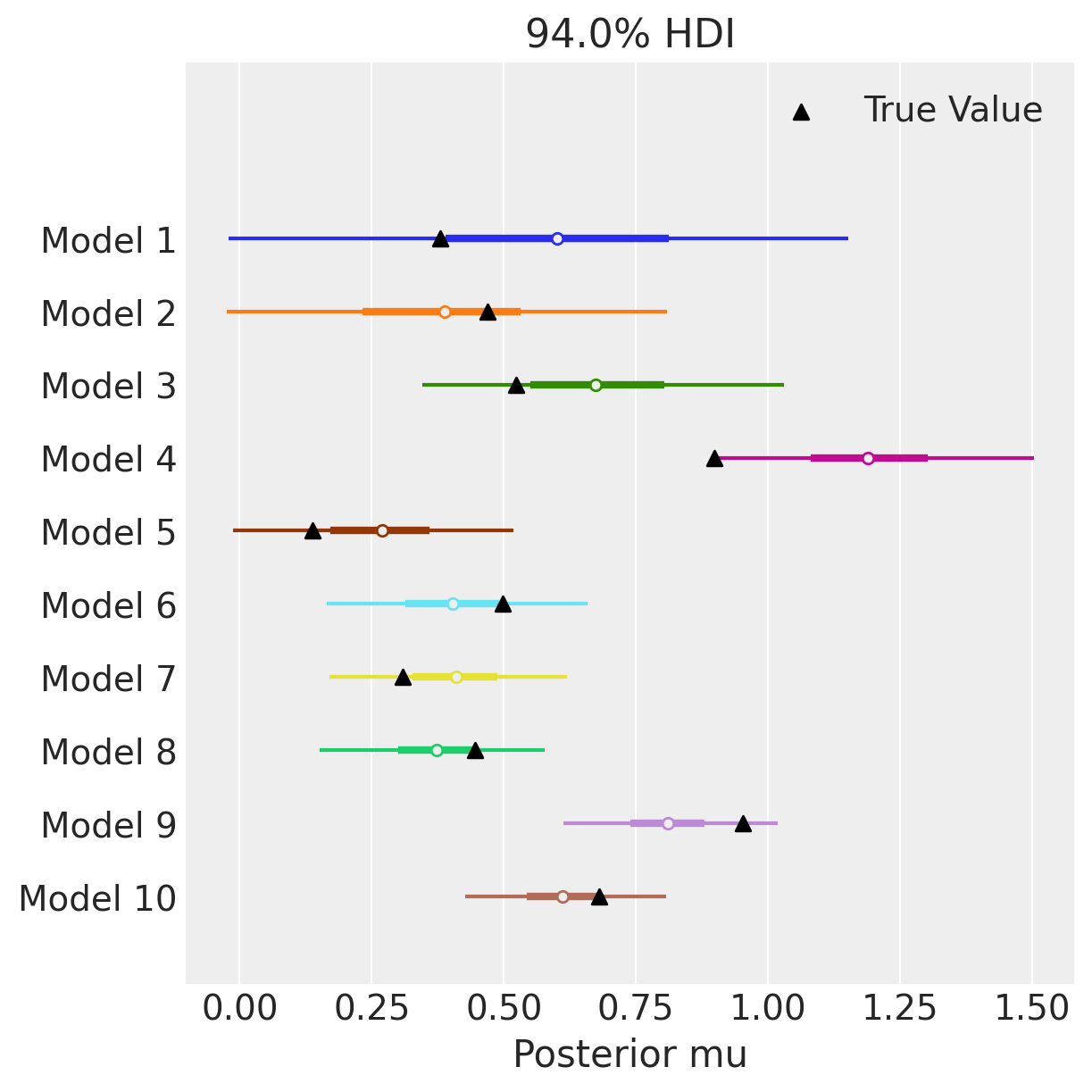
Arviz 的 arviz.plot_forest() 允许您传递具有相同变量名称的 idata 对象列表。通过这种方式,我们可以快速可视化来自 10 个不同数据集的不同估计值。我们还使用 matplotlib 将真实参数值散点在 plot_forest 的顶部。我们可以看到,当我们从模型 1 中的 10 个观测值变为模型 10 中的 100 个观测值时,估计值越来越集中在 mu 的真实值上,并且围绕估计值的不确定性降低。
model_idx = np.arange(n_models, dtype="int")
axes = az.plot_forest(idatas, var_names=["mu"], combined=True, figsize=(6, 6), legend=False)
ax = axes[0]
y_vals = np.stack([ax.get_lines()[i].get_ydata() for i in np.arange(n_models)]).ravel()
ax.scatter(true_mu, y_vals[::-1], marker="^", color="k", zorder=100, label="True Value")
ax.set(yticklabels=[f"Model {i+1}" for i in model_idx[::-1]], xlabel="Posterior mu")
ax.legend()
plt.show()
应用示例:使用数据容器作为二项式 GLM 的输入#
机器学习中的一个常见任务是预测看不见的数据的值,而 Data 容器变量正是我们执行此操作所需的。
在这种情况下,需要注意的一个小细节是输入数据 (x) 和输出数据 (obs) 的形状必须相同。当我们进行样本外预测时,我们通常只更改输入数据,其形状可能与训练观测值不同。天真地只更改一个会导致形状错误。有两种解决方案
为
x数据和y数据使用pymc.Data,并使用pm.set_data将y更改为与测试输入形状相同的内容。告诉 PyMC
obs的形状应始终为输入数据的形状。
在下一个模型中,我们使用选项 2。这样,我们不需要每次想要更改 x 时都将虚拟数据传递给 y。
n_obs = 100
true_beta = 1.5
true_alpha = 0.25
x = rng.normal(size=n_obs)
true_p = 1 / (1 + np.exp(-(true_alpha + true_beta * x)))
y = rng.binomial(n=1, p=true_p)
with pm.Model() as logistic_model:
x_data = pm.Data("x", x)
y_data = pm.Data("y", y)
alpha = pm.Normal("alpha")
beta = pm.Normal("beta")
p = pm.Deterministic("p", pm.math.sigmoid(alpha + beta * x_data))
# Here is were we link the shapes of the inputs (x_data) and the observed variable
# It will be the shape we tell it, rather than the (constant!) shape of y_data
obs = pm.Bernoulli("obs", p=p, observed=y_data, shape=x_data.shape[0])
# fit the model
idata = pm.sample(random_seed=RANDOM_SEED)
# Generate a counterfactual dataset using our model
idata = pm.sample_posterior_predictive(
idata, extend_inferencedata=True, random_seed=RANDOM_SEED
)
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [alpha, beta]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 1 seconds.
Sampling: [obs]
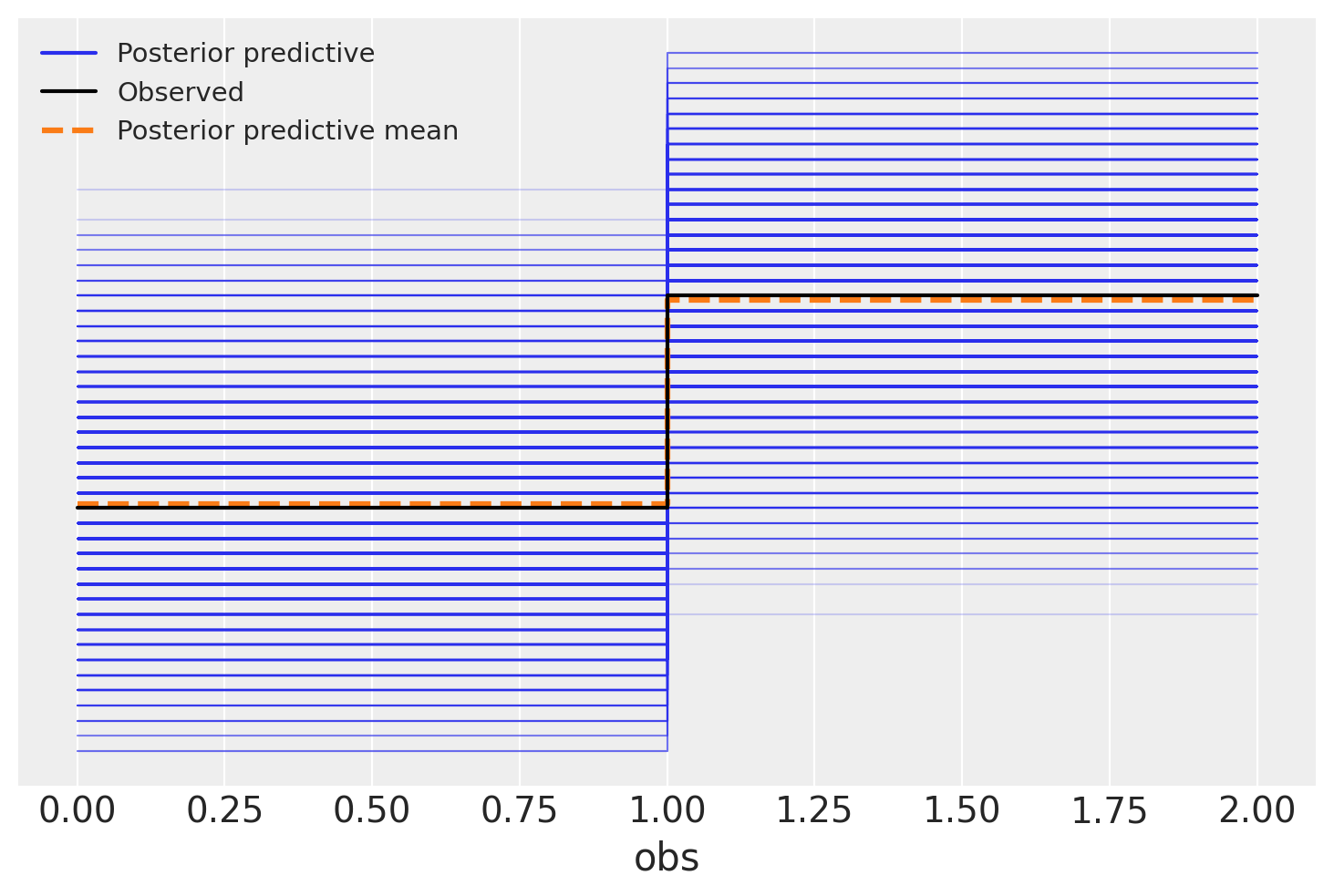
一个常见的后验估计诊断是查看后验预测图,使用 arviz.plot_ppc()。这显示了从您的模型中采样的数据分布以及观察到的数据。如果它们看起来相似,我们有一些证据表明该模型还不错。
然而,在这种情况下,可能难以解释后验预测图。由于我们正在进行逻辑回归,因此观察到的值只能为零或一。因此,后验预测图具有这种俄罗斯方块形状。我们该如何理解它?显然,我们的模型产生的 1 比 0 多,并且平均比例与数据匹配。但该比例也存在很多不确定性。关于模型的性能,我们还能说些什么?
az.plot_ppc(idata)
<Axes: xlabel='obs'>
我们可以制作的另一个图表来查看我们的模型表现如何是查看潜在变量 p 如何在协变量空间中演变。我们期望协变量和数据之间存在某种关系,我们的模型将这种关系编码在变量 p 中。在此模型中,唯一的协变量是 x。如果 x 和 y 之间的关系是正的,我们期望在 x 很小时,p 低且观察到的零很多,而在 x 很大时,p 高且观察到的 1 很多。
这很好,但对于绘图,x 都混在一起了。诚然,我们可以只对值进行排序。但另一种方法(展示如何使用我们的 Data!)是将均匀间隔的 x 值网格传递到我们的模型中。这对应于在网格上的每个点对 p 进行预测,这将为我们提供一个很好的可绘制结果。这也是我们如何使用我们的模型进行插值或外推的方法,因此这是一个非常好的技巧。
在下一个代码块中,我们将(随机打乱的)x 值交换为跨越观察到的 x 范围的均匀间隔的值网格。
grid_size = 250
x_grid = np.linspace(x.min(), x.max(), grid_size)
with logistic_model:
# Switch out the observations and use `sample_posterior_predictive` to predict
# We do not need to set data for the outputs because we told the model to always link the shape of the output to the shape
# of the input.
pm.set_data({"x": x_grid})
post_idata = pm.sample_posterior_predictive(
idata, var_names=["p", "obs"], random_seed=RANDOM_SEED
)
Sampling: [obs]
使用新的 post_idata,它保存了 p 的样本外“预测”,我们制作了 x_grid 对 p 的图。我们还绘制了观察到的数据。我们可以看到,模型期望在 x 很小时概率 (p) 低,并且概率随 x 逐渐变化。
fig, ax = plt.subplots()
hdi = az.hdi(post_idata.posterior_predictive.p).p
ax.scatter(x, y, facecolor="none", edgecolor="k", label="Observed Data")
p_mean = post_idata.posterior_predictive.p.mean(dim=["chain", "draw"])
ax.plot(x_grid, p_mean, color="tab:red", label="Mean Posterior Probability")
ax.fill_between(x_grid, *hdi.values.T, color="tab:orange", alpha=0.25, label="94% HDI")
ax.legend()
ax.set(ylabel="Probability of $y=1$", xlabel="x value")
plt.show()

在笔记本 变分推断:贝叶斯神经网络 中可以看到应用于更复杂模型的相同概念。
应用示例:幼儿的身高作为年龄的函数#
此示例取自 Osvaldo Martin 的书:《Python 贝叶斯分析:使用 PyMC 和 ArviZ 进行统计建模和概率编程简介,第 2 版》 [Martin,2018]。
世界卫生组织和世界各地的其他卫生机构收集新生儿和幼儿的数据,并设计 生长图表标准。这些图表是儿科工具包的重要组成部分,也是衡量人群总体福祉的指标,以便制定卫生政策、规划干预措施和监测其有效性。
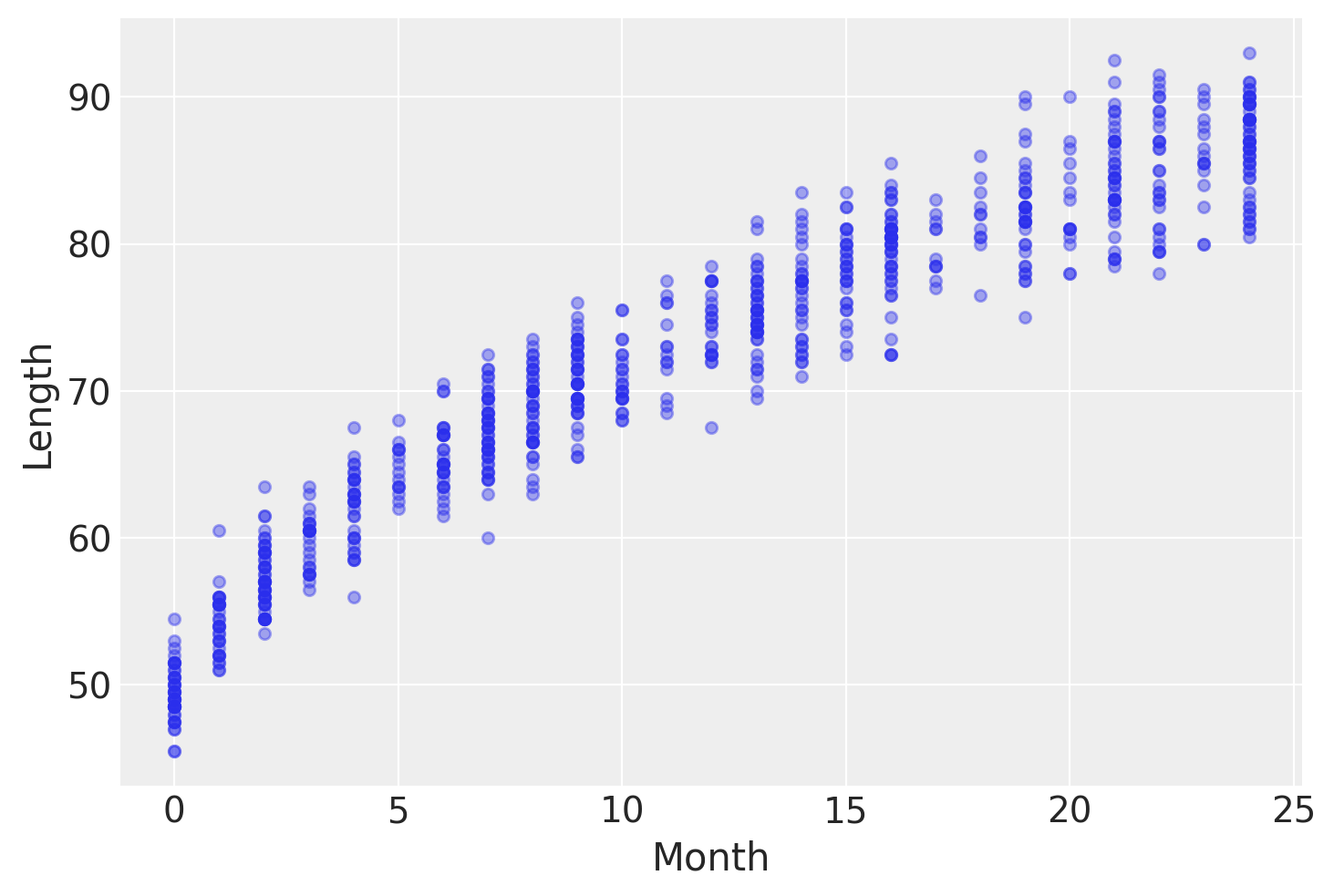
此类数据的一个示例是新生儿/幼儿女孩的长度(身高)与年龄(以月为单位)的函数关系
try:
data = pd.read_csv("../data/babies.csv")
except FileNotFoundError:
data = pd.read_csv(pm.get_data("babies.csv"))
data.plot.scatter("Month", "Length", alpha=0.4);
为了对这些数据进行建模,我们将需要改变 coords,因为数据的索引需要根据测试数据集的索引进行更改。幸运的是,coords 始终是可变的,因此我们可以在样本外预测期间对其进行改变。
在该示例中,我们将更新 obs_idx 的坐标以反映测试数据集的索引。
with pm.Model(
coords={"obs_idx": np.arange(len(data)), "parameter": ["intercept", "slope"]}
) as model_babies:
mean_params = pm.Normal("mean_params", sigma=10, dims=["parameter"])
sigma_params = pm.Normal("sigma_params", sigma=10, dims=["parameter"])
month = pm.Data("month", data.Month.values.astype(float), dims=["obs_idx"])
mu = pm.Deterministic("mu", mean_params[0] + mean_params[1] * month**0.5, dims=["obs_idx"])
sigma = pm.Deterministic("sigma", sigma_params[0] + sigma_params[1] * month, dims=["obs_idx"])
length = pm.Normal("length", mu=mu, sigma=sigma, observed=data.Length, dims=["obs_idx"])
idata_babies = pm.sample(random_seed=RANDOM_SEED)
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [mean_params, sigma_params]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 2 seconds.
下图显示了我们模型的结果。预期长度 \(\mu\) 用蓝色曲线表示,两个半透明的橙色带表示后验预测长度测量的 60% 和 94% 最高后验密度区间
with model_babies:
pm.sample_posterior_predictive(idata_babies, extend_inferencedata=True, random_seed=RANDOM_SEED)
Sampling: [length]
ax = az.plot_hdi(
data.Month,
idata_babies.posterior_predictive["length"],
hdi_prob=0.6,
fill_kwargs={"color": "tab:orange", "alpha": 0.8},
)
ax.plot(
data.Month,
idata_babies.posterior["mu"].mean(("chain", "draw")),
label="Posterior predictive mean",
)
ax = az.plot_lm(
idata=idata_babies,
y="length",
x="month",
kind_pp="hdi",
y_kwargs={"color": "k", "ms": 6, "alpha": 0.15},
y_hat_fill_kwargs=dict(fill_kwargs={"color": "tab:orange", "alpha": 0.4}),
axes=ax,
)

在撰写本文时,Osvaldo 的女儿出生才两周 (\(\approx 0.5\) 个月) 大,因此他想知道女儿的身长与我们刚刚创建的生长图表相比如何。回答这个问题的一种方法是向模型询问 0.5 个月婴儿的变量长度分布。使用 PyMC,我们可以使用函数 sample_posterior_predictive 提出这个问题,因为这将返回以观察到的数据和参数的估计分布为条件的 *Length* 样本,即包括不确定性。
唯一的问题是,默认情况下,此函数将返回 *Length* 的预测值,用于观察到的 *Month* 值,并且 \(0.5\) 个月(Osvaldo 关心的值)尚未被观察到——所有测量值都报告为整数月。获取 *Month* 的非观测值的预测值的更简单方法是将新值传递给我们模型中上面定义的 Data 容器。为此,我们需要使用 pm.set_data,然后我们只需从后验预测分布中采样。我们还需要为这些新观测值设置 coords,这是我们允许在 pm.set_data 函数中执行的操作,因为我们将 obs_idx 坐标设置为可变的。
请注意,在本例中,我们为 obs_idx 传递的实际值完全不相关,因此我们为其赋予值 0。重要的是,我们将其更新为与我们要进行样本外预测的年龄具有相同的长度,并且每个年龄都有唯一的索引标识符。
ages_to_check = [0.5]
with model_babies:
pm.set_data({"month": ages_to_check}, coords={"obs_idx": [0]})
# Setting predictions=True will add a new "predictions" group to our idata. This lets us store the posterior,
# posterior_predictive, and predictions all in the same object.
idata_babies = pm.sample_posterior_predictive(
idata_babies, extend_inferencedata=True, predictions=True, random_seed=RANDOM_SEED
)
Sampling: [length]
现在我们可以绘制 2 周大婴儿的预期长度分布并计算其他量——例如给定儿童身长的百分位数。在这里,让我们假设我们感兴趣的孩子身长为 51.5
ref_length = 51.5
az.plot_posterior(
idata_babies,
group="predictions",
ref_val={"length": [{"ref_val": ref_length}]},
labeller=az.labels.DimCoordLabeller(),
);

参考文献#
Osvaldo Martin. *Python 贝叶斯分析:使用 PyMC3 和 ArviZ 进行统计建模和概率编程简介*。Packt Publishing Ltd,2018 年。
水印#
%load_ext watermark
%watermark -n -u -v -iv -w -p pytensor,xarray
Last updated: Sun Jul 28 2024
Python implementation: CPython
Python version : 3.12.4
IPython version : 8.26.0
pytensor: 2.25.2
xarray : 2024.6.0
arviz : 0.19.0
numpy : 1.26.4
pymc : 5.16.2
matplotlib: 3.9.1
pandas : 2.2.2
Watermark: 2.4.3
许可证声明#
本示例库中的所有笔记本均根据 MIT 许可证 提供,该许可证允许修改和再分发以用于任何用途,前提是保留版权和许可证声明。
引用 PyMC 示例#
要引用此笔记本,请使用 Zenodo 为 pymc-examples 存储库提供的 DOI。
重要提示
许多笔记本改编自其他来源:博客、书籍……在这种情况下,您也应该引用原始来源。
另请记住引用您的代码使用的相关库。
这是一个 bibtex 中的引用模板
@incollection{citekey,
author = "<notebook authors, see above>",
title = "<notebook title>",
editor = "PyMC Team",
booktitle = "PyMC examples",
doi = "10.5281/zenodo.5654871"
}
渲染后可能如下所示