贝叶斯回归与截断或删失数据#
本笔记本提供了一个示例,说明如何在结果变量被删失或截断时进行线性回归。
from copy import copy
import arviz as az
import matplotlib.pyplot as plt
import numpy as np
import pymc as pm
import xarray as xr
from numpy.random import default_rng
from scipy.stats import norm, truncnorm
%config InlineBackend.figure_format = 'retina'
rng = default_rng(12345)
az.style.use("arviz-darkgrid")
截断和删失#
截断和删失是缺失数据问题的示例。有时很容易混淆截断和删失,因此让我们看一些定义。
截断是一种缺失数据问题,在这种问题中,您根本不知道结果变量落在特定范围之外的任何数据。
删失发生在测量具有特定范围的灵敏度时。但是,您不会丢弃这些范围之外的数据,而是会记录超出范围的边界处的测量值。
让我们用一些代码和图表进一步探讨这一点。首先,我们将生成一些真实的 (x, y) 散点数据,其中 y 是我们的结果度量,而 x 是一些预测变量。
slope, intercept, σ, N = 1, 0, 2, 200
x = rng.uniform(-10, 10, N)
y = rng.normal(loc=slope * x + intercept, scale=σ)
对于 (x, y) 散点数据的这个示例,我们可以将截断过程描述为简单地过滤掉结果变量 y 落在某个范围之外的任何数据。
def truncate_y(x, y, bounds):
keep = (y >= bounds[0]) & (y <= bounds[1])
return (x[keep], y[keep])
但是,对于删失,我们将 y 值设置为超出范围的边界值。
def censor_y(x, y, bounds):
cy = copy(y)
cy[y <= bounds[0]] = bounds[0]
cy[y >= bounds[1]] = bounds[1]
return (x, cy)
基于我们生成的 (x, y) 数据(实验者在现实生活中永远看不到),我们可以为截断数据 (xt, yt) 和删失数据 (xc, yc) 生成实际观察到的数据集。
bounds = [-5, 5]
xt, yt = truncate_y(x, y, bounds)
xc, yc = censor_y(x, y, bounds)
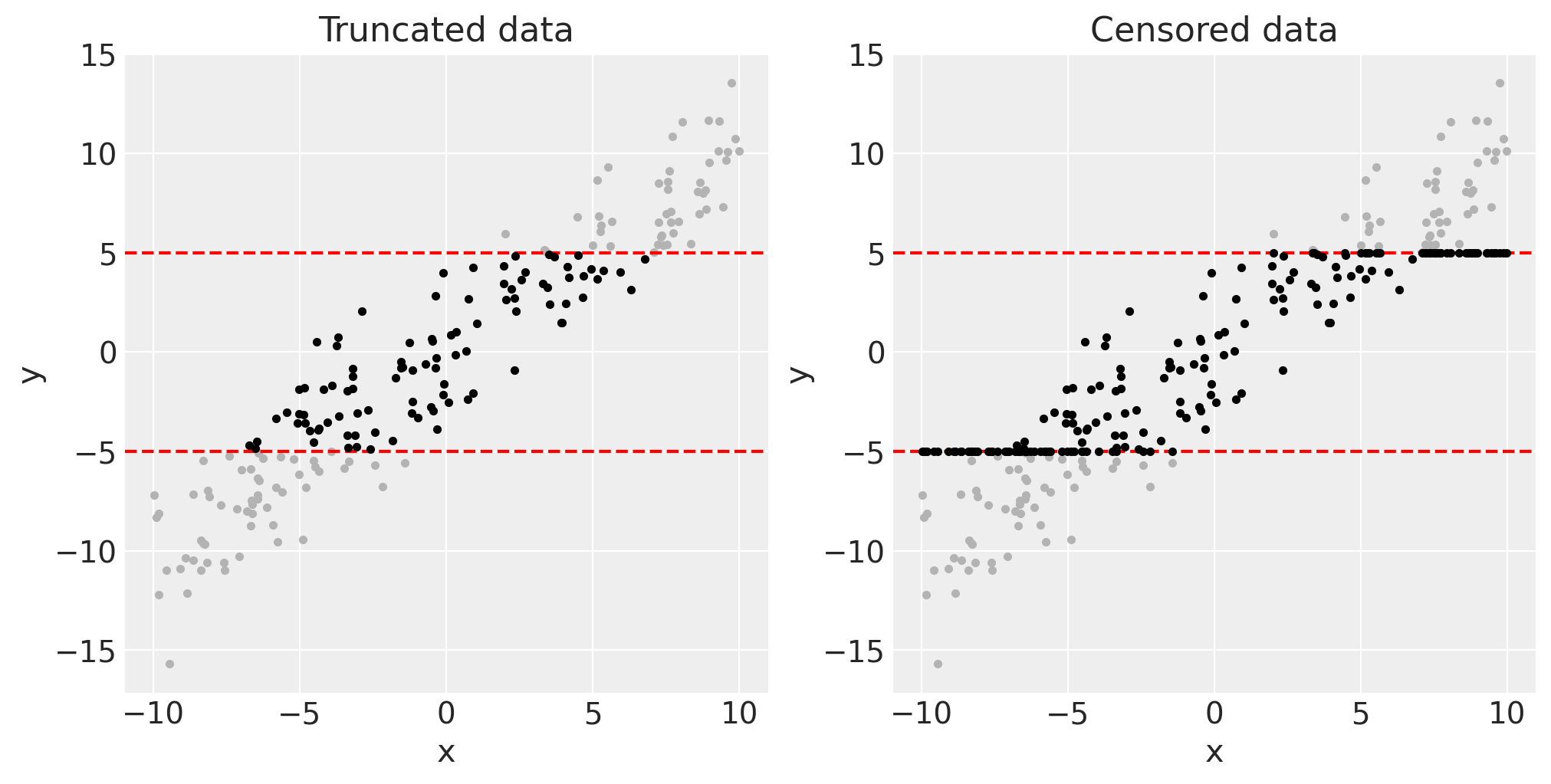
我们可以将此潜在数据(灰色)和剩余的截断或删失数据(黑色)可视化如下。
fig, axes = plt.subplots(1, 2, figsize=(10, 5))
for ax in axes:
ax.plot(x, y, ".", c=[0.7, 0.7, 0.7])
ax.axhline(bounds[0], c="r", ls="--")
ax.axhline(bounds[1], c="r", ls="--")
ax.set(xlabel="x", ylabel="y")
axes[0].plot(xt, yt, ".", c=[0, 0, 0])
axes[0].set(title="Truncated data")
axes[1].plot(xc, yc, ".", c=[0, 0, 0])
axes[1].set(title="Censored data");
截断或删失回归解决的问题#
如果我们在截断或删失数据上运行常规线性回归,那么很直观地可以看出,我们可能会低估斜率。截断回归和删失回归(也称为 Tobit 回归)旨在解决这些缺失数据问题,并希望得出不受截断或删失引入的偏差影响的回归斜率。
在本节中,我们将在这些数据集上运行贝叶斯线性回归,以了解问题的程度。我们首先定义一个函数,该函数定义 PyMC 模型,进行 MCMC 抽样,并返回模型和 MCMC 抽样数据。
def linear_regression(x, y):
with pm.Model() as model:
slope = pm.Normal("slope", mu=0, sigma=1)
intercept = pm.Normal("intercept", mu=0, sigma=1)
σ = pm.HalfNormal("σ", sigma=1)
pm.Normal("obs", mu=slope * x + intercept, sigma=σ, observed=y)
return model
因此,我们可以在截断数据和删失数据上分别运行此函数。
trunc_linear_model = linear_regression(xt, yt)
with trunc_linear_model:
trunc_linear_fit = pm.sample()
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 1 seconds.
cens_linear_model = linear_regression(xc, yc)
with cens_linear_model:
cens_linear_fit = pm.sample()
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 1 seconds.
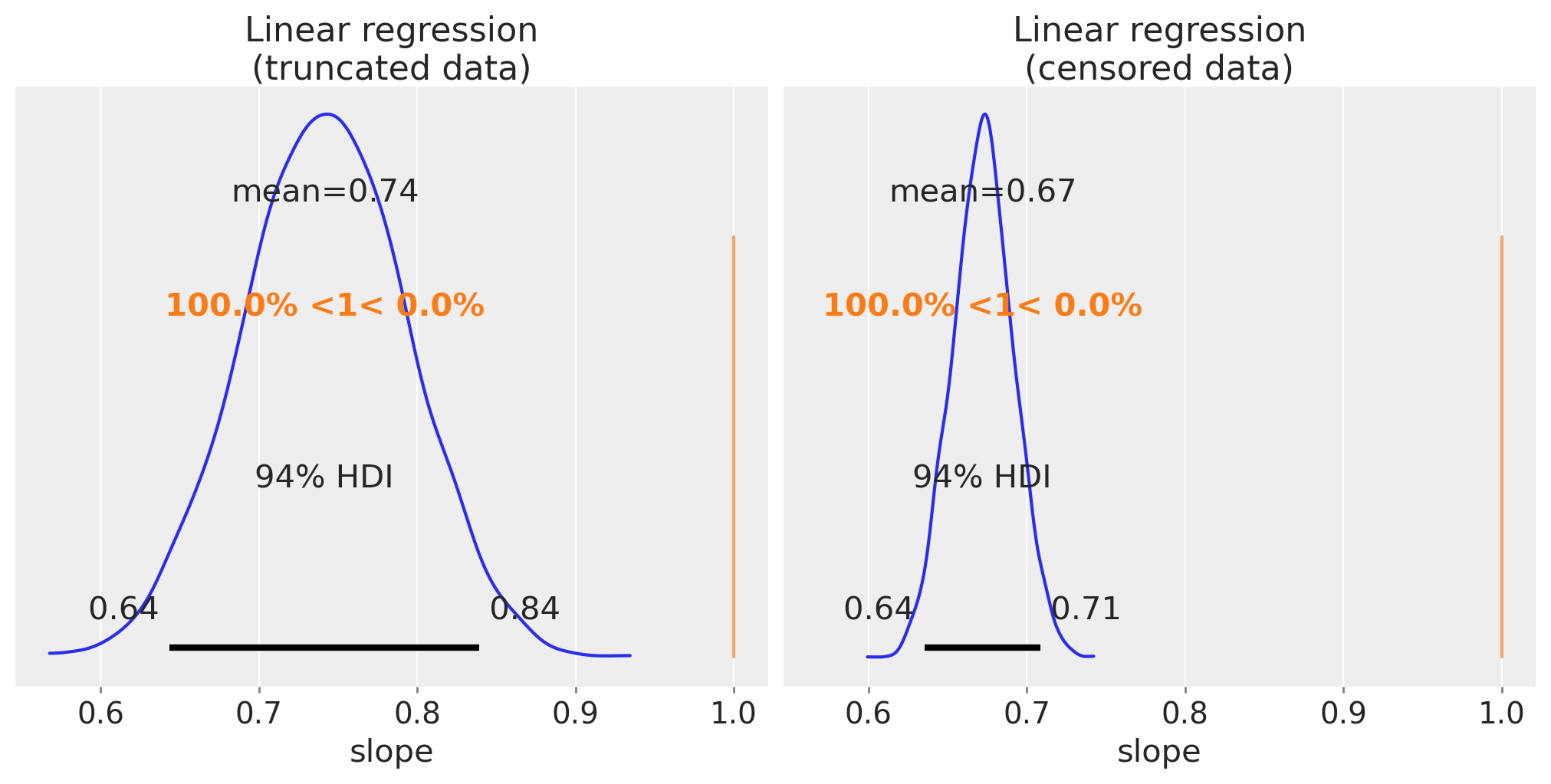
通过绘制斜率参数的后验分布,我们可以看到斜率的估计值相差甚远,因此我们确实低估了回归斜率。
fig, ax = plt.subplots(1, 2, figsize=(10, 5), sharex=True)
az.plot_posterior(trunc_linear_fit, var_names=["slope"], ref_val=slope, ax=ax[0])
ax[0].set(title="Linear regression\n(truncated data)", xlabel="slope")
az.plot_posterior(cens_linear_fit, var_names=["slope"], ref_val=slope, ax=ax[1])
ax[1].set(title="Linear regression\n(censored data)", xlabel="slope");
为了理解问题的程度(对于此数据集),我们可以将后验预测拟合与数据一起可视化。
def pp_plot(x, y, fit, ax):
# plot data
ax.plot(x, y, "k.")
# plot posterior predicted... samples from posterior
xi = xr.DataArray(np.array([np.min(x), np.max(x)]), dims=["obs_id"])
post = az.extract(fit)
y_ppc = xi * post["slope"] + post["intercept"]
ax.plot(xi, y_ppc, c="steelblue", alpha=0.01, rasterized=True)
# plot true
ax.plot(xi, slope * xi + intercept, "k", lw=3, label="True")
# plot bounds
ax.axhline(bounds[0], c="r", ls="--")
ax.axhline(bounds[1], c="r", ls="--")
ax.legend()
ax.set(xlabel="x", ylabel="y")
fig, ax = plt.subplots(1, 2, figsize=(10, 5), sharex=True, sharey=True)
pp_plot(xt, yt, trunc_linear_fit, ax[0])
ax[0].set(title="Truncated data")
pp_plot(xc, yc, cens_linear_fit, ax[1])
ax[1].set(title="Censored data");
通过查看这些图表,我们可以直观地预测哪些因素会影响低估偏差的程度。首先,如果截断或删失边界非常宽,以至于仅影响少量数据点,那么低估偏差将较小。其次,如果测量误差 σ 较低,我们可能会预期低估偏差会减小。在零测量噪声的极限情况下,应该可以完全恢复截断数据的真实斜率,但在删失情况下始终会存在一些偏差。无论如何,除非测量误差接近于零,或者边界非常宽以至于实际上无关紧要,否则使用截断或删失回归模型是明智的。
实施截断和删失回归模型#
现在我们已经看到了在截断或删失数据上进行回归的问题,即低估回归斜率。这就是设计截断或删失回归模型要解决的问题。截断回归和删失回归采用的通用方法是在数据生成过程中编码我们关于截断或删失步骤的先验知识。这是通过以各种方式修改似然函数来完成的。
截断回归模型#
截断回归模型非常容易实现。正态似然以回归斜率为中心,与正常情况一样,但现在我们只需指定一个在边界处截断的正态分布即可。
def truncated_regression(x, y, bounds):
with pm.Model() as model:
slope = pm.Normal("slope", mu=0, sigma=1)
intercept = pm.Normal("intercept", mu=0, sigma=1)
σ = pm.HalfNormal("σ", sigma=1)
normal_dist = pm.Normal.dist(mu=slope * x + intercept, sigma=σ)
pm.Truncated("obs", normal_dist, lower=bounds[0], upper=bounds[1], observed=y)
return model
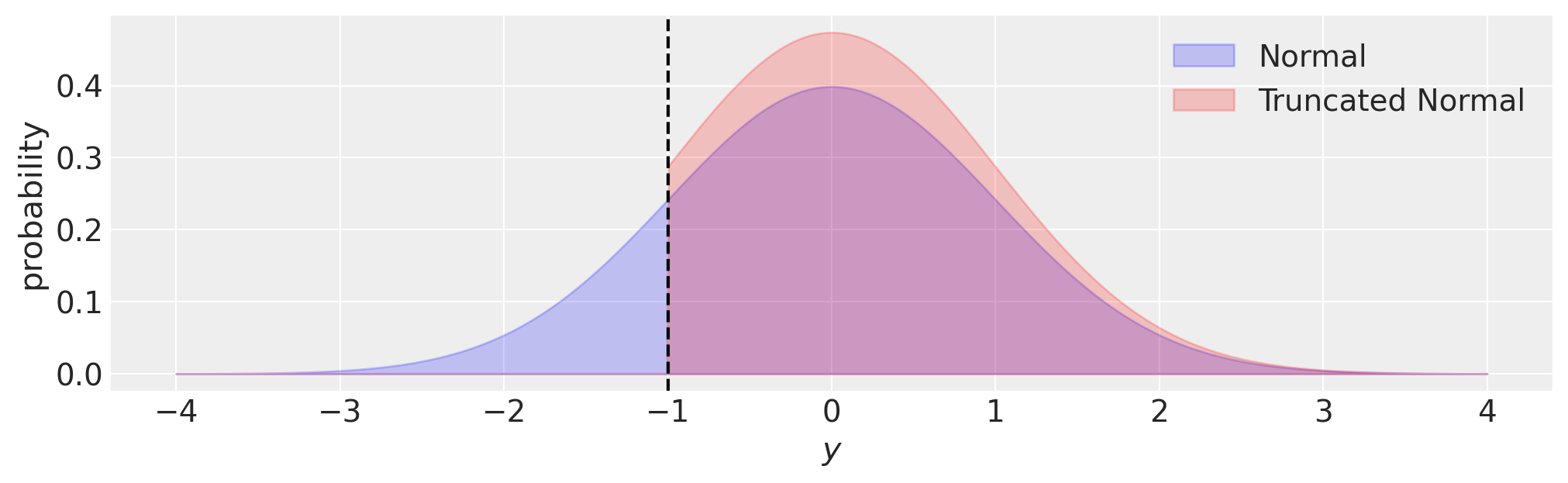
截断回归通过更新似然函数以反映我们对生成观测值的过程的了解来解决偏差问题。也就是说,我们观察到任何超出截断边界的数据的可能性为零,因此似然函数应反映这一点。我们可以在下图的可视化中看到这一点,与正态分布相比,截断正态分布的概率密度在截断边界 \((y<-1)\) 之外为零(在本例中)。
fig, ax = plt.subplots(figsize=(10, 3))
y = np.linspace(-4, 4, 1000)
ax.fill_between(y, norm.pdf(y, loc=0, scale=1), 0, alpha=0.2, ec="b", fc="b", label="Normal")
ax.fill_between(
y,
truncnorm.pdf(y, -1, float("inf"), loc=0, scale=1),
0,
alpha=0.2,
ec="r",
fc="r",
label="Truncated Normal",
)
ax.set(xlabel="$y$", ylabel="probability")
ax.axvline(-1, c="k", ls="--")
ax.legend();
删失回归模型#
def censored_regression(x, y, bounds):
with pm.Model() as model:
slope = pm.Normal("slope", mu=0, sigma=1)
intercept = pm.Normal("intercept", mu=0, sigma=1)
σ = pm.HalfNormal("σ", sigma=1)
y_latent = pm.Normal.dist(mu=slope * x + intercept, sigma=σ)
obs = pm.Censored("obs", y_latent, lower=bounds[0], upper=bounds[1], observed=y)
return model
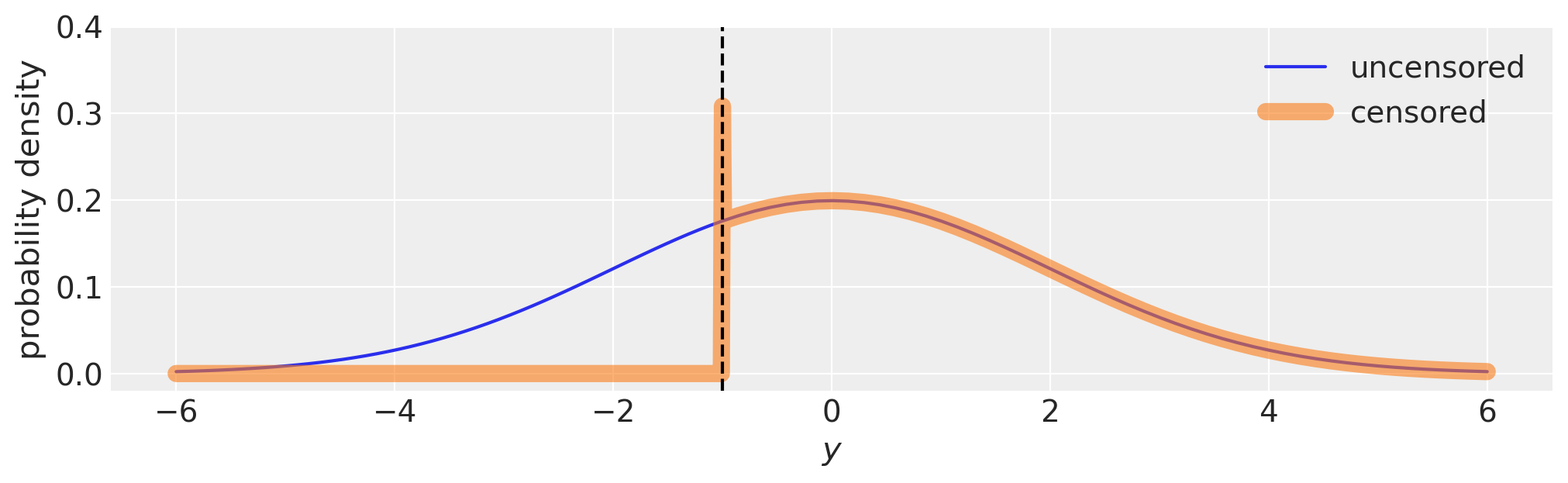
得益于新的 pm.Censored 分布,编写具有删失数据的模型非常简单。唯一要记住的是,被删失的潜在变量必须使用 .dist 方法调用,如上面模型中的 pm.Normal.dist。
在幕后,pm.Censored 调整了似然函数,以考虑到
下限处的概率等于从 \(-\infty\) 到下限的累积分布函数,
上限处的概率等于从上限到 \(\infty\) 的累积分布函数。
这在下图的可视化中得到了演示。从技术上讲,边界处的概率密度是无限的,因为边界处的确切 bin 宽度为零。
fig, ax = plt.subplots(figsize=(10, 3))
with pm.Model() as m:
pm.Normal("y", 0, 2)
with pm.Model() as m_censored:
pm.Censored("y", pm.Normal.dist(0, 2), lower=-1.0, upper=None)
logp_fn = m.compile_logp()
logp_censored_fn = m_censored.compile_logp()
xi = np.hstack((np.linspace(-6, -1.01), [-1.0], np.linspace(-0.99, 6)))
ax.plot(xi, [np.exp(logp_fn({"y": x})) for x in xi], label="uncensored")
ax.plot(xi, [np.exp(logp_censored_fn({"y": x})) for x in xi], label="censored", lw=8, alpha=0.6)
ax.axvline(-1, c="k", ls="--")
ax.legend()
ax.set(xlabel="$y$", ylabel="probability density", ylim=(-0.02, 0.4));
运行截断回归和删失回归#
现在我们可以使用截断回归模型对截断数据进行参数估计了…
truncated_model = truncated_regression(xt, yt, bounds)
with truncated_model:
truncated_fit = pm.sample()
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 1 seconds.
并使用删失回归模型对删失数据进行参数估计。
censored_model = censored_regression(xc, yc, bounds)
with censored_model:
censored_fit = pm.sample(init="adapt_diag")
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 1 seconds.
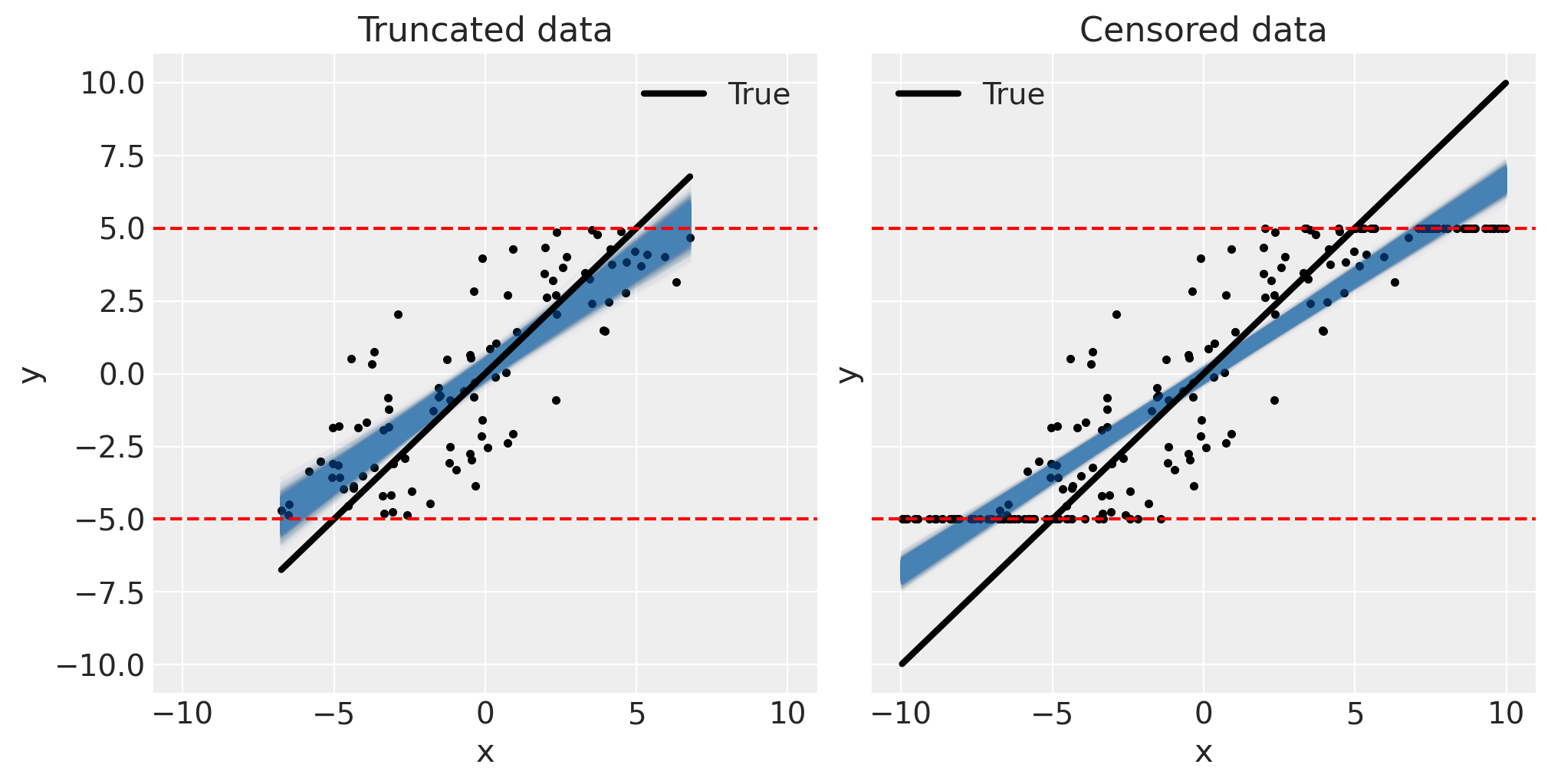
我们可以像以前一样,将斜率的后验估计值可视化。
fig, ax = plt.subplots(1, 2, figsize=(10, 5), sharex=True)
az.plot_posterior(truncated_fit, var_names=["slope"], ref_val=slope, ax=ax[0])
ax[0].set(title="Truncated regression\n(truncated data)", xlabel="slope")
az.plot_posterior(censored_fit, var_names=["slope"], ref_val=slope, ax=ax[1])
ax[1].set(title="Censored regression\n(censored data)", xlabel="slope");
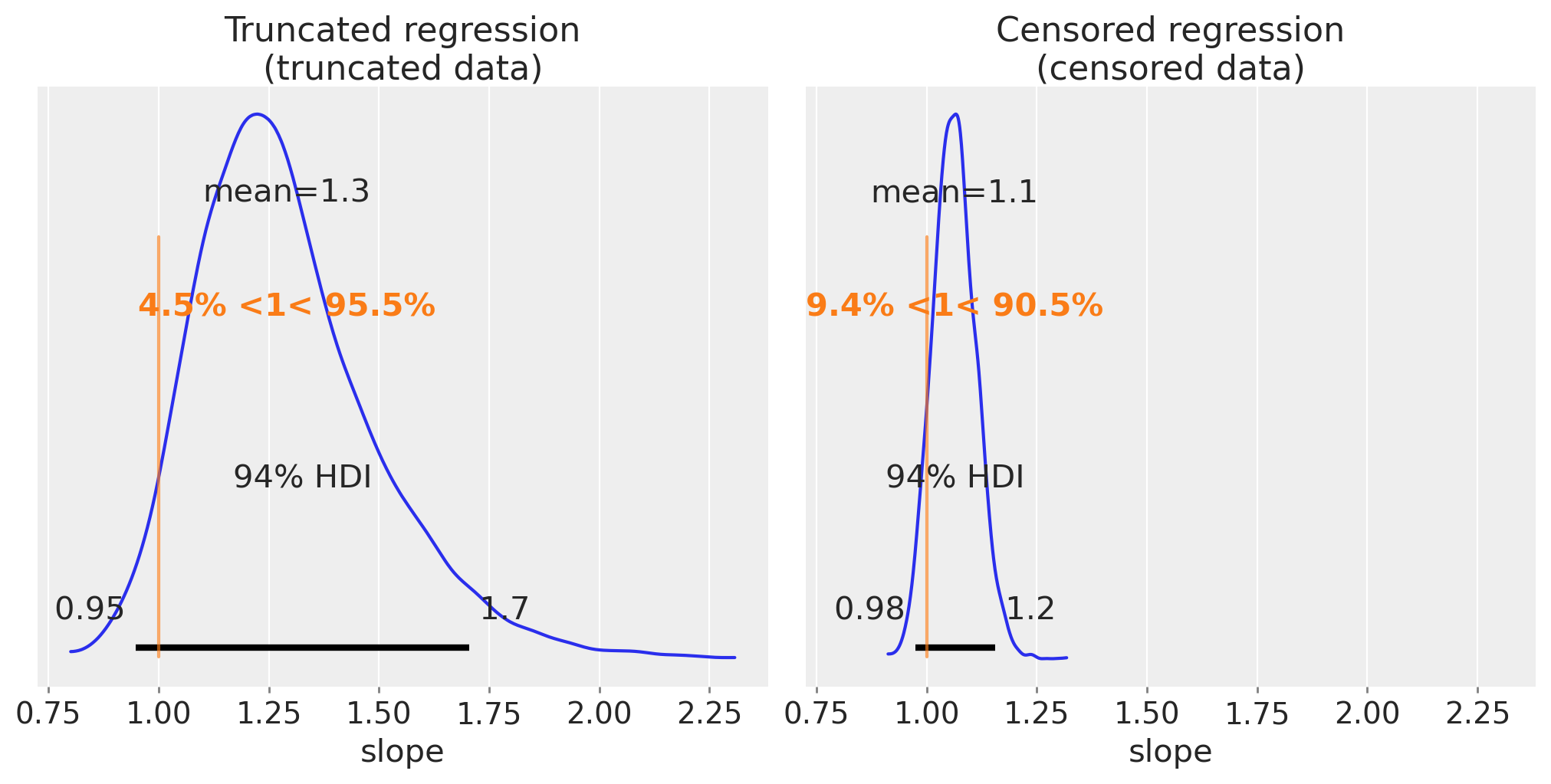
这些是好得多的估计值。有趣的是,我们可以看到删失回归的估计值比截断数据的估计值更精确。情况不一定总是如此,但这里的直觉是,x 和 y 数据在截断时被完全丢弃,但在删失时只有 y 数据变得部分未知。
然后我们可以推测,如果实验者可以选择截断或删失数据,那么选择删失可能比截断更好。
相应地,我们可以通过目视检查后验预测图来确认模型是良好的。
fig, ax = plt.subplots(1, 2, figsize=(10, 5), sharex=True, sharey=True)
pp_plot(xt, yt, truncated_fit, ax[0])
ax[0].set(title="Truncated data")
pp_plot(xc, yc, censored_fit, ax[1])
ax[1].set(title="Censored data");
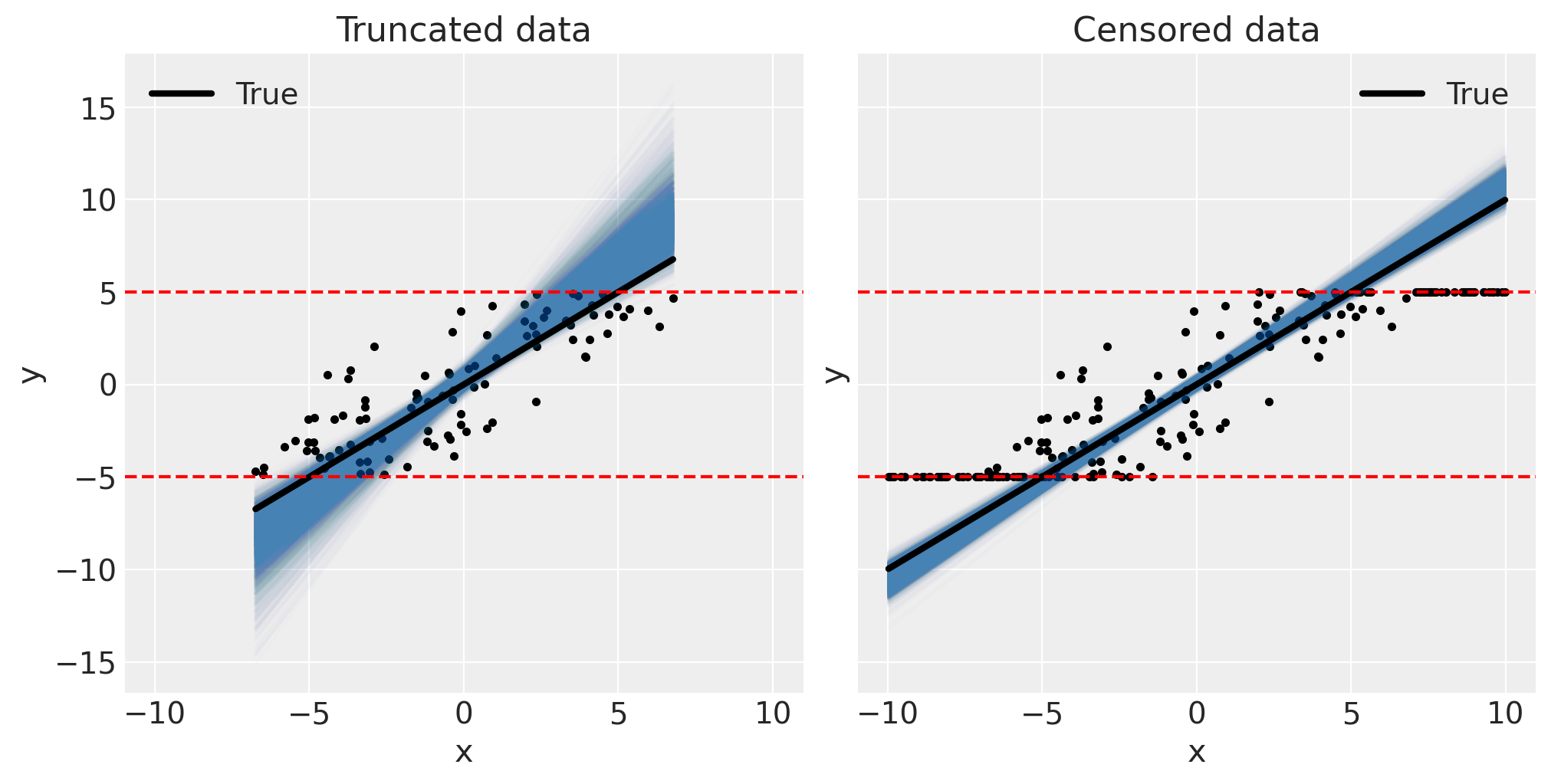
这结束了我们关于 PyMC 中截断和删失数据以及截断和删失回归模型的指南。虽然回归斜率估计偏差的程度会因上面讨论的许多因素而异,但希望这些示例使您确信将您对数据生成过程的了解编码到回归分析中的重要性。
进一步主题#
也可以将边界视为未知的潜在参数。如果这些边界不是完全已知的,并且可以对这些边界制定先验,那么就可以推断出边界是什么。但是,这可能被认为是过度杀伤 - 根据您的数据分析上下文,可能完全足以提取“足够好”的边界点估计值,以便获得合理的回归估计值。
上面介绍的删失回归模型采用了一种特定的方法,还有其他方法。例如,它没有尝试推断删失数据的真实潜在 y 值的后验信念。可以构建删失回归模型来估算这些删失的 y 值,但我们在此处没有讨论这一点,因为 插补 的主题值得单独重点介绍。PyMC 删失数据模型 示例也涵盖了此主题,其中一个特定的 示例模型用于估算删失数据。
进一步阅读#
在研究这个主题时,我发现大多数材料都侧重于最大似然估计方法,重点是数学推导而不是实际实现。Breen 等人 [1996] 撰写的一本简明数学小册子(共 80 页)涵盖了截断和删失以及其他缺失数据情景。也就是说,Gelman et al. [2013] 和 Gelman et al. [2020] 的《贝叶斯数据分析》中也用了几页篇幅介绍了这个主题。
参考文献#
水印#
%load_ext watermark
%watermark -n -u -v -iv -w -p pytensor,aeppl
Last updated: Sun Feb 05 2023
Python implementation: CPython
Python version : 3.10.9
IPython version : 8.9.0
pytensor: 2.8.11
aeppl : not installed
numpy : 1.24.1
arviz : 0.14.0
matplotlib: 3.6.3
pymc : 5.0.1
xarray : 2023.1.0
Watermark: 2.3.1
许可声明#
本示例库中的所有笔记本均根据 MIT 许可证 提供,该许可证允许修改和再分发以用于任何用途,前提是保留版权和许可声明。
引用 PyMC 示例#
要引用此笔记本,请使用 Zenodo 为 pymc-examples 存储库提供的 DOI。
重要提示
许多笔记本改编自其他来源:博客、书籍…… 在这种情况下,您也应引用原始来源。
另请记住引用您的代码使用的相关库。
这是一个 bibtex 中的引用模板
@incollection{citekey,
author = "<notebook authors, see above>",
title = "<notebook title>",
editor = "PyMC Team",
booktitle = "PyMC examples",
doi = "10.5281/zenodo.5654871"
}
渲染后可能看起来像