贝叶斯 copula 估计:描述相关的联合分布#
问题#
当我们处理多个变量(例如 \(a\) 和 \(b\))时,我们通常希望参数化地描述联合分布 \(P(a, b)\)。 如果我们幸运的话,那么这个联合分布在某种程度上可能是“简单的”。 例如,\(a\) 和 \(b\) 可能是统计独立的,在这种情况下,我们可以将联合分布分解为 \(P(a, b) = P(a) P(b)\),因此我们只需要为 \(P(a)\) 和 \(P(b)\) 找到适当的参数化描述。 即使这不合适,\(P(a, b)\) 也可能可以通过简单的多元分布(例如多元正态分布)很好地描述。
然而,通常当我们处理真实数据集时,\(P(a, b)\) 中存在复杂的关联结构,这意味着之前的两种方法都不可用。 因此,需要替代方法。
Copula 来救援#
这就是 copula 的用武之地。 这些 copula 允许您通过简单的多元分布(例如多元高斯分布)、两个边际分布和一些变换来描述具有关联结构的复杂分布 \(P(a, b)\)。 对于 copula 的非常易懂的介绍,我们建议阅读 Thomas Wiecki 的 这篇 博客文章。
本笔记本介绍了如何使用贝叶斯方法描述具有关联结构的分布 \(P(a, b)\),以推断 copula 的参数。 我们将采用的通用方法如下面的示意图所示。
在底部,我们有观测空间,数据存在于其中。
我们可以假设这些数据是通过从上到下的过程生成的 - 我们在多元正态空间中有一个多元正态分布,它经过两个阶段的转换,最终得到观测空间中的数据。
我们可以访问观测空间中的数据,但我们可以通过从一个空间转换到另一个空间,来推断多元正态空间中的参数。
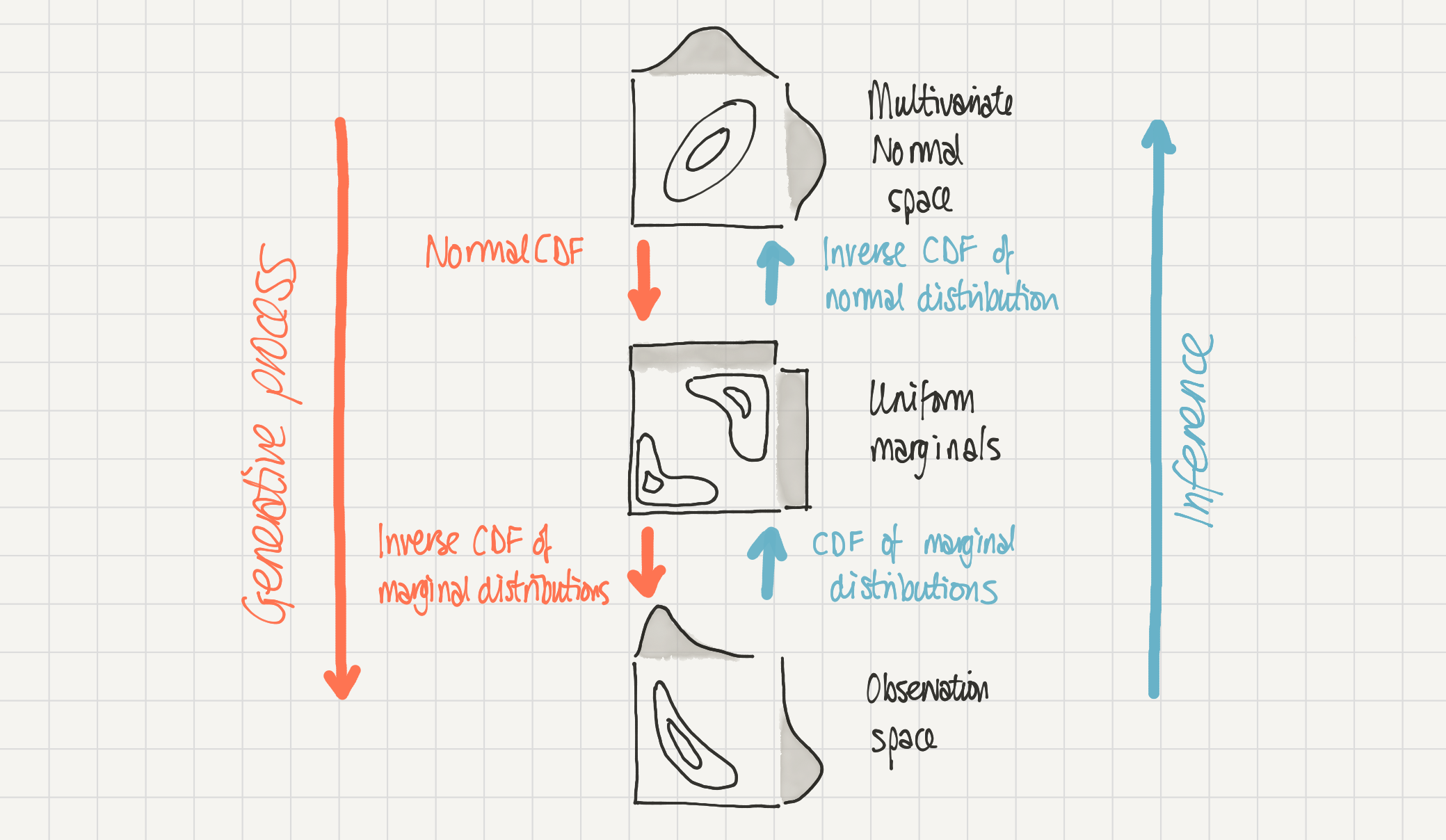
二维高斯 copula 的示意图。 我们在观测空间(底部)中的复杂分布 P(a, b) 被建模为由多元正态空间(顶部)中的二维高斯 copula 生成。 从多元正态空间到观测空间的映射(向下)是通过正态累积密度函数,然后是边际分布的逆累积密度函数来完成的。 相反的,推断过程(向上)可以通过边际分布的累积密度函数,然后是正态分布的逆累积密度函数来完成。#
本笔记本将介绍如何基于具有丰富关联结构的二元数据对 copula 进行推断。 我们 PyMC Labs 完成了这项工作,作为与 盖茨基金会 的一个更大项目的一部分,其中一些内容也在 这里 概述。

import arviz as az
import matplotlib.pyplot as plt
import numpy as np
import pymc as pm
import pytensor.tensor as pt
import seaborn as sns
from scipy.stats import expon, multivariate_normal, norm
%config InlineBackend.figure_format = 'retina'
az.style.use("arviz-darkgrid")
plt.rcParams.update({"font.size": 14, "figure.constrained_layout.use": False})
SEED = 43
rng = np.random.default_rng(SEED)
数据生成过程#
在深入推断之前,我们将花一些时间充实上面示意图中的步骤。 首先,我们将通过描述多元正态 copula 并将其转换为观测空间来演示生成模型。 其次,我们展示逆变换如何允许我们从观测空间返回到多元正态空间。 一旦我们确定了这些细节,我们将在后面的章节中继续进行推断过程。
现在我们将使用嵌套字典定义高斯 copula 的属性。 在顶层,我们有键 a 和 b 以及 rho。
rho描述了多元正态 copula 的相关系数。a和b也是字典,每个字典都包含边际分布(作为 scipy 分布对象)及其参数。
请注意,我们隐式地将多元正态分布定义为具有零均值和单位方差。 这是因为这些矩在通过“均匀空间”(我们 copula 示意图中的第二步)的转换中无法保留。
首先,我们定义真实的多元正态分布并从中抽取一些样本。
n_samples = 5000
# draw random samples in multivariate normal space
mu = [0, 0]
cov = [[1, θ["rho"]], [θ["rho"], 1]]
x = multivariate_normal(mu, cov).rvs(n_samples, random_state=rng)
a_norm = x[:, 0]
b_norm = x[:, 1]
sns.jointplot(x=a_norm, y=b_norm, height=6, kind="hex");
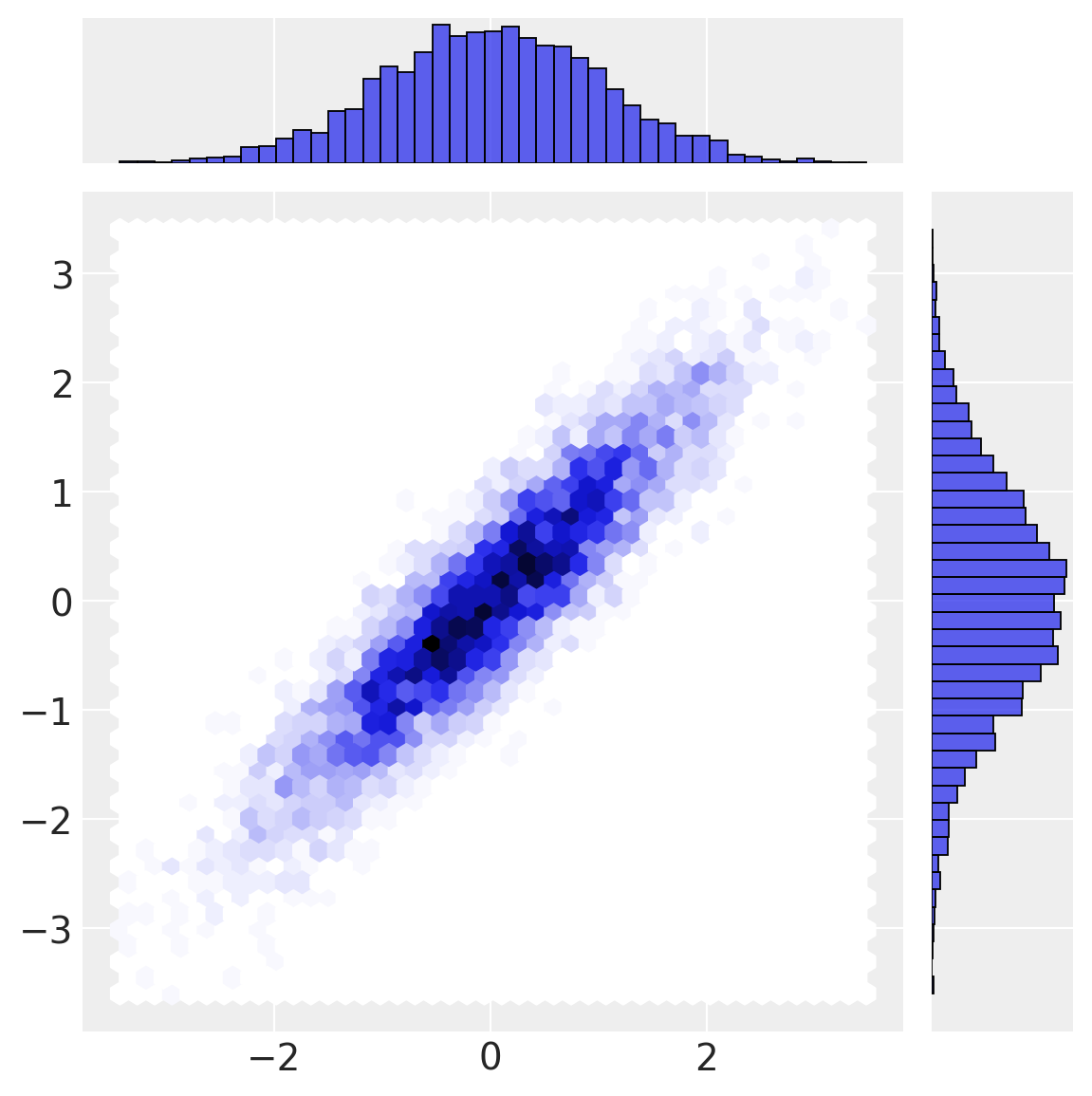
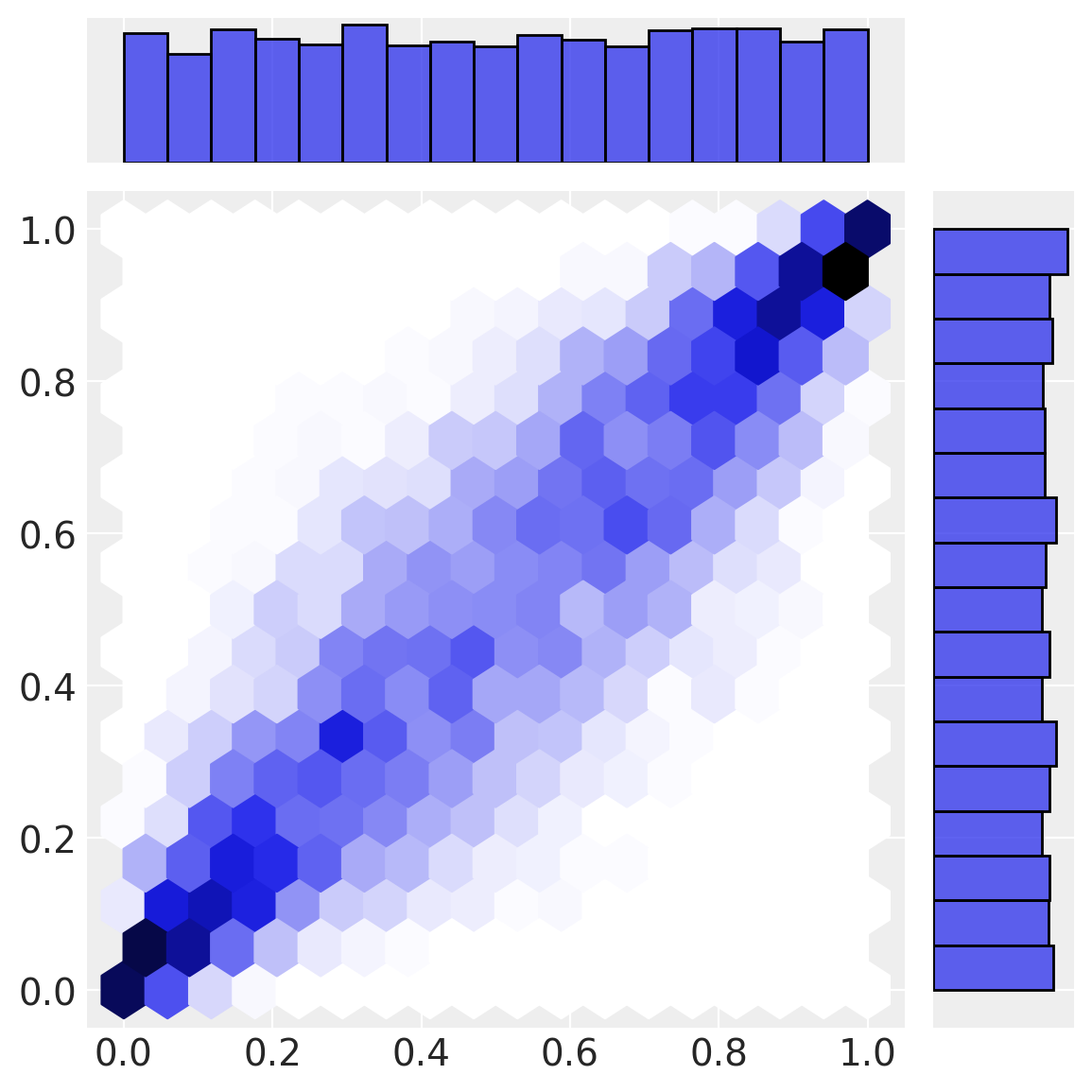
我们的第一个变换(正态 cdf)将数据从多元正态空间转换为均匀空间。 请注意,边际分布如何是均匀的,但来自多元正态空间的相关结构仍然保留在下面有趣的联合密度中。
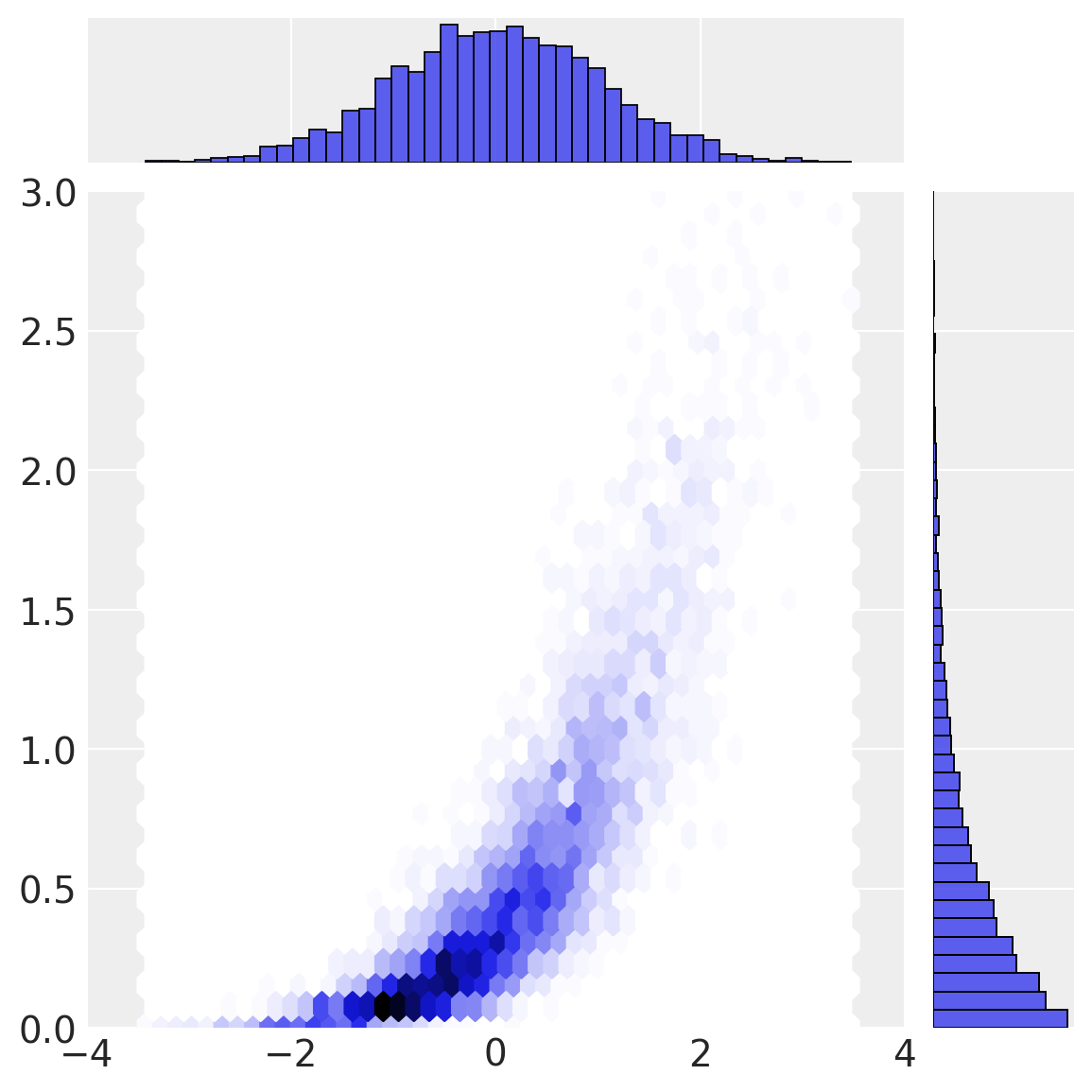
我们的最终变换(边际分布的逆 CDF)产生了观测空间中的 \(a\) 和 \(b\)。
# transform to observation space
a = θ["a_dist"].ppf(a_unif)
b = θ["b_dist"].ppf(b_unif)
sns.jointplot(x=a, y=b, height=6, kind="hex", xlim=(-4, 4), ylim=(0, 3));
Copula 推断过程#
为了理解所采用的方法,我们将逐步完成逆过程,从观测空间到多元正态空间。
# observation -> uniform space
a1 = θ["a_dist"].cdf(a)
b1 = θ["b_dist"].cdf(b)
sns.jointplot(x=a1, y=b1, kind="hex", height=6);


现在我们已经完成了图 1 中概述的内容。我们详细介绍了从多元正态空间到观测空间的数据生成过程。然后,我们看到了如何进行从观测空间到多元正态空间的逆(推断)过程。 这就是我们在 PyMC 模型中使用的方法。
用于 copula 和边际估计的 PyMC 模型#
我们将对多元正态空间中的参数进行推断,通过观测空间中的数据约束合理的参数值。 然而,我们也使用我们对 \(a\) 和 \(b\) 的观测来约束边际分布参数的估计。
在我们的实验中,我们探索了同时估计边际参数和 copula 的协方差参数的模型,但我们发现这不稳定。 我们在下面使用的解决方案被发现更稳健,并且涉及一个两步过程。
估计边际分布的参数。
使用步骤 1 中边际分布参数的点估计值,估计 copula 的协方差参数。
coords = {"obs_id": np.arange(len(a))}
with pm.Model(coords=coords) as marginal_model:
"""
Assumes observed data in variables `a` and `b`
"""
# marginal estimation
a_mu = pm.Normal("a_mu", mu=0, sigma=1)
a_sigma = pm.Exponential("a_sigma", lam=0.5)
pm.Normal("a", mu=a_mu, sigma=a_sigma, observed=a, dims="obs_id")
b_scale = pm.Exponential("b_scale", lam=0.5)
pm.Exponential("b", lam=1 / b_scale, observed=b, dims="obs_id")
pm.model_graph.model_to_graphviz(marginal_model)
with marginal_model:
marginal_idata = pm.sample(random_seed=rng)
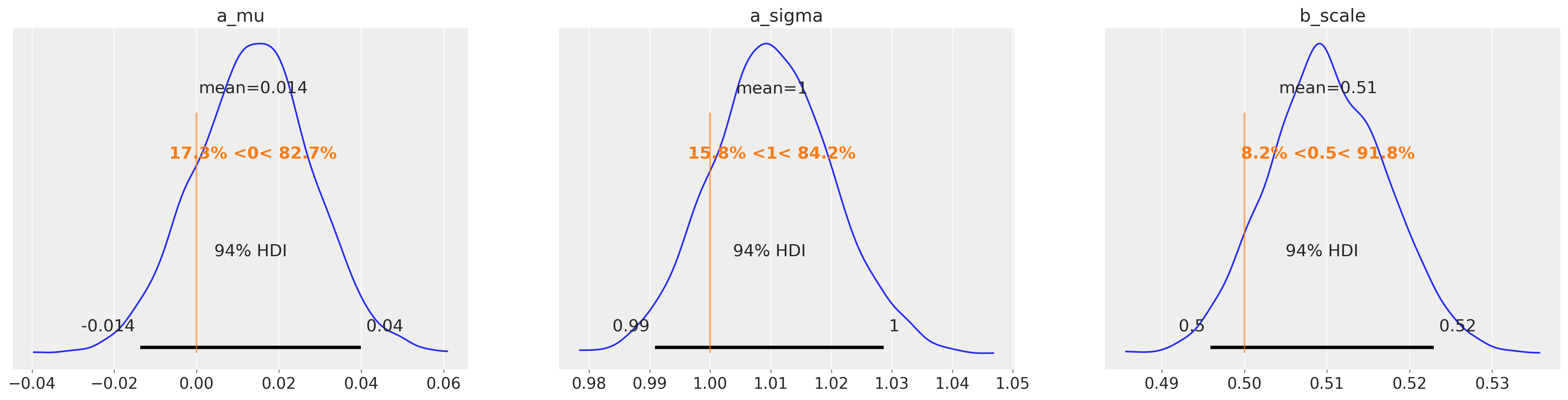
az.plot_posterior(
marginal_idata, var_names=["a_mu", "a_sigma", "b_scale"], ref_val=[0, 1.0, 1 / 2.0]
);
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [a_mu, a_sigma, b_scale]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 1 seconds.
在下面的 copula 模型中,您可以看到我们设置了协方差参数的先验分布。 此参数的后验分布受到多元正态空间中数据的约束。 但是为了做到这一点,我们需要将观测空间中的观测值 [a, b] 转换为多元正态空间,我们将其存储在 data 中。
关于使用点估计:正如您将在下面的代码中看到的那样,我们选择使用步骤 1 中的点估计值,而不是步骤 1 中的完整后验分布。 这是一个简化,我们选择这样做是因为将后验分布作为参数传递给分布时,张量形状处理的复杂性。
然而,在笔记本审查期间,@OriolAbril (PyMC Examples 存储库的维护者之一) 正确指出,使用点估计值在分布下评估的数据点的 logcdf 的指数 不一定会 返回一个值,该值等于在许多可能的分布(从完整后验构建)下评估的数据点的 logcdf 的指数的期望值。 为了确保笔记本的及时进展,我们选择按原样显示代码,但也留下此注释,供我们未来的自己稍后更新笔记本,同时也为未来的读者提供通过修改示例来解决此问题的机会。
def transform_data(marginal_idata):
# point estimates
a_mu = marginal_idata.posterior["a_mu"].mean().item()
a_sigma = marginal_idata.posterior["a_sigma"].mean().item()
b_scale = marginal_idata.posterior["b_scale"].mean().item()
# transformations from observation space -> uniform space
__a = pt.exp(pm.logcdf(pm.Normal.dist(mu=a_mu, sigma=a_sigma), a))
__b = pt.exp(pm.logcdf(pm.Exponential.dist(lam=1 / b_scale), b))
# uniform space -> multivariate normal space
_a = pm.math.probit(__a)
_b = pm.math.probit(__b)
# join into an Nx2 matrix
data = pt.math.stack([_a, _b], axis=1).eval()
return data, a_mu, a_sigma, b_scale
data, a_mu, a_sigma, b_scale = transform_data(marginal_idata)
coords.update({"param": ["a", "b"], "param_bis": ["a", "b"]})
with pm.Model(coords=coords) as copula_model:
# Prior on covariance of the multivariate normal
chol, corr, stds = pm.LKJCholeskyCov(
"chol",
n=2,
eta=2.0,
sd_dist=pm.Exponential.dist(1.0),
compute_corr=True,
)
cov = pm.Deterministic("cov", chol.dot(chol.T), dims=("param", "param_bis"))
# Likelihood function
pm.MvNormal("N", mu=0.0, cov=cov, observed=data, dims=("obs_id", "param"))
pm.model_graph.model_to_graphviz(copula_model)
with copula_model:
copula_idata = pm.sample(random_seed=rng, tune=2000, cores=1)
az.plot_posterior(copula_idata, var_names=["cov"], ref_val=[1.0, θ["rho"], θ["rho"], 1.0]);
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Sequential sampling (2 chains in 1 job)
NUTS: [chol]
/Users/benjamv/opt/mambaforge/envs/pymc_env/lib/python3.11/site-packages/pytensor/compile/function/types.py:970: RuntimeWarning: invalid value encountered in accumulate
self.vm()
Sampling 2 chains for 2_000 tune and 1_000 draw iterations (4_000 + 2_000 draws total) took 7 seconds.
We recommend running at least 4 chains for robust computation of convergence diagnostics
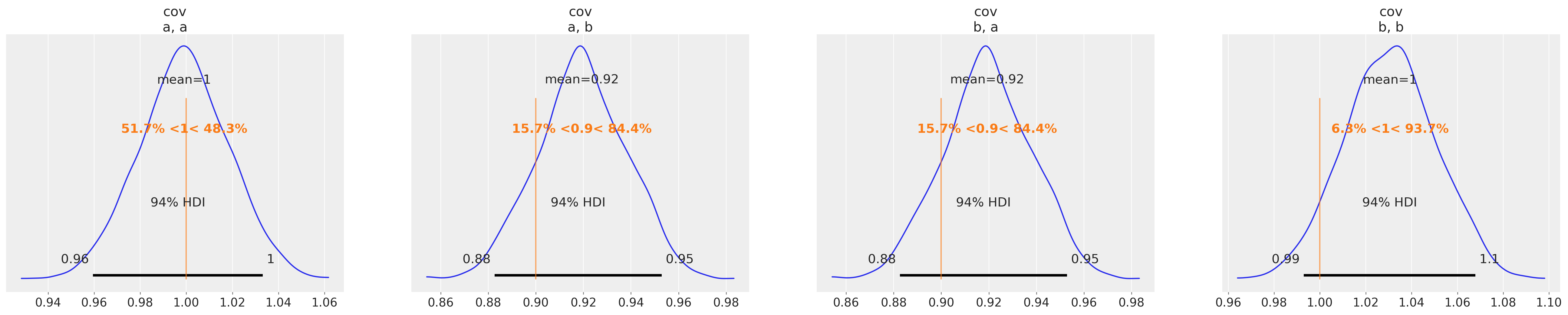
您可以看到,我们已成功恢复用于生成样本数据的多元正态 copula 的协方差矩阵。
在下面的部分中,我们将使用此信息来从我们的 \(P(a, b)\) 参数化描述中进行采样。
将推断结果与真实数据进行比较#
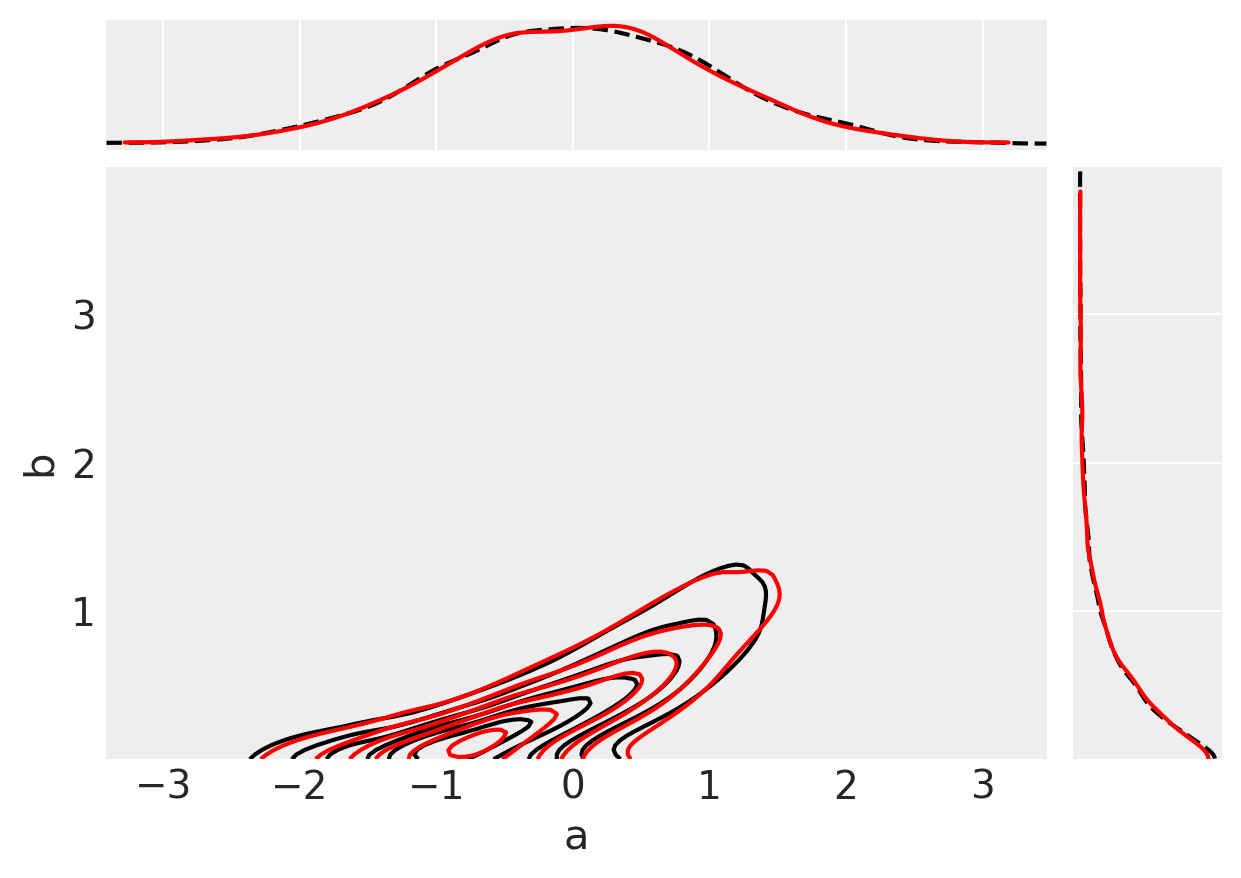
最后,我们可以进行视觉检查,看看我们的推断结果(红色)是否与我们原始观察到的数据(黑色)相符。
# data munging to extract covariance estimates from copula_idata in useful shape
d = {k: v.values.reshape((-1, *v.shape[2:])) for k, v in copula_idata.posterior[["cov"]].items()}
# generate (a, b) samples
ab = np.vstack([multivariate_normal([0, 0], cov).rvs() for cov in d["cov"]])
# transform to uniform space
uniform_a = norm().cdf(ab[:, 0])
uniform_b = norm().cdf(ab[:, 1])
# transform to observation space
# estimated marginal parameters a_mu, a_sigma, b_scale are point estimates from marginal estimation.
ppc_a = norm(loc=a_mu, scale=a_sigma).ppf(uniform_a)
ppc_b = expon(scale=b_scale).ppf(uniform_b)
# plot data in black
ax = az.plot_pair(
{"a": a, "b": b},
marginals=True,
# kind=["kde", "scatter"],
kind="kde",
scatter_kwargs={"alpha": 0.1},
kde_kwargs=dict(
contour_kwargs=dict(colors="k", linestyles="--"), contourf_kwargs=dict(alpha=0)
),
marginal_kwargs=dict(color="k", plot_kwargs=dict(ls="--")),
)
# plot inferences in red
axs = az.plot_pair(
{"a": ppc_a, "b": ppc_b},
marginals=True,
# kind=["kde", "scatter"],
kind="kde",
scatter_kwargs={"alpha": 0.01},
kde_kwargs=dict(contour_kwargs=dict(colors="r", linestyles="-"), contourf_kwargs=dict(alpha=0)),
marginal_kwargs=dict(color="r"),
ax=ax,
);
/Users/benjamv/opt/mambaforge/envs/pymc_env/lib/python3.11/site-packages/arviz/plots/backends/matplotlib/kdeplot.py:166: UserWarning: The following kwargs were not used by contour: 'linestyle'
qcs = ax.contour(x_x, y_y, density, **contour_kwargs)
致谢#
我们要感谢 Jonathan Sedar、Junpeng Lao 和 Oriol Abril 在本笔记本的开发过程中提供的有益建议。
作者#
由 Eric Ma 和 Benjamin T. Vincent 于 2023 年 11 月撰写 (pymc-examples#257)。
水印#
%load_ext watermark
%watermark -n -u -v -iv -w -p pytensor,xarray
Last updated: Mon Dec 04 2023
Python implementation: CPython
Python version : 3.11.6
IPython version : 8.18.1
pytensor: 2.18.1
xarray : 2023.11.0
arviz : 0.16.1
pymc : 5.10.0
seaborn : 0.13.0
matplotlib: 3.8.2
numpy : 1.26.2
pytensor : 2.18.1
Watermark: 2.4.3
许可声明#
本示例库中的所有笔记本均根据 MIT 许可证 提供,该许可证允许修改和再分发以用于任何用途,前提是保留版权和许可声明。
引用 PyMC 示例#
要引用此笔记本,请使用 Zenodo 为 pymc-examples 存储库提供的 DOI。
重要提示
许多笔记本都改编自其他来源:博客、书籍…… 在这种情况下,您也应该引用原始来源。
另请记住引用您的代码使用的相关库。
这是一个 bibtex 中的引用模板
@incollection{citekey,
author = "<notebook authors, see above>",
title = "<notebook title>",
editor = "PyMC Team",
booktitle = "PyMC examples",
doi = "10.5281/zenodo.5654871"
}
渲染后可能看起来像