模型平均#
import os
import arviz as az
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
import pymc as pm
print(f"Running on PyMC v{pm.__version__}")
Running on PyMC v5.16.2+24.g799c98f41
rng = np.random.seed(2741)
az.style.use("arviz-darkgrid")
当我们面对多个模型时,我们有几种选择。其中一种是执行模型选择,PyMC 示例 模型比较 和 GLM:模型选择 就是例证,通常最好也包括后验预测检查,以便决定保留哪个模型。丢弃除一个模型之外的所有模型相当于断言,在评估的模型中,一个是正确的(在某些标准下)概率为 1,其余都是不正确的。在大多数情况下,这将是一种夸大其词,忽略了我们在模型中存在的不确定性。这有点类似于计算完整的后验分布,然后只保留一个点估计,例如后验均值;我们可能会对我们真正知道的东西变得过于自信。您还可以浏览 blog/tag/model-comparison 标签以查找相关帖子。
模型选择的另一种替代方案是执行模型选择,但承认我们丢弃的模型。如果模型数量不是很大,这可以成为论文、演示文稿、论文等技术讨论的一部分。如果听众不够专业,这可能不是一个好主意。
另一种替代方案,也是本示例的主题,是执行模型平均。这个想法是通过每个模型的优点来加权每个模型,并从每个模型生成预测,预测与这些权重成比例。有几种方法可以做到这一点,包括本笔记本中将简要讨论的三种方法。您将在 Yao et al. [2018] 和 Yao et al. [2022] 的工作中找到更全面的解释。
伪贝叶斯模型平均#
贝叶斯模型可以通过其边际似然进行加权,这被称为贝叶斯模型平均。虽然这在理论上很有吸引力,但在实践中却存在问题:一方面,边际似然对先验的规范高度敏感,而参数估计并非如此;另一方面,计算边际似然通常是一项具有挑战性的任务。此外,贝叶斯模型平均在 \(\mathcal{M}\)-开放设置中存在缺陷,在这种设置中,真实的数据生成过程不是正在拟合的候选模型之一 Yao et al. [2018]。一种更稳健的方法是计算期望对数逐点预测密度 (ELPD)。
其中 \(N\) 是数据点的数量,\(y_i\) 是第 i 个数据点,\(\theta\) 是模型的参数,\(p(y_i \mid \theta)\) 是给定参数的第 i 个数据点的似然性,\(p(\theta \mid y)\) 是后验分布。
一旦我们计算出每个模型的 ELPD,我们就可以通过执行以下操作来计算权重
其中 \(dELPD_i\) 是最佳 ELPD 模型与第 i 个模型之间的差异。
这种方法称为伪贝叶斯模型平均,或类 Akaike 加权,是一种启发式方法,用于计算每个模型的相对概率(给定一组固定的模型)。请注意,我们取指数是为了“还原” ELPD 公式中对数的影响,分母是一个归一化项,以确保权重总和为 1。稍微夸张地说,我们可以将这些权重解释为每个模型解释数据的概率。
到目前为止一切顺利,但 ELPD 是一个理论量,在实践中我们需要对其进行近似。为此,ArviZ 提供了两种方法
WAIC,广泛适用信息准则
LOO,帕累托平滑留一法交叉验证。
两者都需要包含对数似然组的 InferenceData,并且计算速度同样快。我们建议使用 LOO,因为它具有更好的实践属性和更好的诊断方法(因此我们知道何时 ELPD 估计出现问题)。
使用贝叶斯 Bootstrap 的伪贝叶斯模型平均#
上述计算权重的公式是一种很好且简单的方法,但有一个主要缺点:它没有考虑 ELPD 计算中的不确定性。我们可以计算 ELPD 值的标准误差(假设高斯近似),并相应地修改上述公式。或者我们可以做一些更稳健的事情,例如使用 贝叶斯 Bootstrap 来估计和纳入这种不确定性。
堆叠#
我们将讨论的第三种方法是 Yao et al. [2018] 提出的预测分布堆叠。我们希望将多个模型组合成一个元模型,以最大限度地减少元模型与真实生成模型之间的差异。当使用对数评分规则时,这等同于
其中 \(n\) 是数据点的数量,\(K\) 是模型的数量。为了强制执行解决方案,我们约束 \(w\) 为 \(w_k \ge 0\) 和 \(\sum_{k=1}^{K} w_k = 1\)。
数量 \(p(y_i \mid y_{-i}, M_k)\) 是 \(M_k\) 模型的留一法预测分布。计算它需要拟合每个模型 \(n\) 次,每次都留出一个数据点。幸运的是,这正是 LOO 以非常有效的方式近似的内容。因此,我们可以将 LOO 和堆叠一起使用。公平地说,即使 WAIC 以不同的方式近似 ELPD,我们也可以使用 WAIC。
加权后验预测样本#
一旦我们使用上述 3 种方法中的任何一种计算出权重,我们就可以使用它们来获得加权后验预测样本。我们将说明如何使用身体脂肪数据集 [Penrose et al., 1985] 来做到这一点。此数据集包含来自 251 个人的测量数据,包括他们的体重、身高、腹围、腕围等。我们的目的是预测身体脂肪的百分比,由 siri 变量估计,也可从数据集中获得。
让我们从加载数据开始
try:
d = pd.read_csv(os.path.join("..", "data", "body_fat.csv"))
except FileNotFoundError:
d = pd.read_csv(pm.get_data("body_fat.csv"))
d.head()
| siri | 年龄 | 体重 | 身高 | 颈部 | 胸部 | 腹部 | 臀部 | 大腿 | 膝盖 | 脚踝 | 肱二头肌 | 前臂 | 手腕 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 12.3 | 23 | 70.1 | 172 | 36.2 | 93.1 | 85.2 | 94.5 | 59.0 | 37.3 | 21.9 | 32.0 | 27.4 | 17.1 |
| 1 | 6.1 | 22 | 78.8 | 184 | 38.5 | 93.6 | 83.0 | 98.7 | 58.7 | 37.3 | 23.4 | 30.5 | 28.9 | 18.2 |
| 2 | 25.3 | 22 | 70.0 | 168 | 34.0 | 95.8 | 87.9 | 99.2 | 59.6 | 38.9 | 24.0 | 28.8 | 25.2 | 16.6 |
| 3 | 10.4 | 26 | 84.0 | 184 | 37.4 | 101.8 | 86.4 | 101.2 | 60.1 | 37.3 | 22.8 | 32.4 | 29.4 | 18.2 |
| 4 | 28.7 | 24 | 83.8 | 181 | 34.4 | 97.3 | 100.0 | 101.9 | 63.2 | 42.2 | 24.0 | 32.2 | 27.7 | 17.7 |
现在我们有了数据,我们将构建两个模型,这两个模型都是简单的线性回归,区别在于第一个模型我们将使用变量 abdomen,第二个模型我们将使用变量 wrist、height 和 weight。
with pm.Model() as model_0:
alpha = pm.Normal("alpha", mu=0, sigma=1)
beta = pm.Normal("beta", mu=0, sigma=1)
sigma = pm.HalfNormal("sigma", 5)
mu = alpha + beta * d["abdomen"]
siri = pm.Normal("siri", mu=mu, sigma=sigma, observed=d["siri"])
idata_0 = pm.sample(idata_kwargs={"log_likelihood": True}, random_seed=rng)
pm.sample_posterior_predictive(idata_0, extend_inferencedata=True, random_seed=rng)
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [alpha, beta, sigma]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 7 seconds.
Sampling: [siri]
with pm.Model() as model_1:
alpha = pm.Normal("alpha", mu=0, sigma=1)
beta = pm.Normal("beta", mu=0, sigma=1, shape=3)
sigma = pm.HalfNormal("sigma", 5)
mu = alpha + pm.math.dot(beta, d[["wrist", "height", "weight"]].T)
siri = pm.Normal("siri", mu=mu, sigma=sigma, observed=d["siri"])
idata_1 = pm.sample(idata_kwargs={"log_likelihood": True}, random_seed=rng)
pm.sample_posterior_predictive(idata_1, extend_inferencedata=True, random_seed=rng)
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [alpha, beta, sigma]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 36 seconds.
Sampling: [siri]
在使用 LOO(或 WAIC)比较或平均模型之前,我们应该检查我们没有抽样问题,并且后验预测检查是合理的。为了简洁起见,我们将跳过这些步骤,直接进行模型平均。
首先我们需要调用 az.compare 来计算每个模型的 LOO 值,并使用 stacking 计算权重。这些是默认选项,如果您想执行伪贝叶斯模型平均,您可以使用 method='BB-pseudo-BMA',其中包括 ELPD 中不确定性的贝叶斯 Bootstrap 估计。
model_dict = dict(zip(["model_0", "model_1"], [idata_0, idata_1]))
comp = az.compare(model_dict)
comp
| rank | elpd_loo | p_loo | elpd_diff | 体重 | se | dse | warning | scale | |
|---|---|---|---|---|---|---|---|---|---|
| model_1 | 0 | -817.216895 | 3.626704 | 0.000000 | 0.639236 | 10.496342 | 0.000000 | False | log |
| model_0 | 1 | -825.344978 | 1.832909 | 8.128083 | 0.360764 | 9.970768 | 8.698358 | False | log |
我们可以从 weight 列中看到,model_1 的权重约为 \(\approx 0.6\),model_2 的权重约为 \(\approx 0.4\)。要使用这些权重生成后验预测样本,我们可以使用 az.weighted_posterior 函数。此函数接受 InferenceData 对象和权重,并返回一个新的 InferenceData 对象。
ppc_w = az.weight_predictions(
[model_dict[name] for name in comp.index],
weights=comp.weight,
)
ppc_w
-
<xarray.Dataset> Dimensions: (siri_dim_2: 251, sample: 3999) Coordinates: * siri_dim_2 (siri_dim_2) int64 0 1 2 3 4 5 6 ... 244 245 246 247 248 249 250 * sample (sample) object MultiIndex * chain (sample) int64 2 3 1 2 1 2 3 3 3 3 3 3 ... 3 3 3 1 0 0 2 0 2 1 2 * draw (sample) int64 682 691 550 397 831 520 ... 638 997 295 483 606 9 Data variables: siri (siri_dim_2, sample) float64 17.75 16.43 14.7 ... 30.98 27.67 Attributes: created_at: 2024-08-23T16:10:41.836182+00:00 arviz_version: 0.20.0.dev0 inference_library: pymc inference_library_version: 5.16.2+24.g799c98f41 -
<xarray.Dataset> Dimensions: (siri_dim_0: 251) Coordinates: * siri_dim_0 (siri_dim_0) int64 0 1 2 3 4 5 6 ... 244 245 246 247 248 249 250 Data variables: siri (siri_dim_0) float64 12.3 6.1 25.3 10.4 ... 33.6 29.3 26.0 31.9 Attributes: created_at: 2024-08-23T16:10:41.440917+00:00 arviz_version: 0.20.0.dev0 inference_library: pymc inference_library_version: 5.16.2+24.g799c98f41
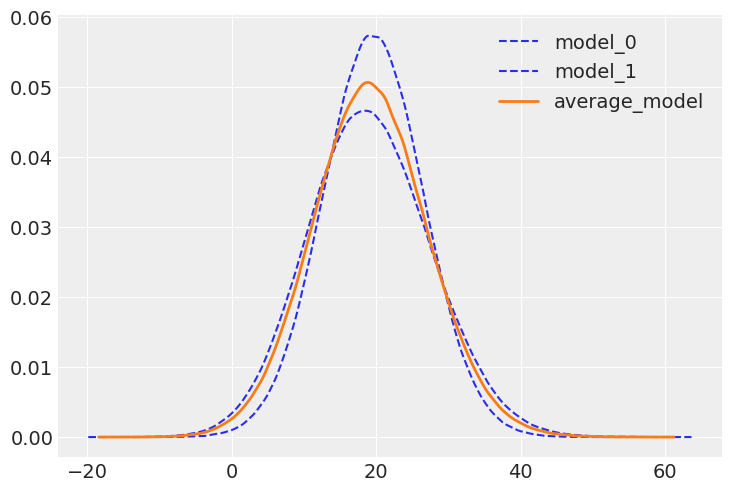
从下面的图中我们可以看到,平均模型是两个模型的组合。
az.plot_kde(
idata_0.posterior_predictive["siri"].values,
plot_kwargs={"color": "C0", "linestyle": "--"},
label="model_0",
)
az.plot_kde(
idata_1.posterior_predictive["siri"].values,
plot_kwargs={"color": "C0", "linestyle": "--"},
label="model_1",
)
az.plot_kde(
ppc_w.posterior_predictive["siri"].values,
plot_kwargs={"color": "C1", "linewidth": 2},
label="average_model",
);
做还是不做?#
当您想提高预测的稳健性时,模型平均是一个好主意。通常,模型的组合将比任何单个模型具有更好的预测性能。当模型是互补的时,尤其如此。我们在本示例中尚未探讨的是以某种方式为模型分配权重,使它们针对数据的不同部分而变化。这可以按照 Yao et al. [2022] 中讨论的那样完成。
何时不进行模型平均?很多时候,我们可以创建新的模型,这些模型可以有效地充当其他模型的平均值。例如,在本示例中,我们可以创建一个包含所有变量的新模型。这实际上是一个非常明智的做法。请注意,如果模型排除一个变量,则相当于将该变量的系数设置为零。如果我们对包含变量和不包含变量的模型进行平均,则相当于将系数设置为零和包含变量的模型中系数的值之间的值。这是一个非常简单的示例,但相同的推理适用于更复杂的模型。
分层模型是另一个例子,我们构建模型的连续版本而不是处理离散版本。一个玩具示例是想象我们有一枚硬币,我们想要估计其偏差程度,一个介于 0 和 1 之间的数字,其中 0.5 表示正面和反面的机会相等(公平硬币)。我们可以考虑两个单独的模型:一个先验偏向正面,一个先验偏向反面。我们可以拟合这两个单独的模型,然后对它们进行平均。另一种方法是构建分层模型来估计先验分布。与其考虑两个离散模型,不如计算一个连续模型,该模型将离散模型视为特殊情况。哪种方法更好?这取决于我们的具体问题。我们是否有充分的理由考虑两个离散模型,还是我们的问题最好用更大的连续模型来表示?
参考文献#
Yuling Yao、Aki Vehtari、Daniel Simpson 和 Andrew Gelman。《使用堆叠来平均贝叶斯预测分布(附讨论)》。贝叶斯分析,2018 年 9 月。网址:https://doi.org/10.1214\%2F17-ba1091,doi:10.1214/17-ba1091。
Yuling Yao、Gregor Pirš、Aki Vehtari 和 Andrew Gelman。《贝叶斯分层堆叠:有些模型(在某些地方)有用》。贝叶斯分析,17(4):1043 – 1071,2022 年。网址:https://doi.org/10.1214/21-BA1287,doi:10.1214/21-BA1287。
K. W. Penrose、A. G. Nelson 和 A. G. Fisher。《使用简单测量技术对男性进行通用身体成分预测方程》。医学与运动科学与锻炼,17(2):189,1985 年 4 月。网址:https://journals.lww.com/acsm-msse/citation/1985/04000/generalized_body_composition_prediction_equation.37.aspx (于 2023-10-17 访问)。
水印#
%load_ext watermark
%watermark -n -u -v -iv -w
Last updated: Fri Aug 23 2024
Python implementation: CPython
Python version : 3.11.5
IPython version : 8.16.1
arviz : 0.20.0.dev0
pandas : 2.1.2
pymc : 5.16.2+24.g799c98f41
numpy : 1.24.4
matplotlib: 3.8.4
Watermark: 2.4.3
许可声明#
本示例 галерея 中的所有笔记本均根据 MIT 许可证 提供,该许可证允许修改和再分发以用于任何用途,前提是保留版权和许可声明。
引用 PyMC 示例#
要引用此笔记本,请使用 Zenodo 为 pymc-examples 仓库提供的 DOI。
重要提示
许多笔记本都改编自其他来源:博客、书籍……在这种情况下,您也应该引用原始来源。
另请记住引用您的代码使用的相关库。
这是一个 bibtex 中的引用模板
@incollection{citekey,
author = "<notebook authors, see above>",
title = "<notebook title>",
editor = "PyMC Team",
booktitle = "PyMC examples",
doi = "10.5281/zenodo.5654871"
}
一旦渲染,它可能看起来像